
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,214,061 – 11,214,221 |
| Length | 160 |
| Max. P | 0.993846 |

| Location | 11,214,061 – 11,214,181 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.50 |
| Mean single sequence MFE | -54.28 |
| Consensus MFE | -47.69 |
| Energy contribution | -48.55 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.08 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.43 |
| SVM RNA-class probability | 0.993846 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11214061 120 + 27905053 GCAGGAGAUUCAGCUAGAGGCCGGUCCGCAGGAUGGUACCAUUCUGGUGACAUGGCAGCCGGUUAACAGGCCCACCUCGACGGGGCCUGUAACCGGCUAUGCUGUGUACGCCGAUGGUAA ...((..((.((((.....((((((((.(((((((....)))))))).))).))))(((((((((.(((((((.........))))))))))))))))..)))).))...))........ ( -58.40) >DroVir_CAF1 14954 120 + 1 GCAAGACAUCCAACUGGAGGCUGGUCCUCAGGAUGGCACCAUUCUGGUGACAUGGCAGCCGGUUAACAGACCUACGUCCACGGGGCCUGUAACCGGCUAUGCUGUGUAUGCCGAUGGUAA ......(((((.....((((.....)))).)))))..((((((..((((((((((((((((((((.(((.(((.((....))))).)))))))))))..)))))))).)))))))))).. ( -51.20) >DroPse_CAF1 24088 120 + 1 GCAGGAGAUUCAGCUUGAGGCUGGCCCACAGGAUGGUACCAUUCUGGUGACAUGGCAGCCGGUCGCCAGGCCCACCGCGACUGGGCCUGUUACCGGCUAUGCUGUGUACGCCGAUGGUAA ..........((((.....))))(((...((((((....))))))((((((((((((((((((.((.(((((((.......))))))))).))))))..)))))))).))))...))).. ( -56.40) >DroWil_CAF1 18661 120 + 1 GCAGGAAAUCCAGCUUGAGGCUGGCCCUCAAGAUGGUACCAUUCUGGUGACAUGGCAGCCGGUUAACAGGCCCACCUCAACGGGGCCAGUAACCGGCUAUGCUGUGUAUGCCGAUGGUAA .........(((.(((((((.....))))))).)))(((((((..((((((((((((((((((((...(((((.........)))))..))))))))..)))))))).))))))))))). ( -57.40) >DroMoj_CAF1 15458 120 + 1 ACAAGACAUCCAAUUGGAGGCUGGUCCUCAGGAUGGCACCAUUCUGGUGACAUGGCAGCCGGUUAACCGACCCACGUCCACGGGGCCCGUAACCGGCUACGCUGUGUACGCCGAUGGUAA ......(((((.....((((.....)))).)))))..((((((..((((((((((((((((((((...(.(((.((....))))))...)))))))))..))))))).)))))))))).. ( -52.80) >DroPer_CAF1 24934 120 + 1 GCAGGAGAUUCAGCUUGAGGCUGGCCCACAGGAUGGUACCAUUCUGGUGACAUGGCAGCCGGUCGCCAGGCCCACCGCGACUGAGCCUGUUACCGGCUAUGCUGUGUACGCCGAUGGUAA ..........((((.....))))(((...((((((....))))))((((((((((((((((((.((.((((.((.......)).)))))).))))))..)))))))).))))...))).. ( -49.50) >consensus GCAGGAGAUCCAGCUUGAGGCUGGCCCUCAGGAUGGUACCAUUCUGGUGACAUGGCAGCCGGUUAACAGGCCCACCUCGACGGGGCCUGUAACCGGCUAUGCUGUGUACGCCGAUGGUAA .........(((((.....))))).((..((((((....))))))((((((((((((((((((((.(((((((.........))))))))))))))))..))))))).))))...))... (-47.69 = -48.55 + 0.86)



| Location | 11,214,061 – 11,214,181 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.50 |
| Mean single sequence MFE | -49.73 |
| Consensus MFE | -40.39 |
| Energy contribution | -40.50 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.830620 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11214061 120 - 27905053 UUACCAUCGGCGUACACAGCAUAGCCGGUUACAGGCCCCGUCGAGGUGGGCCUGUUAACCGGCUGCCAUGUCACCAGAAUGGUACCAUCCUGCGGACCGGCCUCUAGCUGAAUCUCCUGC ........((.(....((((..(((((((((((((((((......).))))))).)))))))))(((..(((..(((.(((....))).)))..))).))).....))))...).))... ( -52.70) >DroVir_CAF1 14954 120 - 1 UUACCAUCGGCAUACACAGCAUAGCCGGUUACAGGCCCCGUGGACGUAGGUCUGUUAACCGGCUGCCAUGUCACCAGAAUGGUGCCAUCCUGAGGACCAGCCUCCAGUUGGAUGUCUUGC ........(((((...((((.((((((((((((((((.((....))..)))))).))))))))))..(((.((((.....)))).)))...((((.....))))..)))).))))).... ( -46.70) >DroPse_CAF1 24088 120 - 1 UUACCAUCGGCGUACACAGCAUAGCCGGUAACAGGCCCAGUCGCGGUGGGCCUGGCGACCGGCUGCCAUGUCACCAGAAUGGUACCAUCCUGUGGGCCAGCCUCAAGCUGAAUCUCCUGC ......(((((...(((((...(((((((..((((((((.......))))))))...)))))))(((((.((....)))))))......)))))((....))....)))))......... ( -49.90) >DroWil_CAF1 18661 120 - 1 UUACCAUCGGCAUACACAGCAUAGCCGGUUACUGGCCCCGUUGAGGUGGGCCUGUUAACCGGCUGCCAUGUCACCAGAAUGGUACCAUCUUGAGGGCCAGCCUCAAGCUGGAUUUCCUGC .(((((((((...(((..(((..(((((((((.((((((......).))))).).)))))))))))..)))..)).).))))))(((.(((((((.....))))))).)))......... ( -50.90) >DroMoj_CAF1 15458 120 - 1 UUACCAUCGGCGUACACAGCGUAGCCGGUUACGGGCCCCGUGGACGUGGGUCGGUUAACCGGCUGCCAUGUCACCAGAAUGGUGCCAUCCUGAGGACCAGCCUCCAAUUGGAUGUCUUGU .(((((((((.(.(((..(.((((((((((((.(((((((....)).))))).).)))))))))))).)))).)).).)))))).(((((.((((.....)))).....)))))...... ( -51.30) >DroPer_CAF1 24934 120 - 1 UUACCAUCGGCGUACACAGCAUAGCCGGUAACAGGCUCAGUCGCGGUGGGCCUGGCGACCGGCUGCCAUGUCACCAGAAUGGUACCAUCCUGUGGGCCAGCCUCAAGCUGAAUCUCCUGC ......(((((...(((((...(((((((..((((((((.......))))))))...)))))))(((((.((....)))))))......)))))((....))....)))))......... ( -46.90) >consensus UUACCAUCGGCGUACACAGCAUAGCCGGUUACAGGCCCCGUCGAGGUGGGCCUGUUAACCGGCUGCCAUGUCACCAGAAUGGUACCAUCCUGAGGACCAGCCUCAAGCUGAAUCUCCUGC ......(((((.......((.(((((((((((((((((.........))))))).))))))))))....(((..(((.(((....))).)))..)))..)).....)))))......... (-40.39 = -40.50 + 0.11)

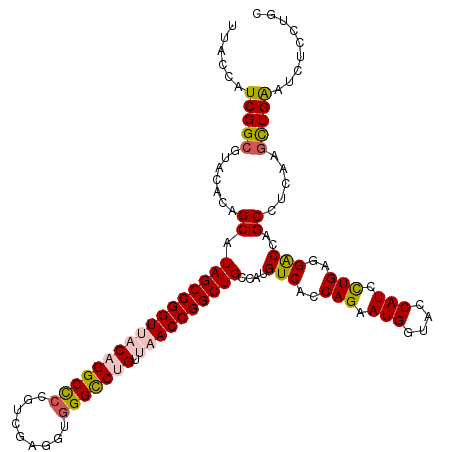

| Location | 11,214,101 – 11,214,221 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.61 |
| Mean single sequence MFE | -49.05 |
| Consensus MFE | -37.85 |
| Energy contribution | -37.05 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.656393 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11214101 120 + 27905053 AUUCUGGUGACAUGGCAGCCGGUUAACAGGCCCACCUCGACGGGGCCUGUAACCGGCUAUGCUGUGUACGCCGAUGGUAAGAAGGUCACCGACAUUAACUCGCCCACCGGCGACCACGCC .....((((((((((((((((((((.(((((((.........)))))))))))))))..)))))))).(((((..((..((...(((...))).....))..))...)))))....)))) ( -56.60) >DroVir_CAF1 14994 120 + 1 AUUCUGGUGACAUGGCAGCCGGUUAACAGACCUACGUCCACGGGGCCUGUAACCGGCUAUGCUGUGUAUGCCGAUGGUAAAAAGGUGACGGACAUUAAUUCCCCGACGGGCGAUCAUGCA .....((((((((((((((((((((.(((.(((.((....))))).)))))))))))..)))))))).))))(((.((.....(....)(((.......))).......)).)))..... ( -43.00) >DroGri_CAF1 16405 120 + 1 AUUCUGGUGACAUGGCAGCCGGUUAACAGACCCACGUCCACGGGGCCUGUAACCGGCUAUGCUGUGUAUGCCGAUGGUAAAAAGGUGACGGACAUCAAUUCACCAACGGGCGAUCACGCA .....((((((((((((((((((((.(((.(((.((....))))).)))))))))))..)))))))).))))(((.((.....(((((..(....)...))))).....)).)))..... ( -49.70) >DroWil_CAF1 18701 120 + 1 AUUCUGGUGACAUGGCAGCCGGUUAACAGGCCCACCUCAACGGGGCCAGUAACCGGCUAUGCUGUGUAUGCCGAUGGUAAAAAGGUCACCGACAUCAAUUCCCCCACUGGCGACCAUGCC ....(((((((((((((((((((((...(((((.........)))))..))))))))..)))))))).)))))..((((....((((.(((................))).)))).)))) ( -46.99) >DroMoj_CAF1 15498 120 + 1 AUUCUGGUGACAUGGCAGCCGGUUAACCGACCCACGUCCACGGGGCCCGUAACCGGCUACGCUGUGUACGCCGAUGGUAAGAAGGUGACCGACAUCAACUCACCUACGGGCGAUCAUGCA .....((((((((((((((((((((...(.(((.((....))))))...)))))))))..))))))).))))(((.((..(.((((((...........)))))).)..)).)))..... ( -50.40) >DroPer_CAF1 24974 120 + 1 AUUCUGGUGACAUGGCAGCCGGUCGCCAGGCCCACCGCGACUGAGCCUGUUACCGGCUAUGCUGUGUACGCCGAUGGUAAGAAGGUCACCGAUAUCAAUUCGCCCACCGGCGACCAUGCC ....(((((((((((((((((((.((.((((.((.......)).)))))).))))))..)))))))).)))))..((((....((((...((.......))(((....))))))).)))) ( -47.60) >consensus AUUCUGGUGACAUGGCAGCCGGUUAACAGACCCACCUCCACGGGGCCUGUAACCGGCUAUGCUGUGUACGCCGAUGGUAAAAAGGUCACCGACAUCAAUUCACCCACGGGCGACCAUGCA ....((((.((((((((((((((((.(((.(((.........))).))))))))))))..))))))).((((...((..((..(((.......)))..))..))....)))))))).... (-37.85 = -37.05 + -0.80)



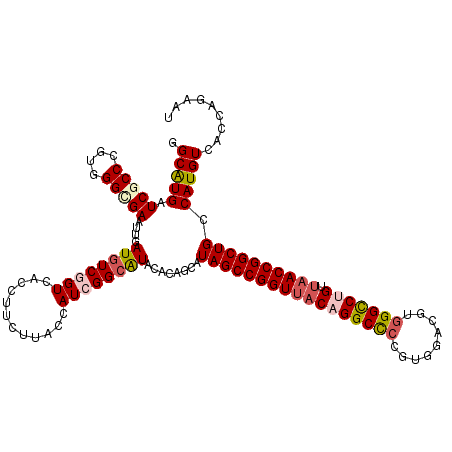
| Location | 11,214,101 – 11,214,221 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.61 |
| Mean single sequence MFE | -52.02 |
| Consensus MFE | -39.32 |
| Energy contribution | -40.02 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.81 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.583259 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11214101 120 - 27905053 GGCGUGGUCGCCGGUGGGCGAGUUAAUGUCGGUGACCUUCUUACCAUCGGCGUACACAGCAUAGCCGGUUACAGGCCCCGUCGAGGUGGGCCUGUUAACCGGCUGCCAUGUCACCAGAAU (((((((((((((((((..(((.....(((...)))...))).)))))))))..........(((((((((((((((((......).))))))).)))))))))))))))))........ ( -61.30) >DroVir_CAF1 14994 120 - 1 UGCAUGAUCGCCCGUCGGGGAAUUAAUGUCCGUCACCUUUUUACCAUCGGCAUACACAGCAUAGCCGGUUACAGGCCCCGUGGACGUAGGUCUGUUAACCGGCUGCCAUGUCACCAGAAU .(((((...(((.(..((..((....((.....))....))..))..))))..........((((((((((((((((.((....))..)))))).)))))))))).)))))......... ( -38.10) >DroGri_CAF1 16405 120 - 1 UGCGUGAUCGCCCGUUGGUGAAUUGAUGUCCGUCACCUUUUUACCAUCGGCAUACACAGCAUAGCCGGUUACAGGCCCCGUGGACGUGGGUCUGUUAACCGGCUGCCAUGUCACCAGAAU ((.(((((.(((...(((((((.((((....))))....)))))))..)))..........(((((((((((((((((((....)).))))))).))))))))))....))))))).... ( -51.50) >DroWil_CAF1 18701 120 - 1 GGCAUGGUCGCCAGUGGGGGAAUUGAUGUCGGUGACCUUUUUACCAUCGGCAUACACAGCAUAGCCGGUUACUGGCCCCGUUGAGGUGGGCCUGUUAACCGGCUGCCAUGUCACCAGAAU ((((((((.(((.((((((..((((....))))..))......)))).)))...........((((((((((.((((((......).))))).).)))))))))))))))))........ ( -52.30) >DroMoj_CAF1 15498 120 - 1 UGCAUGAUCGCCCGUAGGUGAGUUGAUGUCGGUCACCUUCUUACCAUCGGCGUACACAGCGUAGCCGGUUACGGGCCCCGUGGACGUGGGUCGGUUAACCGGCUGCCAUGUCACCAGAAU ..((((...(((((((((((((.((((....))))....))))))((((((.(((.....))))))))))))))))..))))((((((((((((....)))))..)))))))........ ( -52.30) >DroPer_CAF1 24974 120 - 1 GGCAUGGUCGCCGGUGGGCGAAUUGAUAUCGGUGACCUUCUUACCAUCGGCGUACACAGCAUAGCCGGUAACAGGCUCAGUCGCGGUGGGCCUGGCGACCGGCUGCCAUGUCACCAGAAU (((((((((((((((((..((.......))(....).......)))))))))..........(((((((..((((((((.......))))))))...)))))))))))))))........ ( -56.60) >consensus GGCAUGAUCGCCCGUGGGCGAAUUGAUGUCGGUCACCUUCUUACCAUCGGCAUACACAGCAUAGCCGGUUACAGGCCCCGUGGACGUGGGCCUGUUAACCGGCUGCCAUGUCACCAGAAU .(((((.(((((....)))))....((((((((............))))))))........(((((((((((((((((.........))))))).)))))))))).)))))......... (-39.32 = -40.02 + 0.70)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:28 2006