
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,212,408 – 11,212,528 |
| Length | 120 |
| Max. P | 0.650155 |

| Location | 11,212,408 – 11,212,528 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.04 |
| Mean single sequence MFE | -47.60 |
| Consensus MFE | -40.52 |
| Energy contribution | -41.22 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.650155 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
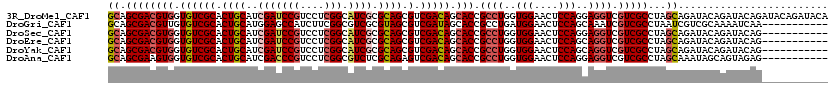
>3R_DroMel_CAF1 11212408 120 + 27905053 UGUAUCUGUAUCUGUAUCUGUAUCUGCUAGGCGACGACCUCCUGGAGUUCCACCAGGCGGUGCUGUCGACGCUGCGCGAUGCCGAGGACGGAUCGAUGCAGUGCGACACCACGUCGCUGC .............................((((((((((.(((((.......))))).)))(.(((((.(((((((((((.(((....))))))).)))))))))))).).))))))).. ( -51.10) >DroGri_CAF1 15251 109 + 1 -----------UUGAUUUUGCGACGAUUAGGCGACGAUUUGCUGGAGUUCCAUCAGGCGGUGCUAUCGACGCUACGCGACGCCGAAGAUGGCUCCAUGCAGUGCGACACAACGUCGCUGC -----------..................(((((((...((((((((..(((((.((((.(((............))).))))...)))))))))).)))(((...)))..))))))).. ( -39.10) >DroSec_CAF1 14432 109 + 1 -----------CUGUAUCUGUAUCUGCUAGGCGACGACCUCCUGGAGUUCCACCAGGCGGUGCUGUCGACGCUGCGCGAUGCCGAGGACGGAUCGAUGCAGUGCGACACCACGUCGCUGC -----------..................((((((((((.(((((.......))))).)))(.(((((.(((((((((((.(((....))))))).)))))))))))).).))))))).. ( -51.10) >DroEre_CAF1 19605 109 + 1 -----------CUGUAUCUGUAUCUGCUAGGCGACGACCUGCUGGAGUUCCACCAGGCGGUGCUGUCGACGCUGCGCGAUGCCGAGGACGGAUCGAUGCAGUGCGACACCACGUCGCUGC -----------..................(((((((.((((.(((....))).))))).(((.(((((.(((((((((((.(((....))))))).)))))))))))).))))))))).. ( -51.20) >DroYak_CAF1 15066 109 + 1 -----------CUGUAUCUGUAUCUGCUAGGCGACGACCUGCUGGAGUUCCACCAGGCGGUGCUGUCGACGCUGCGCGAUGCCGAGGACGGAUCGAUGCAGUGCGACACCACGUCGCUGC -----------..................(((((((.((((.(((....))).))))).(((.(((((.(((((((((((.(((....))))))).)))))))))))).))))))))).. ( -51.20) >DroAna_CAF1 14940 109 + 1 -----------CUCUACUGCUAUUUGCUAGGCGACGACCUCCUGGAGUUCCACCAGGCGGUGCUGUCGACUCUGCGAGACGCCGAGGACGGGUCGAUGCAGUGCGACACCACUUCGCUGC -----------.......((.....))..(((((.((((.(((((.......))))).)))(.(((((.(.(((((.(((.(((....))))))..))))).)))))).).).))))).. ( -41.90) >consensus ___________CUGUAUCUGUAUCUGCUAGGCGACGACCUCCUGGAGUUCCACCAGGCGGUGCUGUCGACGCUGCGCGAUGCCGAGGACGGAUCGAUGCAGUGCGACACCACGUCGCUGC .............................(((((((.(((..(((....)))..)))).(((.(((((.(((((((((((.(((....))))))).)))))))))))).))))))))).. (-40.52 = -41.22 + 0.70)



| Location | 11,212,408 – 11,212,528 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.04 |
| Mean single sequence MFE | -45.92 |
| Consensus MFE | -36.77 |
| Energy contribution | -37.63 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.546888 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11212408 120 - 27905053 GCAGCGACGUGGUGUCGCACUGCAUCGAUCCGUCCUCGGCAUCGCGCAGCGUCGACAGCACCGCCUGGUGGAACUCCAGGAGGUCGUCGCCUAGCAGAUACAGAUACAGAUACAGAUACA ((.((((((.(.((((((.((((..(((((((....))).)))).)))).).))))).)(((.(((((.......))))).)))))))))...))......................... ( -46.50) >DroGri_CAF1 15251 109 - 1 GCAGCGACGUUGUGUCGCACUGCAUGGAGCCAUCUUCGGCGUCGCGUAGCGUCGAUAGCACCGCCUGAUGGAACUCCAGCAAAUCGUCGCCUAAUCGUCGCAAAAUCAA----------- ...((((((..(((.((...(((.((((((((((...((((..((............))..)))).)))))..))))))))...)).))).....))))))........----------- ( -41.30) >DroSec_CAF1 14432 109 - 1 GCAGCGACGUGGUGUCGCACUGCAUCGAUCCGUCCUCGGCAUCGCGCAGCGUCGACAGCACCGCCUGGUGGAACUCCAGGAGGUCGUCGCCUAGCAGAUACAGAUACAG----------- ((.((((((.(.((((((.((((..(((((((....))).)))).)))).).))))).)(((.(((((.......))))).)))))))))...))..............----------- ( -46.50) >DroEre_CAF1 19605 109 - 1 GCAGCGACGUGGUGUCGCACUGCAUCGAUCCGUCCUCGGCAUCGCGCAGCGUCGACAGCACCGCCUGGUGGAACUCCAGCAGGUCGUCGCCUAGCAGAUACAGAUACAG----------- ((.((((((((.((((((.((((..(((((((....))).)))).)))).).))))).))).(((((.(((....))).))))).)))))...))..............----------- ( -50.00) >DroYak_CAF1 15066 109 - 1 GCAGCGACGUGGUGUCGCACUGCAUCGAUCCGUCCUCGGCAUCGCGCAGCGUCGACAGCACCGCCUGGUGGAACUCCAGCAGGUCGUCGCCUAGCAGAUACAGAUACAG----------- ((.((((((((.((((((.((((..(((((((....))).)))).)))).).))))).))).(((((.(((....))).))))).)))))...))..............----------- ( -50.00) >DroAna_CAF1 14940 109 - 1 GCAGCGAAGUGGUGUCGCACUGCAUCGACCCGUCCUCGGCGUCUCGCAGAGUCGACAGCACCGCCUGGUGGAACUCCAGGAGGUCGUCGCCUAGCAAAUAGCAGUAGAG----------- ((.((((.(((.((((((.((((...((((((....))).)))..)))).).))))).)))(.(((((.......))))).).)))).))...((.....)).......----------- ( -41.20) >consensus GCAGCGACGUGGUGUCGCACUGCAUCGAUCCGUCCUCGGCAUCGCGCAGCGUCGACAGCACCGCCUGGUGGAACUCCAGCAGGUCGUCGCCUAGCAGAUACAGAUACAG___________ ((.((((((((.((((((.((((..(((((((....))).)))).)))).).))))).))).((((..(((....)))..)))).)))))...))......................... (-36.77 = -37.63 + 0.86)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:19 2006