
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,193,886 – 11,193,995 |
| Length | 109 |
| Max. P | 0.523030 |

| Location | 11,193,886 – 11,193,995 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.62 |
| Mean single sequence MFE | -38.68 |
| Consensus MFE | -20.84 |
| Energy contribution | -22.02 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.54 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.523030 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
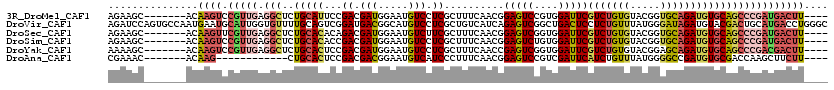
>3R_DroMel_CAF1 11193886 109 - 27905053 AGAAGC-------ACAAGUCCGUUGAGGCUCUGCAUUCCGACGAUGGAAUGUCCUCGCUUUCAACGGAGUCCGUGGAUUCGUCUGUGUACGGUGCAGAUGUGCAGCCCGAUGACUU---- ....((-------(((..(((((((((((...(((((((......)))))))....)).)))))))))(((((((.((......)).))))).))...))))).............---- ( -39.00) >DroVir_CAF1 4198 120 - 1 AGAUCCAGUGCCAAUGAAUGCAUUGGUGUUUUGCAGUCGGAUGACGGCAUGUCCUCGCUGUCAUCAGAGUCGGCUGACUCCUCUGUUUAUGGGAUAGAUGUACGACUGCAUGACCUGGGC ...(((((.(((((((....)))))))(((.(((((((((((((((((........))))))))).(((((....))))).((((((.....))))))....)))))))).)))))))). ( -53.50) >DroSec_CAF1 4395 109 - 1 AGAAGC-------ACAAGUUCGUUGAGGCUCUGCACACAGACGAUGGAAUGUCUUCGCUUUCAACGGAGUCGGUGGAUUCGUCUGUGUACGGUGCAGAUGUGCAGCCCGAUGACUU---- ......-------..((((.(((((.(((.(((.((((((((((.....((.(((((.......))))).))......)))))))))).)))((((....))))))))))))))))---- ( -39.90) >DroSim_CAF1 3810 109 - 1 AGAAGC-------ACAAGUCCGUUGAGGCUCUGCACACCGACGAUGGAAUGUCCUCGCUUUCAACGGAGUCUGUGGAUUCGUCUGUGUACGGUGCAGAUGUGCAGCCCGAUGACUU---- ......-------..(((((.((((.((((.(((((((((((.(((((..((((.((..(((....)))..)).))))...))))))).)))))....)))).)))))))))))))---- ( -34.90) >DroYak_CAF1 3788 109 - 1 AAAAGC-------ACAAGUCCGUUGAGGCUCUGCACUCCGACGAUGGAAUGUCCUCGCUUUCAACCGAGUCGGUGGAUUCGUCUGUGUACGGAGCAGAUGUGCAGCCCGACGACUU---- ......-------..(((((.((((.((((.(((((((((((.(((((..((((((((((......))).))).))))...))))))).)))))....)))).)))))))))))))---- ( -37.70) >DroAna_CAF1 4145 97 - 1 CGAAAC-------ACAAG------------CUGCACUCCGACGACGGAAUGUCAUCCCUUUCAACGGAGUCCGUCGAUUCAUCUGUUUAUGGGGCCGAUGUGCGACCAAGCUUCUU---- ......-------..(((------------(((.((((((..((.(((......)))...))..)))))).)((((...((((.(((.....))).))))..))))..)))))...---- ( -27.10) >consensus AGAAGC_______ACAAGUCCGUUGAGGCUCUGCACUCCGACGAUGGAAUGUCCUCGCUUUCAACGGAGUCGGUGGAUUCGUCUGUGUACGGUGCAGAUGUGCAGCCCGAUGACUU____ ...............((((.(((((.(((..(((((...((.(((.....))).))..........(((((....)))))(((((((.....)))))))))))))))))))))))).... (-20.84 = -22.02 + 1.17)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:02 2006