
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,070,078 – 11,070,195 |
| Length | 117 |
| Max. P | 0.690844 |

| Location | 11,070,078 – 11,070,195 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.71 |
| Mean single sequence MFE | -38.43 |
| Consensus MFE | -34.00 |
| Energy contribution | -34.58 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.690844 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11070078 117 + 27905053 ---CAGAUGUCUAAAGCUCCUGCUGUUGGUAUUGAUUUGGGCACCACCUACUCGUGCGUGGGCGUGUUCCAGCAUGGCAAGGUCGAGAUCAUCGCCAACGACCAGGGUAAUCGUACCACU ---.....(((...(((....)))((((((..((((((.(((....(((((......)))))(((((....))))).....))).))))))..)))))))))...((((....))))... ( -37.90) >DroVir_CAF1 2204 120 + 1 UUGCAGAUGUCUAAAGCUCCAGCUGUUGGUAUUGAUUUGGGCACAACCUACUCUUGCGUGGGUGUGUUCCAGCAUGGCAAGGUUGAGAUCAUUGCCAACGAUCAGGGUAAUCGUACCACC .....(((......(((....)))(((((((.(((((((((((((.(((((......))))))))))))((((........)))))))))).))))))).)))..((((....))))... ( -39.80) >DroGri_CAF1 1 117 + 1 ---CAGAUGUCUAAAGCACCAGCUGUCGGUAUUGAUUUGGGCACAACCUACUCCUGCGUGGGUGUGUUCCAGCAUGGCAAGGUUGAAAUCAUUGCCAACGACCAGGGUAAUCGUACGACA ---....((((((....(((....((((........(((((((((.(((((......))))))))).)))))..(((((((((....))).)))))).))))...))).....)).)))) ( -35.10) >DroWil_CAF1 216 118 + 1 --UUAGAUGUCUAAAGCACCUGCUGUUGGUAUUGAUUUGGGAACCACCUACUCUUGCGUGGGUGUCUUCCAGCAUGGCAAGGUUGAAAUUAUUGCCAACGAUCAGGGUAAUCGUACCACA --((((....)))).((.((((.((((((((.((((((((((..(((((((......)))))))..)))).((...)).......)))))).))))))))..))))))............ ( -37.10) >DroMoj_CAF1 1 117 + 1 ---CAGAUGUCUAAGGCUCCAGCUGUUGGUAUUGAUUUGGGCACAACCUACUCUUGCGUGGGUGUGUUCCAGCAUGGCAAGGUUGAGAUCAUUGCCAACGAUCAGGGUAAUCGUACCACC ---..(((......(((....)))(((((((.(((((((((((((.(((((......))))))))))))((((........)))))))))).))))))).)))..((((....))))... ( -39.80) >DroPer_CAF1 1 116 + 1 ----AGAUGUCUAAAGCACCUGCUGUUGGUAUUGAUUUGGGCACAACCUACUCUUGUGUGGGUGUGUUCCAGCAUGGCAAGGUCGAGAUCAUUGCCAACGACCAGGGUAAUCGUACGACA ----...((((.......((((.((((((((.(((((((((((((.(((((......)))))))))))).....((((...)))))))))).))))))))..))))(((....))))))) ( -40.90) >consensus ___CAGAUGUCUAAAGCACCAGCUGUUGGUAUUGAUUUGGGCACAACCUACUCUUGCGUGGGUGUGUUCCAGCAUGGCAAGGUUGAGAUCAUUGCCAACGACCAGGGUAAUCGUACCACA ........(((...(((....)))(((((((.((((((((((((.((((((......))))))))))))((((........)))))))))).))))))))))...((((....))))... (-34.00 = -34.58 + 0.59)

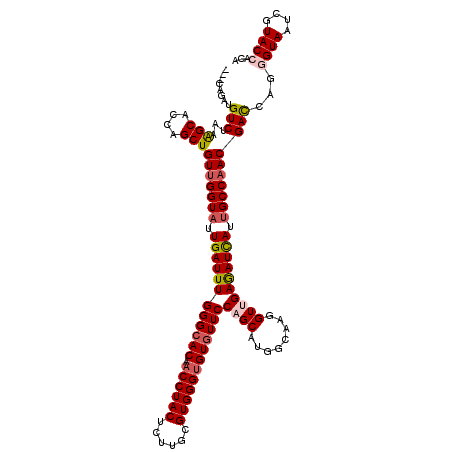

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:17:16 2006