
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,025,003 – 11,025,157 |
| Length | 154 |
| Max. P | 0.998228 |

| Location | 11,025,003 – 11,025,117 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.16 |
| Mean single sequence MFE | -34.87 |
| Consensus MFE | -29.41 |
| Energy contribution | -28.88 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967524 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11025003 114 + 27905053 UGC---UGCGGACCGGC---CGAAAACGAAGGGCACCUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUUAUCGCUGCCAUAAU .((---.((((....((---(..........))).((((((((((((((.....)))))))).((((((((......)))))))).))))))...............)))).))...... ( -36.10) >DroVir_CAF1 220 118 + 1 --UUGAACCGACACGGGAUACUAAGAAAAAGGGCACUUUUAUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUAUUAUUAACAGAGUUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAU --.....(((...)))...............(((......(((((((((.....)))))))))((((((((......))))))))...(((((((((......))))))))))))..... ( -35.40) >DroGri_CAF1 2906 118 + 1 --CGGAUAAGGAACGGGAUGUCAACAUAAAGGGAUCAUUUAUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUAUUGUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUUAU --............(((((.((........)).)))....(((((((((.....)))))))))((((((((......))))))))...(((((((((......))))))))).))..... ( -32.70) >DroWil_CAF1 318 111 + 1 AG---------AACGUGAAGCUAAAGGCAAGGGGAAUUUUAUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUCUUAUUAACGGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAU ..---------..............(((............(((((((((.....)))))))))((((((((......))))))))...(((((((((......))))))))))))..... ( -32.90) >DroMoj_CAF1 3922 120 + 1 UAUUGAACCGGAAAGGGAGAAGAAAAUCAAGGGCAGUUUUAUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUAUUAUUAACAGAGCUAGGUGAUAAGACUUUCUUUAUUGCUGCCAUAAU ..............((.((.....(((.(((((.(((((((((((((((.....))))))...((((((((......))))))))......))))))))).)))))))).)).))..... ( -35.80) >DroAna_CAF1 447 117 + 1 AUC---AUCGGACAGGCAGGCGAAAACGAAGGGAACCUUUAUCGAUGCGUUCACCGCAUCGAUCUCUGUUAUCCUUUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUAGCUGCCAUAAU ...---........(((((.((....))(((((((..((((((((((((.....))))))...((((((((......))))))))......))))))..)))))))....)))))..... ( -36.30) >consensus __C___ACCGGAACGGGAGGCGAAAACAAAGGGCACUUUUAUCGAUGCGUUUACAGCAUCGAUCUCUGUUAUAUUAUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAU ..............................((........(((((((((.....)))))))))((((((((......))))))))...(((((((((......))))))))).))..... (-29.41 = -28.88 + -0.52)


| Location | 11,025,003 – 11,025,117 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.16 |
| Mean single sequence MFE | -32.70 |
| Consensus MFE | -23.77 |
| Energy contribution | -23.43 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.93 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.53 |
| SVM RNA-class probability | 0.961487 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11025003 114 - 27905053 AUUAUGGCAGCGAUAAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAGGUGCCCUUCGUUUUCG---GCCGGUCCGCA---GCA ......((.(((..................(((..((((((((......))))))))(((((((((.....)))))))))..))).(((..(((....))---)..))).))).---)). ( -37.10) >DroVir_CAF1 220 118 - 1 AUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAACUCUGUUAAUAAUAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAUAAAAGUGCCCUUUUUCUUAGUAUCCCGUGUCGGUUCAA-- ....((((((..((((((((((......(((....((((((((......))))))))(((((((((.....)))))))))....)))....)))))))..)))..).)))))......-- ( -31.20) >DroGri_CAF1 2906 118 - 1 AUAAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAACAAUAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAUAAAUGAUCCCUUUAUGUUGACAUCCCGUUCCUUAUCCG-- ..((((((((((.(((((...(((...........((((((((......))))))))(((((((((.....)))))))))....)))..)))))))))).....))))).........-- ( -32.70) >DroWil_CAF1 318 111 - 1 AUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCCGUUAAUAAGAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAUAAAAUUCCCCUUGCCUUUAGCUUCACGUU---------CU .....((((((.((((........)))).))....(((.((((......)))).)))(((((((((.....)))))))))...........))))..............---------.. ( -25.10) >DroMoj_CAF1 3922 120 - 1 AUUAUGGCAGCAAUAAAGAAAGUCUUAUCACCUAGCUCUGUUAAUAAUAUAACAGAGAUCGAUGCGGUAAACGCAUCGAUAAAACUGCCCUUGAUUUUCUUCUCCCUUUCCGGUUCAAUA ....(((........(((((((((...........((((((((......))))))))(((((((((.....)))))))))............)))))))))........)))........ ( -33.79) >DroAna_CAF1 447 117 - 1 AUUAUGGCAGCUAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAAAAGGAUAACAGAGAUCGAUGCGGUGAACGCAUCGAUAAAGGUUCCCUUCGUUUUCGCCUGCCUGUCCGAU---GAU .....((((((....(((.....)))...((((..((((((((......))))))))(((((((((.....)))))))))..)))).............).)))))........---... ( -36.30) >consensus AUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAAUAAGAUAACAGAGAUCGAUGCGGUAAAAGCAUCGAUAAAAGUGCCCUUCAUUUUCGCAUCCCGUUCCGGU___G__ .....((............................((((((((......))))))))(((((((((.....)))))))))........)).............................. (-23.77 = -23.43 + -0.33)


| Location | 11,025,037 – 11,025,157 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.50 |
| Mean single sequence MFE | -39.40 |
| Consensus MFE | -34.50 |
| Energy contribution | -34.58 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.49 |
| Structure conservation index | 0.88 |
| SVM decision value | 3.04 |
| SVM RNA-class probability | 0.998228 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
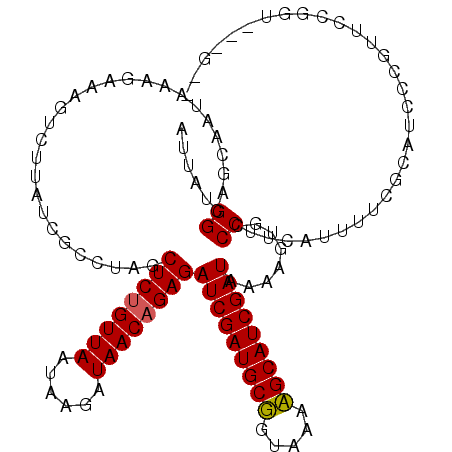
>3R_DroMel_CAF1 11025037 120 + 27905053 AUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUCUUACUAACAGAGCUAGGGGAUAAGACUUUCUUUAUCGCUGCCAUAAUGGCCAUGCGCCAUCCGCGUUUGAUUGUCUUUGGCGGUGCC (((((((((.....)))))))))((((((((......)))))))).(((((((.......))))))).((((((((....(((((.((((.....)))).))...)))..)))))))).. ( -42.70) >DroVir_CAF1 258 120 + 1 AUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUAUUAUUAACAGAGUUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCAAUGCGCCACCCGCGUUUGAUAGUCUUUGGCGGUGCC (((((((((.....)))))))))((((((((......))))))))...(((((((((......)))))))))(((.....)))...(((((.((.((........))....)).))))). ( -43.10) >DroPse_CAF1 352 120 + 1 AUCGAUGCGUUCACAGCAUCGAUAUCUGUUAUACUUUUAACAGAACUAGGCGAUAAGACAUUCUUUAUUGCUGCCAUAAUGGCCAUGCGCCAUCCCCGUUUGAUUGUCUUUGGCGGUGCU ((((((((.......)))))))).(((((((......)))))))....(((((((((......)))))))))(((.....)))...(((((....(((...(.....)..))).))))). ( -38.20) >DroWil_CAF1 349 120 + 1 AUCGAUGCUUUUACAGCAUCGAUCUCUGUUAUCUUAUUAACGGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCCAUGCGUCAUCCCCGUUUAAUUGUCUUUGGCGGUGCA (((((((((.....)))))))))((((((((......))))))))...(((((((((......)))))))))(((.....)))(((.(((((....((......))....)))))))).. ( -36.30) >DroMoj_CAF1 3962 120 + 1 AUCGAUGCGUUUACCGCAUCGAUCUCUGUUAUAUUAUUAACAGAGCUAGGUGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCAAUGCGCCAUCCUCGUCUGAUUGUCUUCCUCGGUGCU (((((((((.....)))))))))((((((((......))))))))...((..(((((......)))))..))(((.....)))...(((((......(.(.....).)......))))). ( -37.90) >DroPer_CAF1 352 120 + 1 AUCGAUGCGUUCACAGCAUCGAUAUCUGUUAUACUUUUAACAGAACUAGGCGAUAAGACAUUCUUUAUUGCUGCCAUAAUGGCCAUGCGCCAUCCCCGUUUGAUUGUCUUUGGCGGUGCU ((((((((.......)))))))).(((((((......)))))))....(((((((((......)))))))))(((.....)))...(((((....(((...(.....)..))).))))). ( -38.20) >consensus AUCGAUGCGUUUACAGCAUCGAUCUCUGUUAUAUUAUUAACAGAGCUAGGCGAUAAGACUUUCUUUAUUGCUGCCAUAAUGGCCAUGCGCCAUCCCCGUUUGAUUGUCUUUGGCGGUGCU (((((((((.....)))))))))((((((((......))))))))...(((((((((......)))))))))(((.....)))...(((((.......................))))). (-34.50 = -34.58 + 0.09)



| Location | 11,025,037 – 11,025,157 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.50 |
| Mean single sequence MFE | -36.18 |
| Consensus MFE | -29.72 |
| Energy contribution | -30.05 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.21 |
| SVM RNA-class probability | 0.930805 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11025037 120 - 27905053 GGCACCGCCAAAGACAAUCAAACGCGGAUGGCGCAUGGCCAUUAUGGCAGCGAUAAAGAAAGUCUUAUCCCCUAGCUCUGUUAGUAAGAUAACAGAGAUCGAUGCGGUAAACGCAUCGAU (((...))).(((((..((...(((.((((((.....))))))......))).....))..))))).........((((((((......))))))))(((((((((.....))))))))) ( -41.80) >DroVir_CAF1 258 120 - 1 GGCACCGCCAAAGACUAUCAAACGCGGGUGGCGCAUUGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAACUCUGUUAAUAAUAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAU (((...))).((((((.((....(((.....))).(((((.....))))).......)).)))))).........((((((((......))))))))(((((((((.....))))))))) ( -42.70) >DroPse_CAF1 352 120 - 1 AGCACCGCCAAAGACAAUCAAACGGGGAUGGCGCAUGGCCAUUAUGGCAGCAAUAAAGAAUGUCUUAUCGCCUAGUUCUGUUAAAAGUAUAACAGAUAUCGAUGCUGUGAACGCAUCGAU ......((..((((((.((.......((((((.....)))))).((....)).....)).))))))...)).....(((((((......))))))).((((((((.(....))))))))) ( -32.70) >DroWil_CAF1 349 120 - 1 UGCACCGCCAAAGACAAUUAAACGGGGAUGACGCAUGGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCCGUUAAUAAGAUAACAGAGAUCGAUGCUGUAAAAGCAUCGAU .......................((.(((((.((...(((.....)))(....).......)).))))).))...(((.((((......)))).)))(((((((((.....))))))))) ( -29.90) >DroMoj_CAF1 3962 120 - 1 AGCACCGAGGAAGACAAUCAGACGAGGAUGGCGCAUUGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCACCUAGCUCUGUUAAUAAUAUAACAGAGAUCGAUGCGGUAAACGCAUCGAU ......((..(((((.(((.......)))...((..((((.....))))))..........))))).))......((((((((......))))))))(((((((((.....))))))))) ( -37.30) >DroPer_CAF1 352 120 - 1 AGCACCGCCAAAGACAAUCAAACGGGGAUGGCGCAUGGCCAUUAUGGCAGCAAUAAAGAAUGUCUUAUCGCCUAGUUCUGUUAAAAGUAUAACAGAUAUCGAUGCUGUGAACGCAUCGAU ......((..((((((.((.......((((((.....)))))).((....)).....)).))))))...)).....(((((((......))))))).((((((((.(....))))))))) ( -32.70) >consensus AGCACCGCCAAAGACAAUCAAACGGGGAUGGCGCAUGGCCAUUAUGGCAGCAAUAAAGAAAGUCUUAUCGCCUAGCUCUGUUAAUAAUAUAACAGAGAUCGAUGCUGUAAACGCAUCGAU ..........(((((.(((.......)))...((...(((.....))).))..........))))).........((((((((......))))))))(((((((((.....))))))))) (-29.72 = -30.05 + 0.33)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:16:22 2006