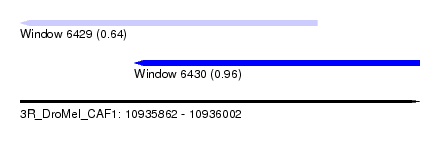
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 10,935,862 – 10,936,002 |
| Length | 140 |
| Max. P | 0.956517 |

| Location | 10,935,862 – 10,935,966 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 81.37 |
| Mean single sequence MFE | -34.94 |
| Consensus MFE | -17.41 |
| Energy contribution | -17.25 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.636850 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10935862 104 - 27905053 UUAAAC--GGG----CUGUGGACUGAGGC-U-AGGUUUAGUUUUAGCUC-UGCGGCCCGAUUAUGGGUUGCUGUUUAUUGCAAAGUCGAGCAAAUAACCCAAUUAGCUCUGAU .....(--(((----(((..(((((((((-(-(....))))))))).))-..)))))))....(((((((.((((((((....))).)))))..)))))))............ ( -39.40) >DroSim_CAF1 12754 104 - 1 UUAAAC--GGG----CUGUGGACUGAGGC-U-AGGUUUAGUUUUAGCUC-UGCGGCCCGAUUAUGGGUUGCUGUUUAUUGCAAAGUCGAGCAAAUAACCCAAUUAGCUCUGAU .....(--(((----(((..(((((((((-(-(....))))))))).))-..)))))))....(((((((.((((((((....))).)))))..)))))))............ ( -39.40) >DroEre_CAF1 5317 104 - 1 UUAAAC--GGG----CUGUGGAUUGGGGC-U-AGGUUUAGUUUUAGCUC-UGCGGCCCGAUUAUGGGCUGCUGUUUAUUGCAAAGUCGAGCAAAUAACCCAAUUAGCUCUGAU ......--(((----((...(((((((((-(-(((......))))))..-.(((((((......)))))))......((((........))))....)))))))))))).... ( -36.70) >DroWil_CAF1 4926 112 - 1 UUGCAUUGGGGGGUUUUGGGGUUUGAGGUCUGGGGUGUGGGUUUUGCUCUUGUGGCCCGAUUACU-GCUGCAGUUUAUUGCAUAGUCGAGCAAAUAACCCAAUUAGCGUUGAU ..((((((((..((((....((((((((((.(((.((..((.......))..)).))))))).((-(.(((((....))))))))))))))))))..))))))..))...... ( -32.20) >DroYak_CAF1 13067 104 - 1 UUAAAU--GGG----CCGUGGACUGAGGC-U-AGAUUUAGUUUUAGCUC-UGCGGCCCGAUUAUGGGCUGCUGUUUAUUGCAAAGUCGAGCAAAUAACCCAAUUAGCUCUGAU .(((.(--(((----(((..(((((((((-(-(....))))))))).))-..))))))).)))..((((..(((.....))).))))((((.(((......))).)))).... ( -39.60) >DroAna_CAF1 5212 98 - 1 UCGACU--GAG----CUGUUGAUUGGGGU-U-GGGUUCGGUU------C-UUUGGGAAGAUUAUGGGUUGCUGUUUAGUGCAUAGUCGAGCAAAUAACUCAAUUAGUCUUGAU ..((((--((.----..........((((-(-(.((((..((------(-.....)))(((((((.((((.....)))).)))))))))))...))))))..))))))..... ( -22.32) >consensus UUAAAC__GGG____CUGUGGACUGAGGC_U_AGGUUUAGUUUUAGCUC_UGCGGCCCGAUUAUGGGCUGCUGUUUAUUGCAAAGUCGAGCAAAUAACCCAAUUAGCUCUGAU ........(((........((((((((.(....).))))))))..((((..((((((((....))))))))(((.....))).....))))......)))............. (-17.41 = -17.25 + -0.16)

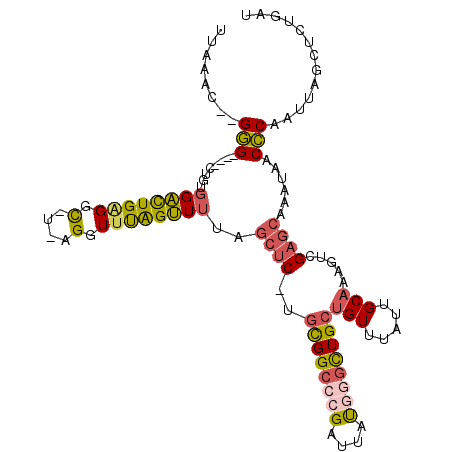
| Location | 10,935,902 – 10,936,002 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 81.26 |
| Mean single sequence MFE | -38.68 |
| Consensus MFE | -30.80 |
| Energy contribution | -30.87 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.40 |
| Mean z-score | -3.10 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.47 |
| SVM RNA-class probability | 0.956517 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10935902 100 - 27905053 AUAGUUCACUUGUU--CGAAGCUCCACUGUAGUGGCUUUUAAACGGGCUGUGGACUGAGGCUAGGUUUAGUUUUAGCUCUGCGGCCCGAUUAUGGGUUGCUG (((((...(((...--..)))....)))))((..(((..(((.(((((((..(((((((((((....))))))))).))..))))))).)))..)))..)). ( -39.70) >DroSec_CAF1 5336 100 - 1 AUGGUUCACUUGCU--CGAAGCUCUACUGUAGUGGCUUUUAAACGGGCUGUGGACUGAGGCUAGGUUUAGUUUUAGCUCUGCGGCCCGAUUAUGGGUUGCUG (((((......((.--....))...)))))((..(((..(((.(((((((..(((((((((((....))))))))).))..))))))).)))..)))..)). ( -40.00) >DroSim_CAF1 12794 100 - 1 AUGGUUCACUUGCU--CGAAGCUCCACUGUAGUGGCUUUUAAACGGGCUGUGGACUGAGGCUAGGUUUAGUUUUAGCUCUGCGGCCCGAUUAUGGGUUGCUG .(((....(((...--..)))..)))...(((..(((..(((.(((((((..(((((((((((....))))))))).))..))))))).)))..)))..))) ( -39.90) >DroEre_CAF1 5357 100 - 1 AUGGUACACUUGGU--CGAGGUUCCACUGUAGCUGCCUUUAAACGGGCUGUGGAUUGGGGCUAGGUUUAGUUUUAGCUCUGCGGCCCGAUUAUGGGCUGCUG .(((.((.(((...--.))))).)))...((((.((((.(((.(((((((..(((((((((((....))))))))).))..))))))).))).)))).)))) ( -42.80) >DroYak_CAF1 13107 100 - 1 UUGGUUCACUUGAU--AGAGGUUCCACUGUAGCUGCCUUUAAAUGGGCCGUGGACUGAGGCUAGAUUUAGUUUUAGCUCUGCGGCCCGAUUAUGGGCUGCUG .(((..(.(((...--.))))..)))...((((.((((.(((.(((((((..(((((((((((....))))))))).))..))))))).))).)))).)))) ( -45.50) >DroAna_CAF1 5252 95 - 1 A-AGCUCCUCUGUUAGCCACGUUCCACGGUACUGGGCUUCGACUGAGCUGUUGAUUGGGGUUGGGUUCGGUU------CUUUGGGAAGAUUAUGGGUUGCUG .-(((..(((...((..(...(((((.((.((((((((.(((((...(((.....)))))))))))))))).------)).))))).)..)).)))..))). ( -24.20) >consensus AUGGUUCACUUGCU__CGAAGCUCCACUGUAGUGGCCUUUAAACGGGCUGUGGACUGAGGCUAGGUUUAGUUUUAGCUCUGCGGCCCGAUUAUGGGUUGCUG .............................(((((((((.(((.(((((((..(((((((((((....))))))))).))..))))))).))).))))))))) (-30.80 = -30.87 + 0.06)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:50 2006