
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 10,671,774 – 10,672,094 |
| Length | 320 |
| Max. P | 0.989125 |

| Location | 10,671,774 – 10,671,894 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.00 |
| Mean single sequence MFE | -48.02 |
| Consensus MFE | -39.22 |
| Energy contribution | -39.78 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.82 |
| SVM decision value | 2.02 |
| SVM RNA-class probability | 0.985691 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10671774 120 + 27905053 CCCGUUACGGCGGCCUAUGUGUCAAAGGCACGGAAACGACGCCUUGAUGAGAUCGAGGCCAGGGACAUUAAUGCCGCUCGGCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAG .((.....((((((....(((((.......((....))..((((((((...))))))))....)))))....)))))).((((((((.....))))))))(((((......))))))).. ( -53.10) >DroSec_CAF1 13208 120 + 1 CCCGUUACGGCUGCCUAUGUGUCAAAGGCGCGGAAACGACGCCUUGAUGAGAUCGAGGCUAGGGACAUUAAUGCCGCUCGCCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAA ..((...((((.((((..((.(((((((((((....)).))))))..))).))..)))).............))))..)).((((((.....))))))(((((((......))))))).. ( -47.74) >DroSim_CAF1 14754 120 + 1 CCCGUUACGGCUGCCUAUGUGUCUAAGGCGCGGAAACGACGCCUUGAUGAGAUCGAGGCUAGGGACAUUAAUGCCGCUCGCCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAA ..((...((((.....((((.((((....(((....)).)((((((((...)))))))))))).))))....))))..)).((((((.....))))))(((((((......))))))).. ( -47.80) >DroEre_CAF1 10475 120 + 1 CCCGUUACGGCGGCCUAUGUGUCCAAGGCACGGAAACGACACCAUCAAGAGAUCGAGGCCAGGGACAUAAAUGCAGCUCGGCUGGGUUUCCAGCUCAGUCAGAUCAAGCACGAUCUGGAG .((((....))))....(((((((..(((.((....))...(.(((....))).)..)))..)))))))......(((.((((((....)))))).)))((((((......))))))... ( -43.10) >DroYak_CAF1 5134 120 + 1 CCCGUUACGGCGGCCUAUGUGUCCAAGGCACAGAAACGACGCCUUCAAGAAAUCGAGGCCAGGGACAUAAAUGCCGCUCGGCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAG .((.....((((((...(((((((..(((...((..................))...)))..)))))))...)))))).((((((((.....))))))))(((((......))))))).. ( -48.37) >consensus CCCGUUACGGCGGCCUAUGUGUCAAAGGCACGGAAACGACGCCUUGAUGAGAUCGAGGCCAGGGACAUUAAUGCCGCUCGGCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAG .......((((.((....((((((..(((.((....))....(((((.....))))))))..))))))....)).)).)).((((((.....))))))(((((((......))))))).. (-39.22 = -39.78 + 0.56)



| Location | 10,671,814 – 10,671,934 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.67 |
| Mean single sequence MFE | -45.52 |
| Consensus MFE | -37.10 |
| Energy contribution | -38.70 |
| Covariance contribution | 1.60 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976665 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10671814 120 + 27905053 GCCUUGAUGAGAUCGAGGCCAGGGACAUUAAUGCCGCUCGGCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAGUCCCGAGAUCAAGUCAUCACCAGUCUGCUAAUCCGCGAGC ((((((((...))))))))..(((((.........(((.((((((....)))))).)))((((((......))))))..)))))(((((...))).))....(..(((......)))..) ( -43.80) >DroSec_CAF1 13248 120 + 1 GCCUUGAUGAGAUCGAGGCUAGGGACAUUAAUGCCGCUCGCCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAAUCCCGAGAUCAAUUGAUCACCAGUCUGCUGAUCCGCGAGC ((((((((...))))))))...((.((....))))((((((((((((.....))))))...(((((.(((.((.((((..(....)((((....)))).)))))))))))))).)))))) ( -46.50) >DroSim_CAF1 14794 120 + 1 GCCUUGAUGAGAUCGAGGCUAGGGACAUUAAUGCCGCUCGCCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAAUCCCGAGAUCAAUUGAUCACCAGUCUGCUGAUCCGCGAGC ((((((((...))))))))...((.((....))))((((((((((((.....))))))...(((((.(((.((.((((..(....)((((....)))).)))))))))))))).)))))) ( -46.50) >DroEre_CAF1 10515 120 + 1 ACCAUCAAGAGAUCGAGGCCAGGGACAUAAAUGCAGCUCGGCUGGGUUUCCAGCUCAGUCAGAUCAAGCACGAUCUGGAGUCCCGAGAUAAACUGAUCACCAGUCUGCUGAUCCGCGAGC ....((..(.(((((((((..(((((.........(((.((((((....)))))).)))((((((......))))))..)))))..(((......)))....))))..))))))..)).. ( -37.90) >DroYak_CAF1 5174 120 + 1 GCCUUCAAGAAAUCGAGGCCAGGGACAUAAAUGCCGCUCGGCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAGUCCCGGGAUCAGCUGAUCACCAGUCUGCUGAUCCGCGAGC (((((..(....).)))))..(((((.........(((.((((((....)))))).)))((((((......))))))..))))).((((((((.(((.....))).))))))))...... ( -52.90) >consensus GCCUUGAUGAGAUCGAGGCCAGGGACAUUAAUGCCGCUCGGCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAGUCCCGAGAUCAACUGAUCACCAGUCUGCUGAUCCGCGAGC (((((((.....)))))))...((.((....))))((((((((((((.....)))))))).(((((.(((.((.((((..(....)((((....)))).))))))))))))))...)))) (-37.10 = -38.70 + 1.60)


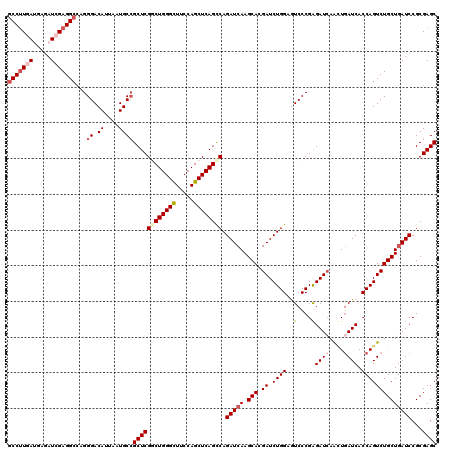
| Location | 10,671,814 – 10,671,934 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.67 |
| Mean single sequence MFE | -45.82 |
| Consensus MFE | -37.96 |
| Energy contribution | -39.28 |
| Covariance contribution | 1.32 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.39 |
| SVM RNA-class probability | 0.717208 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10671814 120 - 27905053 GCUCGCGGAUUAGCAGACUGGUGAUGACUUGAUCUCGGGACUCCAGAUCGUGCUUGAUCUGGCUGAGCUGGAAGCCCAGCCGAGCGGCAUUAAUGUCCCUGGCCUCGAUCUCAUCAAGGC (((.((......))....((((((.((.((((.((.(((((..(((((((....)))))))((((.(((((....)))))....))))......))))).))..)))))))))))).))) ( -43.60) >DroSec_CAF1 13248 120 - 1 GCUCGCGGAUCAGCAGACUGGUGAUCAAUUGAUCUCGGGAUUCCAGAUCGUGCUUGAUCUGGCUGAGCUGGAAGCCCAGGCGAGCGGCAUUAAUGUCCCUAGCCUCGAUCUCAUCAAGGC ((((((...(((((...((((.((((....)))))))).....(((((((....)))))))))))).((((....))))))))))((((....))))....((((.(((...))).)))) ( -48.00) >DroSim_CAF1 14794 120 - 1 GCUCGCGGAUCAGCAGACUGGUGAUCAAUUGAUCUCGGGAUUCCAGAUCGUGCUUGAUCUGGCUGAGCUGGAAGCCCAGGCGAGCGGCAUUAAUGUCCCUAGCCUCGAUCUCAUCAAGGC ((((((...(((((...((((.((((....)))))))).....(((((((....)))))))))))).((((....))))))))))((((....))))....((((.(((...))).)))) ( -48.00) >DroEre_CAF1 10515 120 - 1 GCUCGCGGAUCAGCAGACUGGUGAUCAGUUUAUCUCGGGACUCCAGAUCGUGCUUGAUCUGACUGAGCUGGAAACCCAGCCGAGCUGCAUUUAUGUCCCUGGCCUCGAUCUCUUGAUGGU (((((((((((((((((((((.....(((((......))))))))).)).))).))))))).....(((((....))))))))))................(((((((....)))).))) ( -39.20) >DroYak_CAF1 5174 120 - 1 GCUCGCGGAUCAGCAGACUGGUGAUCAGCUGAUCCCGGGACUCCAGAUCGUGCUUGAUCUGGCUGAGCUGGAAGCCCAGCCGAGCGGCAUUUAUGUCCCUGGCCUCGAUUUCUUGAAGGC ....(.((((((((.(((....).)).)))))))))(((((..(((((((....)))))))((((.(((((....)))))....))))......)))))..(((((((....))).)))) ( -50.30) >consensus GCUCGCGGAUCAGCAGACUGGUGAUCAAUUGAUCUCGGGACUCCAGAUCGUGCUUGAUCUGGCUGAGCUGGAAGCCCAGCCGAGCGGCAUUAAUGUCCCUGGCCUCGAUCUCAUCAAGGC (((((....(((((...((((.((((....)))))))).....(((((((....))))))))))))(((((....))))))))))((((....))))....((((.((.....)).)))) (-37.96 = -39.28 + 1.32)



| Location | 10,671,854 – 10,671,974 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.42 |
| Mean single sequence MFE | -44.82 |
| Consensus MFE | -38.86 |
| Energy contribution | -38.90 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.53 |
| SVM RNA-class probability | 0.961466 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10671854 120 + 27905053 GCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAGUCCCGAGAUCAAGUCAUCACCAGUCUGCUAAUCCGCGAGCACAAGGCCAAGGAGAACGCGAAGGCCUUGGAACAGGCUAG (((((((.....)))))))((((((......))))))..............((((.......(..(((......)))..).(((((((..............))))))).....)))).. ( -38.64) >DroSec_CAF1 13288 120 + 1 CCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAAUCCCGAGAUCAAUUGAUCACCAGUCUGCUGAUCCGCGAGCACAAGGCCAAGGAGAACGCGAAGGCCUUGGAACAGGCCAG .((((.(((((.((((.((..(((((.(((.((.((((..(....)((((....)))).)))))))))))))).))))))..((((((..............)))))))))..)).)))) ( -44.34) >DroSim_CAF1 14834 120 + 1 CCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAAUCCCGAGAUCAAUUGAUCACCAGUCUGCUGAUCCGCGAGCACAAGGCCAAGGAGAACGCGAAGGCCUUGGAACAGGCCAG .((((.(((((.((((.((..(((((.(((.((.((((..(....)((((....)))).)))))))))))))).))))))..((((((..............)))))))))..)).)))) ( -44.34) >DroEre_CAF1 10555 120 + 1 GCUGGGUUUCCAGCUCAGUCAGAUCAAGCACGAUCUGGAGUCCCGAGAUAAACUGAUCACCAGUCUGCUGAUCCGCGAGCACAAGGCCAAGGAGAACGCGAUGGCCUUGGAGCAGGCGAG (((((....)))))(((((((((((......))))))..(((....)))..)))))......(((((((....(....)..((((((((..(......)..)))))))).)))))))... ( -43.80) >DroYak_CAF1 5214 120 + 1 GCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAGUCCCGGGAUCAGCUGAUCACCAGUCUGCUGAUCCGCGAGCAGAAGGCCAAGGAGAACGCGCUGGCUUUGGAGCAGGCCAG (((((.((((..((((..(((((((......))))))).......((((((((.(((.....))).))))))))..)))).)))).))).(.....))).(((((((......))))))) ( -53.00) >consensus GCUGGGCUUCCAGCUCAGCCAGAUCAAGCACGAUCUGGAGUCCCGAGAUCAACUGAUCACCAGUCUGCUGAUCCGCGAGCACAAGGCCAAGGAGAACGCGAAGGCCUUGGAACAGGCCAG .((((.(((((.((((.((..(((((.(((.((.((((..(....)((((....)))).)))))))))))))).))))))..((((((..............)))))))))..)).)))) (-38.86 = -38.90 + 0.04)

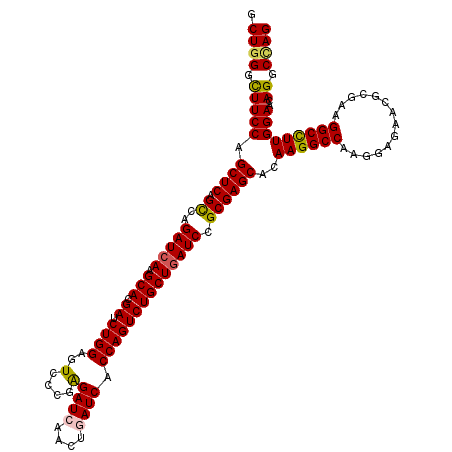
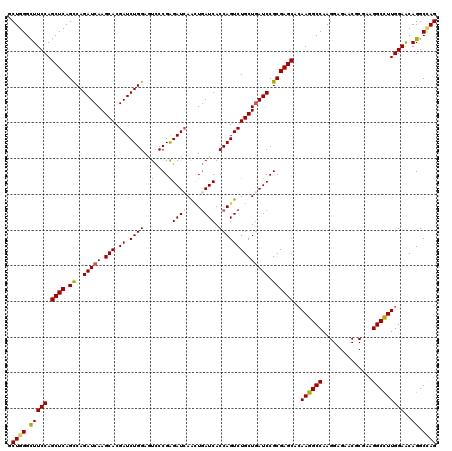
| Location | 10,671,974 – 10,672,094 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -42.55 |
| Consensus MFE | -37.05 |
| Energy contribution | -37.89 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.15 |
| SVM RNA-class probability | 0.989125 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10671974 120 - 27905053 CACAUGGGAUCCUUGGGCAUGACCAGGGCAUCCUGCACACCCUUAUCUCGGAAGAACUUCUGCUCUUCCCGCUGGCUGAGGAGCUGUUCGCGAAUCUCCUGCUUCUUGGCCAUUUUGGUU .....(((((((((((......)))))).)))))....(((........((((((.(....).))))))...((((..((((((................))))))..))))....))). ( -43.29) >DroSec_CAF1 13408 120 - 1 CACAUGGGAUCCUUGGGCAUGACCAGGGCAUCCUGCACUCCUUUAUCUCGGAAGAACUUCUGCUCUUCCCGCUGGCUGAGGAGCUGUUCGCGAAUCUCCUGCUUCUUGGCCAUUUUGGUU .....(((((((((((......)))))).))))).....((........((((((.(....).))))))...((((..((((((................))))))..))))....)).. ( -42.29) >DroSim_CAF1 14954 120 - 1 CACAUGGGAUCCUUGGGCAUGAGCAGGGCAUCCUGCACACCCUUAUCUCGGAAGAACUUCUGCUCUUCCCGCUGGCUGAGGAGCUGUUCGCGAAUCUCCUGCUUCUUGGCCAUUUUGGUU .....(((((....(((..((.(((((....))))).)))))..)))))((((((.(....).))))))...((((..((((((................))))))..))))........ ( -42.69) >DroEre_CAF1 10675 120 - 1 CACAUGGGAUCCUUUGGCAUGACCAGGGCAUCCUGCACUCCCUUAUCGCGGAAGAACUUCUGCUCUUCCCGCUGGCUGAGCAGCUGUUCGCGGAUCUCCUGCUUCUUGGCCAUUUUGGUU ..........((..((((..((.(((((.((((.((..(((........))).((((..((((((..((....))..))))))..)))))))))).)))))..))...))))....)).. ( -41.70) >DroYak_CAF1 5334 120 - 1 CACAUGGGAUCCUUGGGCAUGACCAGGGCAUCCUUCACUGCCUUGUCUCGGAAGAACUUCUGCUCCUCCCGCUGGCUGAGCAGCUGUUCGCGGAUCUCCUGCUUCUUGGCCAUCUUGGUU .....(((((((((((......)))))).))))).....(((.((.((.((((((((..((((((..((....))..))))))..))))((((.....)))))))).)).))....))). ( -42.80) >consensus CACAUGGGAUCCUUGGGCAUGACCAGGGCAUCCUGCACUCCCUUAUCUCGGAAGAACUUCUGCUCUUCCCGCUGGCUGAGGAGCUGUUCGCGAAUCUCCUGCUUCUUGGCCAUUUUGGUU .....(((((((((((......)))))).)))))......((.......((((((.(....).))))))...((((..((((((................))))))..))))....)).. (-37.05 = -37.89 + 0.84)

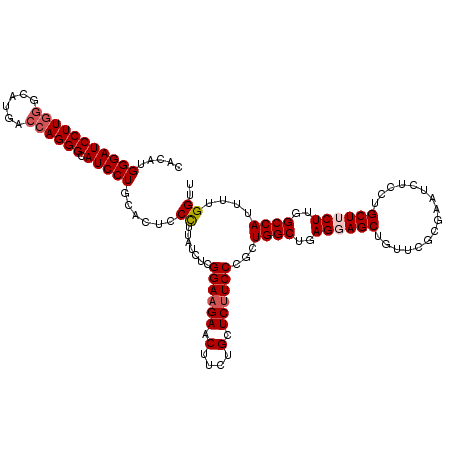

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:13:54 2006