
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 10,498,649 – 10,498,772 |
| Length | 123 |
| Max. P | 0.966626 |

| Location | 10,498,649 – 10,498,742 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 80.50 |
| Mean single sequence MFE | -27.27 |
| Consensus MFE | -15.93 |
| Energy contribution | -17.60 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.58 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.903747 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10498649 93 + 27905053 AGCAC--AAGCACGUCUAAUUAAUGCAAGCCGC------CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC--------UUGUGCUCAGCAA (((((--(((..............(((.(..(.------((((((((...)))))))).)...).)))..((((((.....)))))))--------)))))))...... ( -27.90) >DroSec_CAF1 147434 93 + 1 AGCAC--AAGCACGUCUAAUUAAUGCAAGCCGC------CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC--------UUGUGCUUAGCAA (((((--(((..............(((.(..(.------((((((((...)))))))).)...).)))..((((((.....)))))))--------)))))))...... ( -27.90) >DroSim_CAF1 45343 93 + 1 AGCAC--AAGCACGUCUAAUUAAUGCAAGCCGC------CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC--------UUGUGCUUAGCAA (((((--(((..............(((.(..(.------((((((((...)))))))).)...).)))..((((((.....)))))))--------)))))))...... ( -27.90) >DroEre_CAF1 45694 90 + 1 AG-----AAACACGUCUAAUUAAUGCAAGCCGG------CAGACAUCGCAGAUGUCUGACAACCCUGGUAUGUAUCUCGCAGAUACAG--------AUGCGCUUAGCAA ..-----................(((((((.(.------((((((((...)))))))).).......(((((((((.....)))))).--------.))))))).))). ( -24.20) >DroYak_CAF1 45253 98 + 1 AG-----AAACACGUCUAAUUAAUGCAAGCCGG------CAGACAGCGCCGAUGUCUGACAACCCUGGUAUGUAUCUCGCAGAUACAGAUACACAGAUACGCUUAGCAA ..-----................((((((((((------(.......)))).((((((...((....)).((((((.....))))))......)))))).)))).))). ( -25.50) >DroAna_CAF1 39175 99 + 1 UGCCCAGAAAGGCGUCUAAUUAAGGCA-GCUGCCAGCAGCAGAACACGCAGAUGUCUGAG-ACCGGGCUGAGCAUCUUGCAGAUACAG--------AUGCGAUUCGCAU .((((.(..((((((((......(((.-...)))....((.......))))))))))...-.).))))...((((((.(......)))--------))))......... ( -30.20) >consensus AGCAC__AAACACGUCUAAUUAAUGCAAGCCGC______CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC________AUGCGCUUAGCAA .......................(((((((.........((((((((...))))))))............((((((.....)))))).............)))).))). (-15.93 = -17.60 + 1.67)

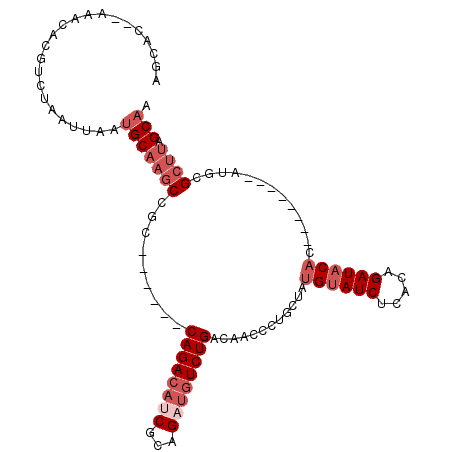
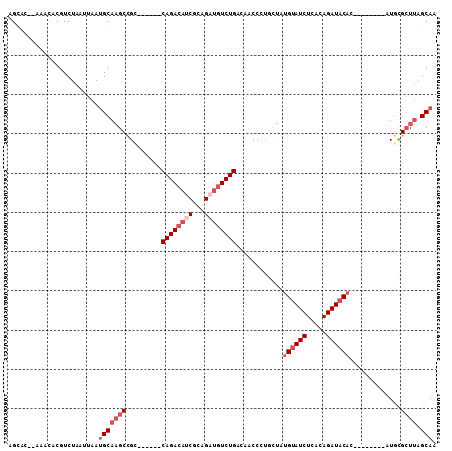
| Location | 10,498,649 – 10,498,742 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 80.50 |
| Mean single sequence MFE | -35.15 |
| Consensus MFE | -19.25 |
| Energy contribution | -21.12 |
| Covariance contribution | 1.86 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.42 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966626 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10498649 93 - 27905053 UUGCUGAGCACAA--------GUGUAUCUGUGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG------GCGGCUUGCAUUAAUUAGACGUGCUU--GUGCU ..(((((((((..--------(((((((.....))))))).(((((.((((((((((((...))))))))------)))))))))...........))))))--).)). ( -41.60) >DroSec_CAF1 147434 93 - 1 UUGCUAAGCACAA--------GUGUAUCUGUGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG------GCGGCUUGCAUUAAUUAGACGUGCUU--GUGCU ..(((((((((..--------(((((((.....))))))).(((((.((((((((((((...))))))))------)))))))))...........))))))--).)). ( -42.50) >DroSim_CAF1 45343 93 - 1 UUGCUAAGCACAA--------GUGUAUCUGUGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG------GCGGCUUGCAUUAAUUAGACGUGCUU--GUGCU ..(((((((((..--------(((((((.....))))))).(((((.((((((((((((...))))))))------)))))))))...........))))))--).)). ( -42.50) >DroEre_CAF1 45694 90 - 1 UUGCUAAGCGCAU--------CUGUAUCUGCGAGAUACAUACCAGGGUUGUCAGACAUCUGCGAUGUCUG------CCGGCUUGCAUUAAUUAGACGUGUUU-----CU .....((((((.(--------(((((((.....)))))......((((((.((((((((...))))))))------.)))))).........))).))))))-----.. ( -27.50) >DroYak_CAF1 45253 98 - 1 UUGCUAAGCGUAUCUGUGUAUCUGUAUCUGCGAGAUACAUACCAGGGUUGUCAGACAUCGGCGCUGUCUG------CCGGCUUGCAUUAAUUAGACGUGUUU-----CU .....(((((..(((((((((((((....)).))))))))....((((((.((((((.......))))))------.)))))).........)))..)))))-----.. ( -26.50) >DroAna_CAF1 39175 99 - 1 AUGCGAAUCGCAU--------CUGUAUCUGCAAGAUGCUCAGCCCGGU-CUCAGACAUCUGCGUGUUCUGCUGCUGGCAGC-UGCCUUAAUUAGACGCCUUUCUGGGCA .........((((--------((((....)).)))))).(((((((((-..(((((((....))).))))..)))))..))-))............((((....)))). ( -30.30) >consensus UUGCUAAGCACAA________CUGUAUCUGCGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG______GCGGCUUGCAUUAAUUAGACGUGCUU__GUGCU ..((.((((((...........((((((.....))))))..(((((.((((((((((((...))))))))......)))))))))...........))))))....)). (-19.25 = -21.12 + 1.86)

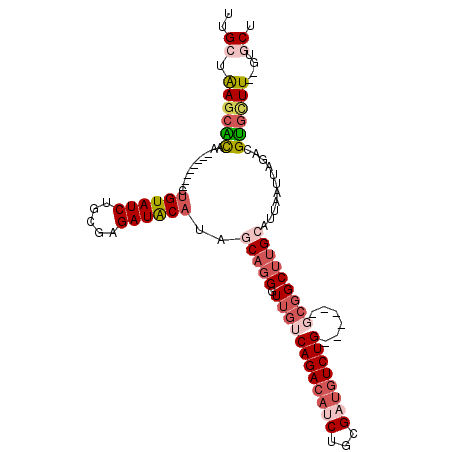

| Location | 10,498,680 – 10,498,772 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -31.23 |
| Consensus MFE | -17.04 |
| Energy contribution | -18.43 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.923719 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10498680 92 + 27905053 -CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC--------UUGUGCUCAGCAAUUGCAUU--GCAGGGGGAUACACAGUCGC------GGU -((((((((...))))))))...((((.(((.((((((((((((......--------)))))(((.(((((...)))--)).)))))))))).))).).------))) ( -30.90) >DroSec_CAF1 147465 91 + 1 -CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC--------UUGUGCUUAGCAAUUGCAU---GCAGGGCGAUACACAGUCGA------GGU -((((((((...))))))))....((((((..((((((.....)))))).--------..((((.........))))---))))))((((.....)))).------... ( -32.30) >DroSim_CAF1 45374 91 + 1 -CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC--------UUGUGCUUAGCAAUUGCAU---GCAGGGCGAUACACAGUCGC------GGU -((((((((...)))))))).....(((((..((((((.....)))))).--------..((((.........))))---)))))(((((.....)))))------... ( -33.50) >DroEre_CAF1 45722 91 + 1 -CAGACAUCGCAGAUGUCUGACAACCCUGGUAUGUAUCUCGCAGAUACAG--------AUGCGCUUAGCAAUUGCAU---GCAGAGCGAAACACAGUCAC------AGU -((((((((...))))))))......(((...((((((.....))))))(--------((.(((((.(((......)---)).))))).......))).)------)). ( -28.30) >DroYak_CAF1 45281 99 + 1 -CAGACAGCGCCGAUGUCUGACAACCCUGGUAUGUAUCUCGCAGAUACAGAUACACAGAUACGCUUAGCAAUUGCAU---GCAGGGCGAAACACAGUCGC------GGU -........((((.((.(((......((((((((((((.....))))))..))).)))...((((..(((......)---))..)))).....))).)))------))) ( -29.70) >DroAna_CAF1 39212 100 + 1 GCAGAACACGCAGAUGUCUGAG-ACCGGGCUGAGCAUCUUGCAGAUACAG--------AUGCGAUUCGCAUCUUCACUUACCAGUGGAAGACACGCCCGCCUCUGCGGC (((((...(.(((....))).)-..(((((((.((.....)).......(--------(((((...))))))((((((....))))))...)).)))))..)))))... ( -32.70) >consensus _CAGACAUCGCAGAUGUCUGACAACCCUGCUAUGUAUCUCACAGAUACAC________AUGCGCUUAGCAAUUGCAU___GCAGGGCGAUACACAGUCGC______GGU .((((((((...))))))))....(((((...((((((.....)))))).............((.........))......)))))....................... (-17.04 = -18.43 + 1.39)


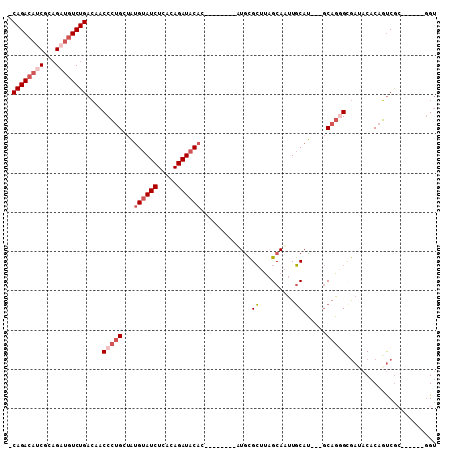
| Location | 10,498,680 – 10,498,772 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -32.98 |
| Consensus MFE | -17.38 |
| Energy contribution | -18.27 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831332 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10498680 92 - 27905053 ACC------GCGACUGUGUAUCCCCCUGC--AAUGCAAUUGCUGAGCACAA--------GUGUAUCUGUGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG- ...------((((((.(((.......(((--...((....))...)))...--------(((((((.....))))))).))).))))))((((((((...))))))))- ( -30.10) >DroSec_CAF1 147465 91 - 1 ACC------UCGACUGUGUAUCGCCCUGC---AUGCAAUUGCUAAGCACAA--------GUGUAUCUGUGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG- ...------.............(((((((---.(((.........)))...--------(((((((.....))))))).)))))))...((((((((...))))))))- ( -30.70) >DroSim_CAF1 45374 91 - 1 ACC------GCGACUGUGUAUCGCCCUGC---AUGCAAUUGCUAAGCACAA--------GUGUAUCUGUGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG- ...------((((((.(((...((...((---(......)))...))....--------(((((((.....))))))).))).))))))((((((((...))))))))- ( -31.50) >DroEre_CAF1 45722 91 - 1 ACU------GUGACUGUGUUUCGCUCUGC---AUGCAAUUGCUAAGCGCAU--------CUGUAUCUGCGAGAUACAUACCAGGGUUGUCAGACAUCUGCGAUGUCUG- (((------.((..((((((((((...(.---(((((..(((.....))).--------.)))))).))))))))))...)).)))...((((((((...))))))))- ( -32.80) >DroYak_CAF1 45281 99 - 1 ACC------GCGACUGUGUUUCGCCCUGC---AUGCAAUUGCUAAGCGUAUCUGUGUAUCUGUAUCUGCGAGAUACAUACCAGGGUUGUCAGACAUCGGCGCUGUCUG- ...------(((.((((((((.((((((.---((((.........))))...(((((((((((....)).))))))))).))))))....))))).))))))......- ( -30.90) >DroAna_CAF1 39212 100 - 1 GCCGCAGAGGCGGGCGUGUCUUCCACUGGUAAGUGAAGAUGCGAAUCGCAU--------CUGUAUCUGCAAGAUGCUCAGCCCGGU-CUCAGACAUCUGCGUGUUCUGC ((((((((..(((((((((((((.(((....))))))))))).....((((--------((((....)).))))))...)))))((-(...))).)))))).))..... ( -41.90) >consensus ACC______GCGACUGUGUAUCGCCCUGC___AUGCAAUUGCUAAGCACAA________CUGUAUCUGCGAGAUACAUAGCAGGGUUGUCAGACAUCUGCGAUGUCUG_ ......................((((((.....(((.........)))............((((((.....))))))...))))))...((((((((...)))))))). (-17.38 = -18.27 + 0.89)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:11:37 2006