
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 10,343,332 – 10,343,452 |
| Length | 120 |
| Max. P | 0.500000 |

| Location | 10,343,332 – 10,343,452 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.83 |
| Mean single sequence MFE | -43.15 |
| Consensus MFE | -32.20 |
| Energy contribution | -32.15 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.75 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10343332 120 + 27905053 GGUGCUCCAUCGUGCUAGUGGUGUACAAAAUGGCCAGCGGGGACUCCGUUUUGGUCUGGAAUGCAUUGUUGGUGCCACGAUGAUCCAAGUCAUGGCACAAACAGGCCACCAACAGACCCA ((.(.(((.((((((((.((.....))...))))..)))))))).)).....((((((....((.(((((.((((((.(((.......))).)))))).)))))))......)))))).. ( -41.90) >DroVir_CAF1 6070 120 + 1 GAUGCUCCAUGGUGCUGGUGGUAUAGAGUAUAGCCAAUGGGGAUUCGGUUUUGGUUUGGAAUGCAUUGUUGGUGCCGCGAUGAUCGAGAUCGUGACAUAAACAGGCCACCAGCAGGCCAA .........(((((((((((((.....((((..(((((.(((((...))))).).)))).)))).(((((.(((.((((((.......)))))).))).))))))))))))))..)))). ( -40.50) >DroGri_CAF1 8286 120 + 1 GAUGCUCCAUGGUGCUGGUUGUGUAUAAAAUAGCUAGAGGUGAUUCAGUUUUGGUCUGGAAGGCAUUGUUGGUGCCGCGAUGAUCCAGAUCGUGGCAUAAACAGGCCACCAACAGACCCA ((..(.((......((((((((.......)))))))).)).)..))......((((((...(((.(((((.((((((((((.......)))))))))).)))))))).....)))))).. ( -43.40) >DroWil_CAF1 5937 120 + 1 GAUGCUCCAUUGUGCUGGUCGUAUACAAUAUGGCCAAUGGGGAUUCGGUUUUGGUUUGGAAUGCAUUGUUAGUGCCACGAUGAUCUAGAUCGUGACAUAAACAUGCCACCAAUAGACCCA ((..((((((.....(((((((((...)))))))))))))))..))(((((....((((...((((.(((.(((.((((((.......)))))).))).)))))))..)))).))))).. ( -39.70) >DroAna_CAF1 2241 120 + 1 GGUGCUCCAUGGUACUGGUGGUGUACAGAAUGGCCAGCGGAGACUCGGUUUUGGUUUGGAACGCGUUGUUGGUGCCGCGGUGGUCCAGGUCGUGGCACAAGCAGGCCACCAGCAGGCCCA ((....)).(((..(((.(((((..((((..((((.(.(....).)))))....))))....((.(((((.(((((((((.........))))))))).)))))))))))).)))..))) ( -46.30) >DroPer_CAF1 6827 120 + 1 GGUGCUCCAUCGUGCUGGUCGUGUAGAGAAUGGCCAGCGGGGACUCGGUCUUUGUCUGAAAGGCGUUAUUGGUGCCUCGAUGGUCGAGGUCAUGGCACAGGCAGGCCACCAGCAGGCCCA .((((((((((.((((((((((.......)))))))))).)).....((((((.....)))))).....))).((((((.....))))))...))))).(((..((.....))..))).. ( -47.10) >consensus GAUGCUCCAUGGUGCUGGUGGUGUACAAAAUGGCCAGCGGGGACUCGGUUUUGGUCUGGAAUGCAUUGUUGGUGCCGCGAUGAUCCAGAUCGUGGCACAAACAGGCCACCAACAGACCCA ((..((((.....((((((.((.......)).))))))))))..))......((((((....((.(((((.((((((((((.......)))))))))).)))))))......)))))).. (-32.20 = -32.15 + -0.05)

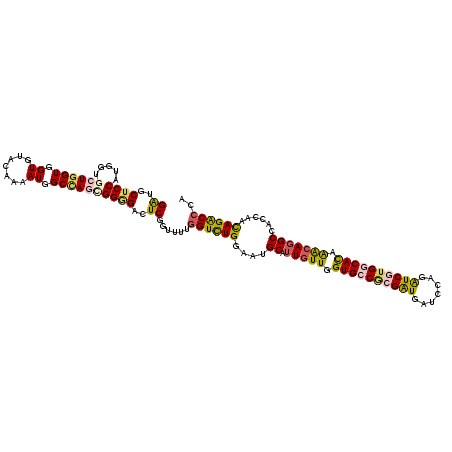
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:10:39 2006