
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 10,283,732 – 10,283,864 |
| Length | 132 |
| Max. P | 0.994522 |

| Location | 10,283,732 – 10,283,852 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.53 |
| Mean single sequence MFE | -34.64 |
| Consensus MFE | -25.94 |
| Energy contribution | -25.78 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.740198 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10283732 120 + 27905053 AAUUAUUUCUGAGACACAAGUAUGCCUCAUUUAAGAGACGCAACAUGUGUGGGUCCUAAACUAGUGACCCCAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUU ....((((((((((((((.((.((((((......)))..))))).)))))((((((((...))).)))))..(((((((......)))))))..)).)))))))((((......)))).. ( -35.40) >DroSec_CAF1 141036 120 + 1 AAUUAUUCCUGAGACAUAUGUAUGCCUCAUCGAAGAAACGCAGCAUGUGUGGGUCCUAAACUAGUGACCCUAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUU .....((((..((((((((((.(((.((......))...)))))))))))((((((((...))).)))))..(((((((......)))))))..))..))))..((((......)))).. ( -36.20) >DroSim_CAF1 140646 120 + 1 AAUUAUUUCUAAGACACAUGUAUGCCUCAUCUAAGAAACGCAACAUGUGUGGAUCCUAAACUAGUGACCCCAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUU .............((((((((.(((.((......))...)))))))))))(((((((((((((((.......))))...)))))).....(((((..(....)..)))))...))))).. ( -33.70) >DroEre_CAF1 141293 111 + 1 AAUUCUCUCUGAGACACAUGAGA-----AGG----CAGCGCAACAUGUGUGGGUCCUAUGCUAGUGACCCCAGCUGUACGCCUAGAUGCAGCAUCUGUGGCAGUGGAUGCAAUGAUCCUU ..(((((............))))-----).(----(((((((.....)))((((((((...))).)))))..)))))..(((((((((...)))))).)))...((((......)))).. ( -33.50) >DroYak_CAF1 149650 116 + 1 AAUUAUUUCAGAGACAUAUGUGUUUCUCAAC----UGAUACAACAUGUGUGGGAUCUAUGCUAGUGACCCCAGCUGUAUGCUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUU .....(((((.((((((((((((.((.....----.)).)).)))))))((((.((.........)).))))(((((((......))))))).))).)))))..((((......)))).. ( -34.40) >consensus AAUUAUUUCUGAGACACAUGUAUGCCUCAUC_AAGAAACGCAACAUGUGUGGGUCCUAAACUAGUGACCCCAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUU .............((((((((.....................))))))))(((((((((((((((.......))))...)))))).....(((((..(....)..)))))...))))).. (-25.94 = -25.78 + -0.16)



| Location | 10,283,732 – 10,283,852 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.53 |
| Mean single sequence MFE | -31.04 |
| Consensus MFE | -20.49 |
| Energy contribution | -22.74 |
| Covariance contribution | 2.25 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.66 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.511722 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10283732 120 - 27905053 AAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUGGGGUCACUAGUUUAGGACCCACACAUGUUGCGUCUCUUAAAUGAGGCAUACUUGUGUCUCAGAAAUAAUU ..((((......))))..(((...((((((.((((.....((......))(((((.(((...))))))))....)))).))))))......((((((((....)))))))))))...... ( -32.00) >DroSec_CAF1 141036 120 - 1 AAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUAGGGUCACUAGUUUAGGACCCACACAUGCUGCGUUUCUUCGAUGAGGCAUACAUAUGUCUCAGGAAUAAUU ..((((......))))..((((..((((((.((((((........))...(((((.(((...))))))))....)))).))))))......((((((((....))))))))))))..... ( -33.40) >DroSim_CAF1 140646 120 - 1 AAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUGGGGUCACUAGUUUAGGAUCCACACAUGUUGCGUUUCUUAGAUGAGGCAUACAUGUGUCUUAGAAAUAAUU ..(((((...(((((..........)))))...............((((((.....))))))...)))))((((((((...(((((......)))))..))))))))............. ( -32.10) >DroEre_CAF1 141293 111 - 1 AAGGAUCAUUGCAUCCACUGCCACAGAUGCUGCAUCUAGGCGUACAGCUGGGGUCACUAGCAUAGGACCCACACAUGUUGCGCUG----CCU-----UCUCAUGUGUCUCAGAGAGAAUU ..((((......)))).(((....((((((.......(((((..(((((((((((.(((...)))))))).))...))))..).)----)))-----......)))))))))........ ( -30.92) >DroYak_CAF1 149650 116 - 1 AAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAGCAUACAGCUGGGGUCACUAGCAUAGAUCCCACACAUGUUGUAUCA----GUUGAGAAACACAUAUGUCUCUGAAAUAAUU ..(((((...(((((..........)))))(((......)))....(((((.....)))))...)))))...(((((((((.((.----.....)).))).))))))............. ( -26.80) >consensus AAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUGGGGUCACUAGUUUAGGACCCACACAUGUUGCGUUUCUU_GAUGAGGCAUACAUGUGUCUCAGAAAUAAUU ..((((......))))............((((.((......)).))))..(((((.(((...))))))))((((((((...(((((......)))))..))))))))............. (-20.49 = -22.74 + 2.25)


| Location | 10,283,772 – 10,283,864 |
|---|---|
| Length | 92 |
| Sequences | 5 |
| Columns | 92 |
| Reading direction | forward |
| Mean pairwise identity | 94.57 |
| Mean single sequence MFE | -28.94 |
| Consensus MFE | -28.02 |
| Energy contribution | -28.30 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.97 |
| SVM decision value | 2.49 |
| SVM RNA-class probability | 0.994522 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10283772 92 + 27905053 CAACAUGUGUGGGUCCUAAACUAGUGACCCCAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUUCACCCCCACAGC .......((.((((((((...))).)))))))(((((((......)))))))..((((((..(((((...........)))))..)))))). ( -31.50) >DroSec_CAF1 141076 92 + 1 CAGCAUGUGUGGGUCCUAAACUAGUGACCCUAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUUCACCCCCACAGC ..........((((((((...))).)))))..(((((((......)))))))..((((((..(((((...........)))))..)))))). ( -30.60) >DroSim_CAF1 140686 92 + 1 CAACAUGUGUGGAUCCUAAACUAGUGACCCCAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUUCACCCCCACAGC .......((.((.(((((...))).)).))))(((((((......)))))))..((((((..(((((...........)))))..)))))). ( -24.90) >DroEre_CAF1 141324 92 + 1 CAACAUGUGUGGGUCCUAUGCUAGUGACCCCAGCUGUACGCCUAGAUGCAGCAUCUGUGGCAGUGGAUGCAAUGAUCCUUCACCCCCACAGC .......((.((((((((...))).)))))))(((((((.....).))))))..((((((..(((((...........)))))..)))))). ( -30.10) >DroYak_CAF1 149686 92 + 1 CAACAUGUGUGGGAUCUAUGCUAGUGACCCCAGCUGUAUGCUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUUCACCCCCACAGC .........((((.((.........)).))))(((((((......)))))))..((((((..(((((...........)))))..)))))). ( -27.60) >consensus CAACAUGUGUGGGUCCUAAACUAGUGACCCCAGCUGUAUGUUUAGAUGCAGCAUCUGUGGAAGUGGAUGCAAUGAUCCUUCACCCCCACAGC .......((.((((((((...))).)))))))(((((((......)))))))..((((((..(((((...........)))))..)))))). (-28.02 = -28.30 + 0.28)


| Location | 10,283,772 – 10,283,864 |
|---|---|
| Length | 92 |
| Sequences | 5 |
| Columns | 92 |
| Reading direction | reverse |
| Mean pairwise identity | 94.57 |
| Mean single sequence MFE | -30.46 |
| Consensus MFE | -27.62 |
| Energy contribution | -27.90 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.05 |
| SVM RNA-class probability | 0.986614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10283772 92 - 27905053 GCUGUGGGGGUGAAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUGGGGUCACUAGUUUAGGACCCACACAUGUUG .((((((((((...((((......))))))))))))))..((((.((......)).))))..(((((.(((...)))))))).......... ( -30.30) >DroSec_CAF1 141076 92 - 1 GCUGUGGGGGUGAAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUAGGGUCACUAGUUUAGGACCCACACAUGCUG .((((((((((...((((......))))))))))))))..((((.((......)).))))..(((((.(((...)))))))).......... ( -30.50) >DroSim_CAF1 140686 92 - 1 GCUGUGGGGGUGAAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUGGGGUCACUAGUUUAGGAUCCACACAUGUUG .((((((((((...((((......))))))))))))))..((((.((......)).))))..(((((.(((...)))))))).......... ( -27.70) >DroEre_CAF1 141324 92 - 1 GCUGUGGGGGUGAAGGAUCAUUGCAUCCACUGCCACAGAUGCUGCAUCUAGGCGUACAGCUGGGGUCACUAGCAUAGGACCCACACAUGUUG .((((((.(((...((((......))))))).))))))(((((.......))))).(((((((((((.(((...)))))))).))...)))) ( -31.50) >DroYak_CAF1 149686 92 - 1 GCUGUGGGGGUGAAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAGCAUACAGCUGGGGUCACUAGCAUAGAUCCCACACAUGUUG .((((((((((...((((......))))))))))))))(((((.......))))).(((((((((((.........))))))).....)))) ( -32.30) >consensus GCUGUGGGGGUGAAGGAUCAUUGCAUCCACUUCCACAGAUGCUGCAUCUAAACAUACAGCUGGGGUCACUAGUUUAGGACCCACACAUGUUG .((((((((((...((((......))))))))))))))..((((.((......)).))))..(((((.(((...)))))))).......... (-27.62 = -27.90 + 0.28)

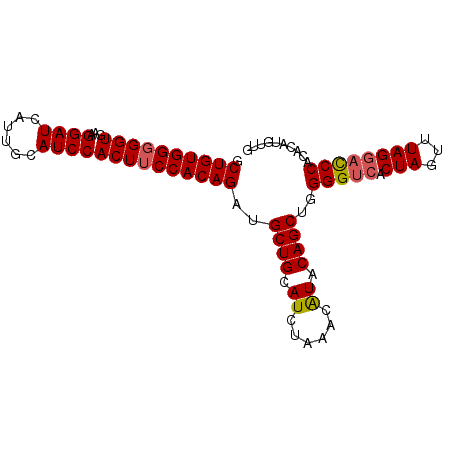
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:10:16 2006