
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 10,234,776 – 10,234,873 |
| Length | 97 |
| Max. P | 0.801462 |

| Location | 10,234,776 – 10,234,873 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 72.67 |
| Mean single sequence MFE | -46.52 |
| Consensus MFE | -22.56 |
| Energy contribution | -23.37 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.48 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.801462 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
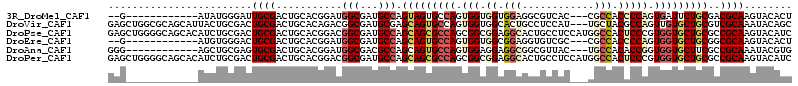
>3R_DroMel_CAF1 10234776 97 + 27905053 AGUGUACUUGCGUCGCAGAAUCACUGGGGUGGCG---GUGACGCCUCCACCACCACUGGCACUACUGGCAUCGCCAUCCGUGCAGUCGCAAUCCCAUAU------------C-- .(((...(((((.(.(((......(((((.((((---....)))).)).)))...)))((((...((((...))))...)))).).)))))...)))..------------.-- ( -36.40) >DroVir_CAF1 109979 111 + 1 GCUGUAUUUGCGACGCAGCACAACUGGCGUAGCA---AUGGAGGCAGUGCCACCACUGGCACUGCUCGCAUCGCCGUCUGUGCAGUCGCAGUCGCAGUAAUGCUGCGCCAGCUC ((((...(((((((...(((((..(((((.....---(((..((((((((((....))))))))))..))))))))..))))).))))))).(((((.....))))).)))).. ( -56.80) >DroPse_CAF1 101149 114 + 1 GAUGUACUUGCGGCGCAGCACCACGGGAGUGGCCAUGGAGGCAGUGCCUCCGCCGCUGGCGCUGCUGGCAUCGCCGUCCGUGCAGUCGCAGUCGCAGAUGUGCUGCCCCAGCUC .........((((.(((((((((((((((((((...((((((...))))))))))))((((.((....)).)))).))))))..(((((....)).)))))))))).)).)).. ( -55.30) >DroEre_CAF1 93763 97 + 1 AGUGUACUUGCGCCGCAGCACCACUGGGGUGGCG---GCGACACCUCCGCCACCACUGGCACUGCUGGCAUCGCCAUCCGUGCAGUCGCAGUCCCACAU------------C-- .(((.(((.((((.((((..(((....(((((((---(.(....).))))))))..)))..))))((((...))))...))))))))))..........------------.-- ( -40.90) >DroAna_CAF1 84360 99 + 1 CACGUAUUUGCGGCGAAGCACCACCGGUGUGGCA---GUAACGCCGCCUCCUCCACUGGCACUGCUGGCGUCGCCAUCCGUGCAGUCGCACUCGCAGCU------------CCC .......(((((((((....(((.((((..(((.---.....)))(((.........))))))).)))..)))))....((((....))))..))))..------------... ( -34.40) >DroPer_CAF1 103775 114 + 1 GAUGUACUUGCGGCGCAGCACCACGGGAGUGGCCAUGGAGGCAGUGCCUCCGCCGCUGGCGCUGCUGGCAUCGCCGUCCGUGCAGUCGCAGUCGCAGAUGUGCUGCCCCAGCUC .........((((.(((((((((((((((((((...((((((...))))))))))))((((.((....)).)))).))))))..(((((....)).)))))))))).)).)).. ( -55.30) >consensus GAUGUACUUGCGGCGCAGCACCACUGGAGUGGCA___GUGACACCGCCUCCACCACUGGCACUGCUGGCAUCGCCAUCCGUGCAGUCGCAGUCGCAGAU____________CUC ..(((..(((((((.(((..((((....)))).....(((.......))).....)))((((.(.((((...)))).).)))).)))))))..))).................. (-22.56 = -23.37 + 0.81)


| Location | 10,234,776 – 10,234,873 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 72.67 |
| Mean single sequence MFE | -48.65 |
| Consensus MFE | -24.83 |
| Energy contribution | -26.00 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.750850 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 10234776 97 - 27905053 --G------------AUAUGGGAUUGCGACUGCACGGAUGGCGAUGCCAGUAGUGCCAGUGGUGGUGGAGGCGUCAC---CGCCACCCCAGUGAUUCUGCGACGCAAGUACACU --.------------....(((((..(.(((((((...((((...))))...))).))))((((((((........)---)))))))...)..)))))...((....))..... ( -37.90) >DroVir_CAF1 109979 111 - 1 GAGCUGGCGCAGCAUUACUGCGACUGCGACUGCACAGACGGCGAUGCGAGCAGUGCCAGUGGUGGCACUGCCUCCAU---UGCUACGCCAGUUGUGCUGCGUCGCAAAUACAGC ..((.((((((((((.((((((.(((.(....).)))..(((((((.(((((((((((....)))))))).))))))---)))).)).)))).))))))))))))......... ( -57.80) >DroPse_CAF1 101149 114 - 1 GAGCUGGGGCAGCACAUCUGCGACUGCGACUGCACGGACGGCGAUGCCAGCAGCGCCAGCGGCGGAGGCACUGCCUCCAUGGCCACUCCCGUGGUGCUGCGCCGCAAGUACAUC ..((.((.(((((((...(((....)))....(((((..((((.((....)).))))((.(((((((((...))))))...))).)).)))))))))))).))))......... ( -53.90) >DroEre_CAF1 93763 97 - 1 --G------------AUGUGGGACUGCGACUGCACGGAUGGCGAUGCCAGCAGUGCCAGUGGUGGCGGAGGUGUCGC---CGCCACCCCAGUGGUGCUGCGGCGCAAGUACACU --.------------..(((..((((((.((.......((((...))))((((..(((.(((((((((........)---)))))))..).)))..))))))))).))).))). ( -46.60) >DroAna_CAF1 84360 99 - 1 GGG------------AGCUGCGAGUGCGACUGCACGGAUGGCGACGCCAGCAGUGCCAGUGGAGGAGGCGGCGUUAC---UGCCACACCGGUGGUGCUUCGCCGCAAAUACGUG .((------------.(((((..((((....))))((.((....)))).))))).)).(((......((((((.(((---..((.....))..)))...))))))...)))... ( -41.80) >DroPer_CAF1 103775 114 - 1 GAGCUGGGGCAGCACAUCUGCGACUGCGACUGCACGGACGGCGAUGCCAGCAGCGCCAGCGGCGGAGGCACUGCCUCCAUGGCCACUCCCGUGGUGCUGCGCCGCAAGUACAUC ..((.((.(((((((...(((....)))....(((((..((((.((....)).))))((.(((((((((...))))))...))).)).)))))))))))).))))......... ( -53.90) >consensus GAG____________AUCUGCGACUGCGACUGCACGGACGGCGAUGCCAGCAGUGCCAGUGGUGGAGGCGCCGCCAC___CGCCACCCCAGUGGUGCUGCGCCGCAAGUACAUC ........................((((...........(((...))).(((((((((.(((.((.(((............))).))))).)))))))))..))))........ (-24.83 = -26.00 + 1.17)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:09:39 2006