
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 1,419,726 – 1,419,837 |
| Length | 111 |
| Max. P | 0.910763 |

| Location | 1,419,726 – 1,419,837 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 82.58 |
| Mean single sequence MFE | -38.75 |
| Consensus MFE | -30.29 |
| Energy contribution | -29.47 |
| Covariance contribution | -0.82 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.880435 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
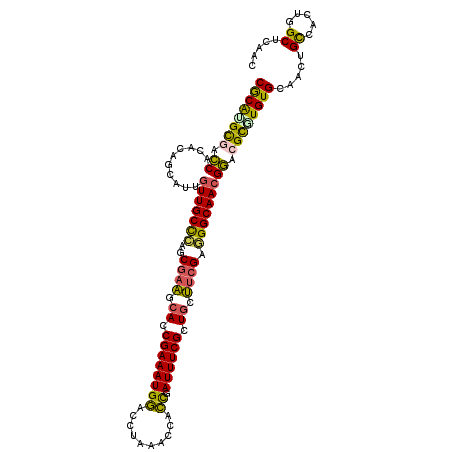
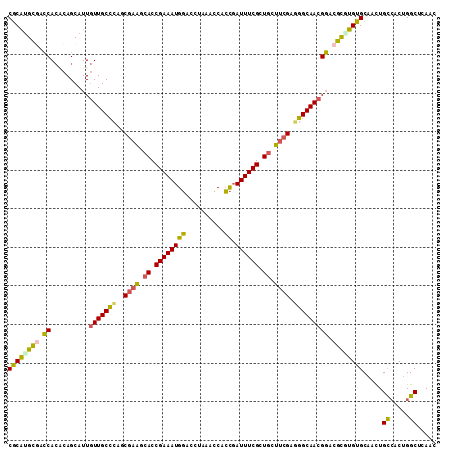
>3R_DroMel_CAF1 1419726 111 + 27905053 CGCAUGCGACCACACAGCAUUGUUGCUCAGCGAAGCACCGAAAUGGACCUAAACCCCCGAUUUCGCUUCUUCGAGGGCAACGGACGCUUGUGCAACUGCCACUGGCUCAAC .((..(((..((((.(((.(((((((((..(((((...((((((((..........)).))))))...))))).)))))))))..)))))))....))).....))..... ( -31.50) >DroGri_CAF1 1495 109 + 1 CACGGGCGCACAAUCAGCAUUAUUGCCA-GCUUGCCACCGAAAUGUGUGAAU-GCAAUCAUUUCGCUGACUCGCUGGCAAAGUGCGUCUGUGCACUUGCAACUGGCACAUU ((((((((((((((....))).((((((-((..(.((.((((((((((....-)))..))))))).)).)..)))))))).)))))))))))....(((.....))).... ( -42.30) >DroSec_CAF1 1441 111 + 1 CGCAUGCGACCACACAGCAUUGUUGCCCAGCGAAGCACCGAAAUGGACAUAAACCCCCGAUUUCGCUGCUUCGAGGGCAACGGACGCUUGUGCAACUGUCACUGGCUCAAC .((..(.(((((((.(((.(((((((((..(((((((.((((((((..........)).)))))).))))))).)))))))))..))))))).....))).)..))..... ( -42.10) >DroSim_CAF1 1438 111 + 1 CGCAUGCGACCACACAGCAUUGUUGCCCAGCGAAGCACCGAAAUGGACCUAAACCCCCGAUUUCGCUGCUUCGAGGGCAACGGACGCGUGUGCAACUGUCACUGGCUCAAC .((..(.((((((((.((.(((((((((..(((((((.((((((((..........)).)))))).))))))).)))))))))..))))))).....))).)..))..... ( -43.70) >DroEre_CAF1 1423 111 + 1 CGCAUGCUACCACUCAGCAUUGUUGCCCAGCGAAACACCGAAAUGGUCAUCAACCACCGAUUUCGCUGCUUCGAUGGCAACGGACGCGUGUGCAGCUGCCACUGGCUCAAC .(((.(((..(((...((.((((((((...((((.((.(((((((((........))).)))))).)).))))..))))))))..))..))).))))))............ ( -35.80) >DroYak_CAF1 1424 111 + 1 CGCAUGUUACCACACAGCAUUGUUGCCCAGCGAAACACCGAAAUGGACCUAAACCACCGAUUUCGCUGCUUCGUGGGCAACGGACGCAUGUGCAGCUGCCACCGGCUCAAC ............((((((.((((((((((.((((.((.((((((((..........)).)))))).)).))))))))))))))..)).)))).(((((....))))).... ( -37.10) >consensus CGCAUGCGACCACACAGCAUUGUUGCCCAGCGAAGCACCGAAAUGGACCUAAACCACCGAUUUCGCUGCUUCGAGGGCAACGGACGCGUGUGCAACUGCCACUGGCUCAAC ((((((((.((..........(((((((..((((.((.((((((((..........)).)))))).)).)))).))))))))).)))))))).....((.....))..... (-30.29 = -29.47 + -0.82)

| Location | 1,419,726 – 1,419,837 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 82.58 |
| Mean single sequence MFE | -48.75 |
| Consensus MFE | -35.01 |
| Energy contribution | -35.73 |
| Covariance contribution | 0.73 |
| Combinations/Pair | 1.26 |
| Mean z-score | -3.15 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.910763 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
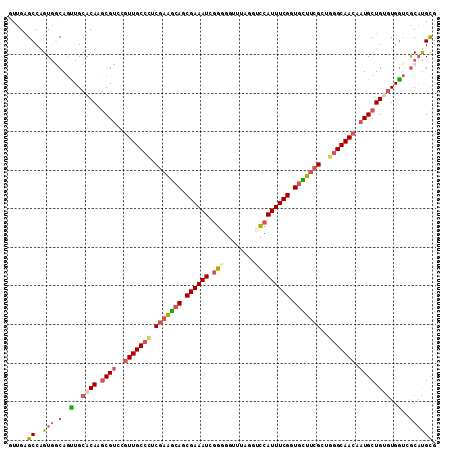
>3R_DroMel_CAF1 1419726 111 - 27905053 GUUGAGCCAGUGGCAGUUGCACAAGCGUCCGUUGCCCUCGAAGAAGCGAAAUCGGGGGUUUAGGUCCAUUUCGGUGCUUCGCUGAGCAACAAUGCUGUGUGGUCGCAUGCG .....((..(((((....(((((.((((..((((((..(((((.(.((((((.(((........))))))))).).)))))..).))))).))))))))).)))))..)). ( -39.40) >DroGri_CAF1 1495 109 - 1 AAUGUGCCAGUUGCAAGUGCACAGACGCACUUUGCCAGCGAGUCAGCGAAAUGAUUGC-AUUCACACAUUUCGGUGGCAAGC-UGGCAAUAAUGCUGAUUGUGCGCCCGUG ...(((((((((((((((((......)))))((((((((..((((.(((((((..((.-...))..))))))).))))..))-))))))...))).))))).))))..... ( -46.70) >DroSec_CAF1 1441 111 - 1 GUUGAGCCAGUGACAGUUGCACAAGCGUCCGUUGCCCUCGAAGCAGCGAAAUCGGGGGUUUAUGUCCAUUUCGGUGCUUCGCUGGGCAACAAUGCUGUGUGGUCGCAUGCG .....((..(((((....(((((.((((..(((((((.(((((((.((((((.(((.(....).))))))))).)))))))..))))))).))))))))).)))))..)). ( -53.80) >DroSim_CAF1 1438 111 - 1 GUUGAGCCAGUGACAGUUGCACACGCGUCCGUUGCCCUCGAAGCAGCGAAAUCGGGGGUUUAGGUCCAUUUCGGUGCUUCGCUGGGCAACAAUGCUGUGUGGUCGCAUGCG .....((..(((((....(((((.((((..(((((((.(((((((.((((((.(((........))))))))).)))))))..))))))).))))))))).)))))..)). ( -53.40) >DroEre_CAF1 1423 111 - 1 GUUGAGCCAGUGGCAGCUGCACACGCGUCCGUUGCCAUCGAAGCAGCGAAAUCGGUGGUUGAUGACCAUUUCGGUGUUUCGCUGGGCAACAAUGCUGAGUGGUAGCAUGCG ............((((((((.(((((((..((((((..(((((((.((((((.(((........))))))))).)))))))...)))))).))))...)))))))).))). ( -48.00) >DroYak_CAF1 1424 111 - 1 GUUGAGCCGGUGGCAGCUGCACAUGCGUCCGUUGCCCACGAAGCAGCGAAAUCGGUGGUUUAGGUCCAUUUCGGUGUUUCGCUGGGCAACAAUGCUGUGUGGUAACAUGCG (((..(((...))).((..((((.((((..(((((((((((((((.((((((.((..........)))))))).))))))).)))))))).))))))))..)))))..... ( -51.20) >consensus GUUGAGCCAGUGGCAGUUGCACAAGCGUCCGUUGCCCUCGAAGCAGCGAAAUCGGGGGUUUAGGUCCAUUUCGGUGCUUCGCUGGGCAACAAUGCUGUGUGGUCGCAUGCG .....((..(((.(..(..((((.((((..(((((((.(((((((.((((((.(((........))))))))).)))))))..))))))).))))))))..)..)))))). (-35.01 = -35.73 + 0.73)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:41:33 2006