
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 9,708,556 – 9,708,716 |
| Length | 160 |
| Max. P | 0.921405 |

| Location | 9,708,556 – 9,708,648 |
|---|---|
| Length | 92 |
| Sequences | 5 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 83.86 |
| Mean single sequence MFE | -33.52 |
| Consensus MFE | -21.54 |
| Energy contribution | -22.02 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.893337 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 9708556 92 + 27905053 CAGCCACUGGUUGGCCAUCGGCCCGACUUGGCCAUGCAACGCCAGUGGUCUCAGAUCUCAGAUCGCAGAU-------CUCCGAUCUUGGG-----UCUCCCAUU ..(((((((((..((.((.((((......))))))))...)))))))))...((((((.((((((.....-------...)))))).)))-----)))...... ( -37.80) >DroSec_CAF1 32517 97 + 1 CAGCCACUGGUUGGCCAUCGGUUCGACUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGGUCUCAGAUCGCAGAU-------CUUGGAUCCGAUCUCCCAUC ..((((((((.(((((((((...)))..)))))).......))))))))...(((((...(((((.(((((...)))-------)).))))).)))))...... ( -32.40) >DroSim_CAF1 32245 104 + 1 CAGCCACUGGUUGGCCAUCGGUUCGACUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGAUCUCAGAUCGCAGAUCUCCGAUCUUGGAUCCGAUCUCCCAUC ..((((((((.(((((((((...)))..)))))).......))))))))...(((((...(((((.((((((........)))))).))))).)))))...... ( -36.20) >DroEre_CAF1 33896 92 + 1 CAGCCACUGGUUGGCCAUCGCUUCGUCUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGGUCUCAGAU-------CUGUGAUCUUGGA-----UCUCCCAUC ..((((((((.((((((..((...))..)))))).......))))))))((.(((((.((((((...)))-------))).))))).)).-----......... ( -32.70) >DroYak_CAF1 33731 92 + 1 CAGCCACUGGUUGGCCAUCGCUUCGACUUGGCCAUGCAACGUCAGUGGUCUCAGAAAUCAGAUCUCUGAU-------CUUGGAUCUUGGA-----UCUCCCAUC ..((((((((.(((((((((...)))..)))))).......))))))))..........((((((..(((-------(...))))..)))-----)))...... ( -28.50) >consensus CAGCCACUGGUUGGCCAUCGGUUCGACUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGAUCUCAGAU_______CUCCGAUCUUGGA_____UCUCCCAUC ..((((((((.(((((((((...)))..)))))).......))))))))((.((((.....)))).))..................(((..........))).. (-21.54 = -22.02 + 0.48)

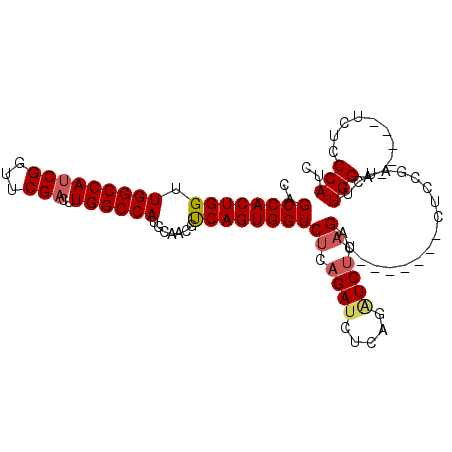

| Location | 9,708,581 – 9,708,683 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 75.82 |
| Mean single sequence MFE | -32.95 |
| Consensus MFE | -13.77 |
| Energy contribution | -15.23 |
| Covariance contribution | 1.46 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.42 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921405 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 9708581 102 + 27905053 ACUUGGCCAUGCAACGCCAGUGGUCUCAGAUCUCAGAUCGCAGAU-------CUCCGAUCUUGGG-----UCUCCCAUUCCGGGUCCAUCUCAUCUGGUCCAAAUUGGAGCGGC ...((((........))))((((.((((((((..(((((...)))-------))..)))))((((-----...))))....))).)))).....(((.(((.....))).))). ( -31.30) >DroSec_CAF1 32542 107 + 1 ACUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGGUCUCAGAUCGCAGAU-------CUUGGAUCCGAUCUCCCAUCCCGGGUCCAUCUCAUCCUGUCCAAAUUGGAGCGGC .....(((..((.......(((((((.((((.....)))).))))))).((.-------..((((((((...........))))))))..))......(((.....)))))))) ( -32.70) >DroSim_CAF1 32270 114 + 1 ACUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGAUCUCAGAUCGCAGAUCUCCGAUCUUGGAUCCGAUCUCCCAUCCCGGGUCCAUCUCAUCUGGUCCAAAUUGGAGCGGC .....(((.......(((.(((((((.(((((...))))).))))))).)))(((((((.(((((((.(((..(((.....)))........))).)))))))))))))).))) ( -39.50) >DroEre_CAF1 33921 102 + 1 UCUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGGUCUCAGAU-------CUGUGAUCUUGGA-----UCUCCCAUCCCAGAUCCAUCUCAUCUGGUCCAAAUUGGAGCGGC ....((((((.........))))))..(((((.((((((...)))-------))).)))))((((-----(((........)))))))......(((.(((.....))).))). ( -33.50) >DroYak_CAF1 33756 102 + 1 ACUUGGCCAUGCAACGUCAGUGGUCUCAGAAAUCAGAUCUCUGAU-------CUUGGAUCUUGGA-----UCUCCCAUCCCAGAUCCAUCUCAUCUGGUCCAAAUUGGAGCGGC .....(((.......(((((.(((((.(....).))))).)))))-------..(((((((.(((-----(.....)))).)))))))........(.(((.....))).)))) ( -33.60) >DroAna_CAF1 30057 80 + 1 AGC--AACAUGCAACAUG-----------------CAUCUCUGAU-------CUCCGAU-UUGGA-----U-UCAGAU-UCAGAUUCAACUCAUCUGGUCCAAAUUGGAGCUGC .((--(..((((.....)-----------------))).......-------(((((((-(((((-----(-.(((((-..((......)).)))))))))))))))))).))) ( -27.10) >consensus ACUUGGCCAUGCAACGUCAGUGGUCUCAGAUCUCAGAUCUCAGAU_______CUCCGAUCUUGGA_____UCUCCCAUCCCAGAUCCAUCUCAUCUGGUCCAAAUUGGAGCGGC ....((((((.........))))))...........................(((((((.(((((..............((((((.......)))))))))))))))))).... (-13.77 = -15.23 + 1.46)



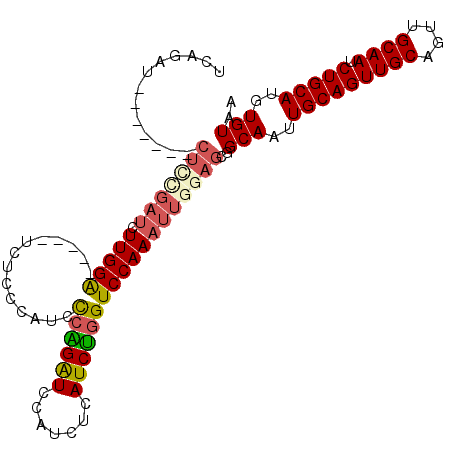
| Location | 9,708,620 – 9,708,716 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 84.50 |
| Mean single sequence MFE | -35.95 |
| Consensus MFE | -24.13 |
| Energy contribution | -24.71 |
| Covariance contribution | 0.58 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.80 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.870109 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 9708620 96 + 27905053 GCAGAU-------CUCCGAUCUUGGG-----UCUCCCAUUCCGGGUCCAUCUCAUCUGGUCCAAAUUGGAGCGGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA ((((((-------(((((((.((((.-----.(.(((.....)))............)..)))))))))))..((((((((....)))))))).))))))........ ( -37.62) >DroSec_CAF1 32581 101 + 1 UCAGAUCGCAGAU-------CUUGGAUCCGAUCUCCCAUCCCGGGUCCAUCUCAUCCUGUCCAAAUUGGAGCGGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA .((((((((.((.-------..((((((((...........))))))))..))......(((.....))))))((((((((....)))))))).)))))......... ( -34.80) >DroSim_CAF1 32309 108 + 1 UCAGAUCGCAGAUCUCCGAUCUUGGAUCCGAUCUCCCAUCCCGGGUCCAUCUCAUCUGGUCCAAAUUGGAGCGGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA .((.((.(((((((((((((.(((((((.(((..(((.....)))........))).))))))))))))))..((((((((....)))))))).)))))))).))... ( -41.70) >DroEre_CAF1 33960 96 + 1 UCAGAU-------CUGUGAUCUUGGA-----UCUCCCAUCCCAGAUCCAUCUCAUCUGGUCCAAAUUGGAGCGGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA ((((((-------.((.(((((.(((-----(.....)))).)))))))....))))))(((.....)))...(((..(((((((((....)))).)))))..))).. ( -30.50) >DroYak_CAF1 33795 96 + 1 UCUGAU-------CUUGGAUCUUGGA-----UCUCCCAUCCCAGAUCCAUCUCAUCUGGUCCAAAUUGGAGCGGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA ..(((.-------..(((((((.(((-----(.....)))).)))))))..)))..((.(((.....))).))(((..(((((((((....)))).)))))..))).. ( -33.70) >DroAna_CAF1 30077 93 + 1 UCUGAU-------CUCCGAU-UUGGA-----U-UCAGAU-UCAGAUUCAACUCAUCUGGUCCAAAUUGGAGCUGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA ......-------(((((((-(((((-----(-.(((((-..((......)).)))))))))))))))))).((((..(((((((((....)))).)))))..)))). ( -37.40) >consensus UCAGAU_______CUCCGAUCUUGGA_____UCUCCCAUCCCAGAUCCAUCUCAUCUGGUCCAAAUUGGAGCGGCAAUUGCAGUUGCAGUUGCAAUCUGCAUGUGUAA .............(((((((.(((((..............((((((.......))))))))))))))))))..(((..(((((((((....)))).)))))..))).. (-24.13 = -24.71 + 0.58)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:05:34 2006