
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 1,392,739 – 1,392,899 |
| Length | 160 |
| Max. P | 0.990116 |

| Location | 1,392,739 – 1,392,859 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.44 |
| Mean single sequence MFE | -49.48 |
| Consensus MFE | -28.52 |
| Energy contribution | -28.38 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.58 |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.943729 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
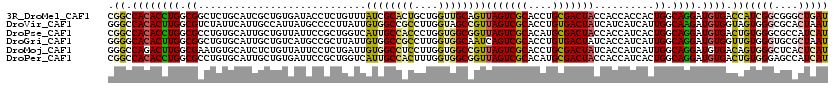
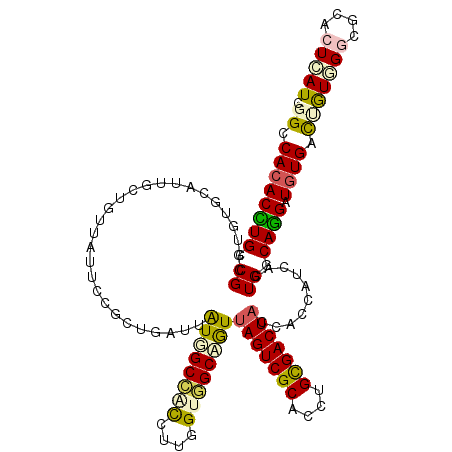
>3R_DroMel_CAF1 1392739 120 + 27905053 CGGCCACACCUGGCGGCUCUGCAUCGCUGUGAUACCUCUGUUUAUCGCACUGCUGGUUGCAGUUAGUCGCACCUGCGACUACCACCACCACUGGCAGGAUGUGACCAUCGGCGGGCUGAU (((((.(.((.(..((.((.(((((..(((((((........)))))))((((..((.(..(((((((((....)))))))..))..).))..)))))))))))))..))).)))))).. ( -48.20) >DroVir_CAF1 8664 120 + 1 GGGCCACACUUGGCGUCUAUUCAUUGCCAUUAUGCCCCUUAUUGUGGCCGCCUUGGUAGCCGUUAGUCGCACCUGUGACUAUCAUCAUCAUUGGCAAGAUGUGGUAGUGGGCGCACUAAU (.(((.(((((.((((((......(((((..(((...........(((..(....)..)))(.(((((((....))))))).)..)))...))))))))))).).))))))).)...... ( -42.00) >DroPse_CAF1 9615 120 + 1 CGGCCACACCUGGCGCCUGUGCAUUGCUGUUAUUCCGCUGGUCAUUGCCACCCUGGUGGCGGUUAGUCGCACAUGCGACUACCACCAUCACUGGCAGGAUGUGACUGUGGGCGCCAUCAU .((.....))(((((((..(((...)).((((((((((..((....(((((....)))))(((.((((((....)))))))))......))..)).))).))))).)..))))))).... ( -53.20) >DroGri_CAF1 8950 120 + 1 GGGGCACACUUGGCGGCUGUGCAUUGCUGUCAUGCCGCUUAUUGUGGCCGCCUUGGUGGCAAUCAGUCGCACCUGUGACUAUCACCAUCAUUGGCAGGAUGUGGUUGUGGGUGCGCUAAU .((.((((...((((((.(((((....)).)))))))))...)))).))(((.....)))....((.((((((((..((((((.(((....)))...)))..)))..))))))))))... ( -51.20) >DroMoj_CAF1 8142 120 + 1 GGGCCAGACUUGGCGAAUGUGCAUCUCUGUUAUUCCUCUGAUUGUGGCCUCCUUGGUGGCCGUUAGUCGCACCUGCGACUAUCACCAUCAUUGGCAGGAUGUGACAGUGGGCUCACUCAU ..(((......)))((.((.((.((.((((((((((((..((.((((((.(....).))))(.(((((((....))))))).)...)).))..).)))).))))))).)))).)).)).. ( -47.20) >DroPer_CAF1 11080 120 + 1 CGGCCACACCUGGCGCCUGUGCAUUGCUGUGAUUCCGCUGGUCAUUGCCACUUUGGUGGCGGUUAGUCGCACAUGCGACUACCACCAUCACUGGCAGGAUGUGACUGUGGGAGCCAUCAU .(((((((.(..(((....)))...).)))).((((((.(((((((((((...((((((.(((.((((((....))))))))).)))))).)))))....)))))))))))))))..... ( -55.10) >consensus CGGCCACACCUGGCGCCUGUGCAUUGCUGUUAUUCCGCUGAUUAUGGCCACCUUGGUGGCAGUUAGUCGCACCUGCGACUACCACCAUCACUGGCAGGAUGUGACUGUGGGCGCACUCAU .((.((((((((.((............................((((((((....))))))))(((((((....)))))))..........)).)))).)))).))(((((....))))) (-28.52 = -28.38 + -0.13)
| Location | 1,392,739 – 1,392,859 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.44 |
| Mean single sequence MFE | -45.00 |
| Consensus MFE | -23.03 |
| Energy contribution | -24.73 |
| Covariance contribution | 1.70 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.20 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.40 |
| SVM RNA-class probability | 0.719191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1392739 120 - 27905053 AUCAGCCCGCCGAUGGUCACAUCCUGCCAGUGGUGGUGGUAGUCGCAGGUGCGACUAACUGCAACCAGCAGUGCGAUAAACAGAGGUAUCACAGCGAUGCAGAGCCGCCAGGUGUGGCCG ....(((((((((((....)))).......(((((((..(((((((....))))))).(((((....((..((.((((........)))).))))..))))).))))))))))).))).. ( -48.00) >DroVir_CAF1 8664 120 - 1 AUUAGUGCGCCCACUACCACAUCUUGCCAAUGAUGAUGAUAGUCACAGGUGCGACUAACGGCUACCAAGGCGGCCACAAUAAGGGGCAUAAUGGCAAUGAAUAGACGCCAAGUGUGGCCC .((((((((((.((((.((((((........)))).)).))))....))))).))))).(((..(....)..))).......(((.((((.((((...........))))..)))).))) ( -40.30) >DroPse_CAF1 9615 120 - 1 AUGAUGGCGCCCACAGUCACAUCCUGCCAGUGAUGGUGGUAGUCGCAUGUGCGACUAACCGCCACCAGGGUGGCAAUGACCAGCGGAAUAACAGCAAUGCACAGGCGCCAGGUGUGGCCG ....(((((((....((((....(..((..((.(((((((((((((....)))))).))))))).)).))..)...))))..((.........))........)))))))((.....)). ( -50.20) >DroGri_CAF1 8950 120 - 1 AUUAGCGCACCCACAACCACAUCCUGCCAAUGAUGGUGAUAGUCACAGGUGCGACUGAUUGCCACCAAGGCGGCCACAAUAAGCGGCAUGACAGCAAUGCACAGCCGCCAAGUGUGCCCC (((((((((((........((((........))))(((.....))).)))))).))))).(((.....)))(((.(((....(((((.((.((....))))..)))))....)))))).. ( -42.80) >DroMoj_CAF1 8142 120 - 1 AUGAGUGAGCCCACUGUCACAUCCUGCCAAUGAUGGUGAUAGUCGCAGGUGCGACUAACGGCCACCAAGGAGGCCACAAUCAGAGGAAUAACAGAGAUGCACAUUCGCCAAGUCUGGCCC ..(((((.((.(.((((....((((((((....))))(((((((((....)))))))..((((.(....).))))....))..))))...)))).)..)).)))))((((....)))).. ( -41.90) >DroPer_CAF1 11080 120 - 1 AUGAUGGCUCCCACAGUCACAUCCUGCCAGUGAUGGUGGUAGUCGCAUGUGCGACUAACCGCCACCAAAGUGGCAAUGACCAGCGGAAUCACAGCAAUGCACAGGCGCCAGGUGUGGCCG .((.(((...)))))((((((((.((((.(((.(((((((((((((....)))))).)))(((((....)))))....))))((.........))....))).))))...)))))))).. ( -46.80) >consensus AUGAGCGCGCCCACAGUCACAUCCUGCCAAUGAUGGUGAUAGUCGCAGGUGCGACUAACCGCCACCAAGGCGGCCACAAUCAGCGGAAUAACAGCAAUGCACAGCCGCCAAGUGUGGCCC ...............((((((.((((((..((.(((((((((((((....))))))).)))))).))....)))..........((.....................))))))))))).. (-23.03 = -24.73 + 1.70)


| Location | 1,392,779 – 1,392,899 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.06 |
| Mean single sequence MFE | -47.62 |
| Consensus MFE | -26.02 |
| Energy contribution | -26.28 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.48 |
| Mean z-score | -3.61 |
| Structure conservation index | 0.55 |
| SVM decision value | 2.20 |
| SVM RNA-class probability | 0.990116 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1392779 120 + 27905053 UUUAUCGCACUGCUGGUUGCAGUUAGUCGCACCUGCGACUACCACCACCACUGGCAGGAUGUGACCAUCGGCGGGCUGAUUGGCCUGUUUGCAGGCUAUAUAAGCUACACACAAUACUAU .....(((.((((..((.(..(((((((((....)))))))..))..).))..))))((((....)))).)))((((...(((((((....)))))))....)))).............. ( -45.60) >DroVir_CAF1 8704 120 + 1 AUUGUGGCCGCCUUGGUAGCCGUUAGUCGCACCUGUGACUAUCAUCAUCAUUGGCAAGAUGUGGUAGUGGGCGCACUAAUCGGCCUGGCCACCGGAUACAUUAGCUACCGACAAUAUUAU ...((((((((....)).(((((((((.((.(((...(((((((.((((........)))))))))))))).))))))).))))..))))))(((............))).......... ( -42.80) >DroPse_CAF1 9655 120 + 1 GUCAUUGCCACCCUGGUGGCGGUUAGUCGCACAUGCGACUACCACCAUCACUGGCAGGAUGUGACUGUGGGCGCCAUCAUUGGCCUGCUCACGGGCUAUAUCAGCUACAGACAAUAUUAU (((.((((((...((((((.(((.((((((....))))))))).)))))).))))))((((((.((((((((((((....))))..))))))))..)))))).......)))........ ( -57.60) >DroGri_CAF1 8990 120 + 1 AUUGUGGCCGCCUUGGUGGCAAUCAGUCGCACCUGUGACUAUCACCAUCAUUGGCAGGAUGUGGUUGUGGGUGCGCUAAUUGGCCUCGUCAGCGGAUACUUUAGCUACAGGCAAUAUUAU ((((..(((((....)))))...))))((((((((..((((((.(((....)))...)))..)))..)))))))).((((..((((.((.(((..........)))))))))...)))). ( -42.90) >DroMoj_CAF1 8182 120 + 1 AUUGUGGCCUCCUUGGUGGCCGUUAGUCGCACCUGCGACUAUCACCAUCAUUGGCAGGAUGUGACAGUGGGCUCACUCAUUGGUCUAUUCAGUGGAUACAUCAGUUAUAGACAAUAUUAU (((((.(((....((((((..(.(((((((....))))))).).))))))..)))..(((((..(((((((....)))))))((((((...)))))))))))........)))))..... ( -41.40) >DroPer_CAF1 11120 120 + 1 GUCAUUGCCACUUUGGUGGCGGUUAGUCGCACAUGCGACUACCACCAUCACUGGCAGGAUGUGACUGUGGGAGCCAUCAUUGGCCUGCUCACGGGCUAUAUCAGCUACAGACAAUAUUAU (((.((((((...((((((.(((.((((((....))))))))).)))))).))))))((((((.(((((((.((((....))))...)))))))..)))))).......)))........ ( -55.40) >consensus AUUAUGGCCACCUUGGUGGCAGUUAGUCGCACCUGCGACUACCACCAUCACUGGCAGGAUGUGACUGUGGGCGCACUCAUUGGCCUGCUCACCGGAUACAUCAGCUACAGACAAUAUUAU ......((((...((((((....(((((((....)))))))))))))....)))).....((((..((((((..........))))))))))............................ (-26.02 = -26.28 + 0.26)
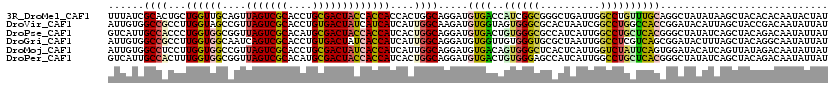

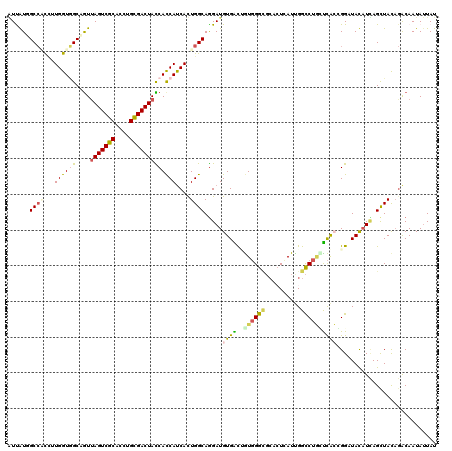
| Location | 1,392,779 – 1,392,899 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.06 |
| Mean single sequence MFE | -41.78 |
| Consensus MFE | -19.32 |
| Energy contribution | -20.52 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.782874 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1392779 120 - 27905053 AUAGUAUUGUGUGUAGCUUAUAUAGCCUGCAAACAGGCCAAUCAGCCCGCCGAUGGUCACAUCCUGCCAGUGGUGGUGGUAGUCGCAGGUGCGACUAACUGCAACCAGCAGUGCGAUAAA ...(((((((.(((((........(((((....))))).......(((((((.((((........)))).)))))).).(((((((....))))))).)))))....)))))))...... ( -44.50) >DroVir_CAF1 8704 120 - 1 AUAAUAUUGUCGGUAGCUAAUGUAUCCGGUGGCCAGGCCGAUUAGUGCGCCCACUACCACAUCUUGCCAAUGAUGAUGAUAGUCACAGGUGCGACUAACGGCUACCAAGGCGGCCACAAU ..........(((..((....))..)))((((((.(((((.((((((((((.((((.((((((........)))).)).))))....))))).)))))))))).(....).))))))... ( -44.00) >DroPse_CAF1 9655 120 - 1 AUAAUAUUGUCUGUAGCUGAUAUAGCCCGUGAGCAGGCCAAUGAUGGCGCCCACAGUCACAUCCUGCCAGUGAUGGUGGUAGUCGCAUGUGCGACUAACCGCCACCAGGGUGGCAAUGAC ....(((((((((((.....))))((((.((.(((((...(((.((((.......)))))))))))))).((.(((((((((((((....)))))).))))))).))))))))))))).. ( -48.60) >DroGri_CAF1 8990 120 - 1 AUAAUAUUGCCUGUAGCUAAAGUAUCCGCUGACGAGGCCAAUUAGCGCACCCACAACCACAUCCUGCCAAUGAUGGUGAUAGUCACAGGUGCGACUGAUUGCCACCAAGGCGGCCACAAU .....................((..(((((...(.(((.((((((((((((........((((........))))(((.....))).)))))).))))))))).)...)))))..))... ( -37.30) >DroMoj_CAF1 8182 120 - 1 AUAAUAUUGUCUAUAACUGAUGUAUCCACUGAAUAGACCAAUGAGUGAGCCCACUGUCACAUCCUGCCAAUGAUGGUGAUAGUCGCAGGUGCGACUAACGGCCACCAAGGAGGCCACAAU ....((((((((((...((.......))....))))).)))))((((....))))((((((((........))).)))))((((((....))))))...((((.(....).))))..... ( -31.00) >DroPer_CAF1 11120 120 - 1 AUAAUAUUGUCUGUAGCUGAUAUAGCCCGUGAGCAGGCCAAUGAUGGCUCCCACAGUCACAUCCUGCCAGUGAUGGUGGUAGUCGCAUGUGCGACUAACCGCCACCAAAGUGGCAAUGAC .......((.((((.(((.((.......)).))).(((((....)))))...)))).))(((..(((((.((.(((((((((((((....)))))).))))))).))...)))))))).. ( -45.30) >consensus AUAAUAUUGUCUGUAGCUGAUAUAGCCCGUGAACAGGCCAAUGAGCGCGCCCACAGUCACAUCCUGCCAAUGAUGGUGAUAGUCGCAGGUGCGACUAACCGCCACCAAGGCGGCCACAAU ........((((((..................))))))...........................(((..((.(((((((((((((....))))))).)))))).))....)))...... (-19.32 = -20.52 + 1.20)

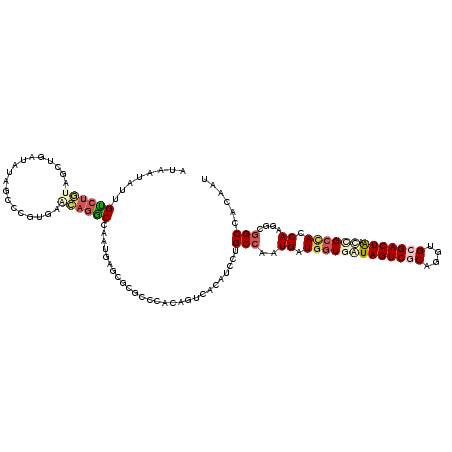
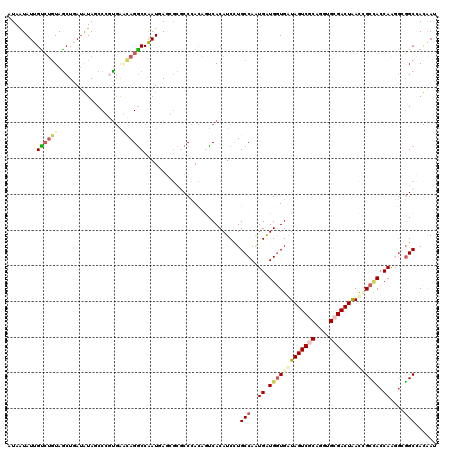
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:41:12 2006