
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 9,598,042 – 9,598,143 |
| Length | 101 |
| Max. P | 0.772234 |

| Location | 9,598,042 – 9,598,143 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.55 |
| Mean single sequence MFE | -29.21 |
| Consensus MFE | -22.34 |
| Energy contribution | -23.57 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.772234 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 9598042 101 + 27905053 CUCGAGGAUAAUUUGUUGUGUUGAUUAUUAUGGGGCGGCUAACCAUUUGGUUCCUACUGGGACUAACGUUAAUCGCCGUUUCCAAUAAUGGUAAGUA-CA-----C-------------U .................((((..(((((((((..(((((((((...((((((((....)))))))).))))...)))))..)..))))))))....)-))-----)-------------. ( -30.20) >DroVir_CAF1 4580 102 + 1 CUCAAGGAUAAUUUGUUGAUUUUAUGAUUAUGGGGCGGCUAACAAUUUGGUUCCUGCUGGGACUUACGUUAAUCGCCGUUUCCAACAAUGGUGAGUUUCC-----A-------------G .....(((.((((..((((((....)))).(((((((..((((.....((((((....))))))...))))..))))....))).....))..)))))))-----.-------------. ( -25.90) >DroGri_CAF1 5190 101 + 1 CUCAAGGAUAAUUUGUGGAUUUGAUUAUUAUGGGGCGACUAACAAUUUGGUUCCUGCUGGGACUGACGUUAAUCGCCGUAUCCAAUAAUGGUGAGUG-UG-----U-------------C ((((..(((((((.........)))))))(((.(((((.((((...(..(((((....)))))..).)))).))))).)))(((....)))))))..-..-----.-------------. ( -27.40) >DroWil_CAF1 5405 106 + 1 CUCAAGGAUAAUUUGUUGAUUUGAUCAUAAUGGGGCGGCUAACUAUAUGGUUCCUAUUGGGACUAACGUUAAUCGCCGUCUCCAACAAUGGUGAGUU-UAAAAUCC-------------A .....((((......((((.((.(((((..(((((((((((((....(((((((....)))))))..))))...)))))).)))...))))).)).)-))).))))-------------. ( -31.40) >DroMoj_CAF1 4517 101 + 1 CUCAAGGAUAAUUUGUUGAUUUGAUUAUUAUGGGGCGUUUAACACUUUGGUUCCUGCUGGGACUGACGUUAAUCGCCGUUUCCUAUAAUGGUGAGUG-CG-----A-------------G .((((.((....)).)))).((.((((((((((((((.(((((...(..(((((....)))))..).))))).))).....))))))))))).))..-..-----.-------------. ( -28.70) >DroAna_CAF1 5385 114 + 1 CUCGAGGAUAAUUUGUUGUUUUGAUUAUUAUGGGGCGGCUAACAAUUUGGUUCCUUCUGGGACUAACGUUAUUCGCCGUCUCCAAUCAUGGUGAGUA-CU-----UCCUAACUUGAUCCU ....(((((.....((((......(((((((((((((((((((...((((((((....)))))))).))))...))))))).....))))))))...-..-----...))))...))))) ( -31.64) >consensus CUCAAGGAUAAUUUGUUGAUUUGAUUAUUAUGGGGCGGCUAACAAUUUGGUUCCUGCUGGGACUAACGUUAAUCGCCGUUUCCAAUAAUGGUGAGUA_CA_____A_____________G .((((.((....)).)))).((.((((((((((((((..((((...((((((((....)))))))).))))..))))....)).)))))))).))......................... (-22.34 = -23.57 + 1.22)
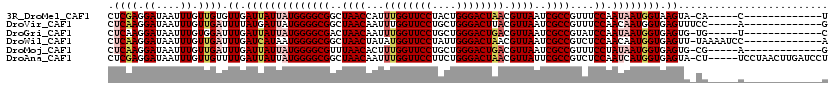

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:03:57 2006