
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 9,451,177 – 9,451,286 |
| Length | 109 |
| Max. P | 0.900430 |

| Location | 9,451,177 – 9,451,286 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.90 |
| Mean single sequence MFE | -34.87 |
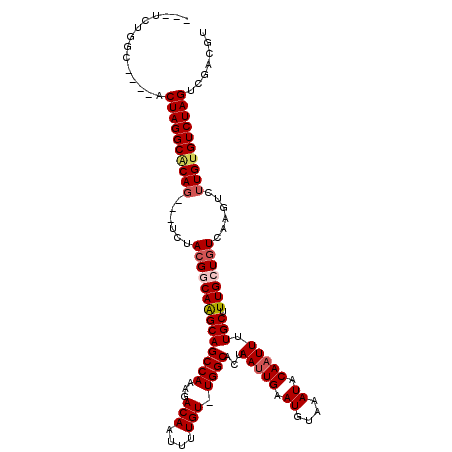
| Consensus MFE | -28.53 |
| Energy contribution | -29.28 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.900430 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
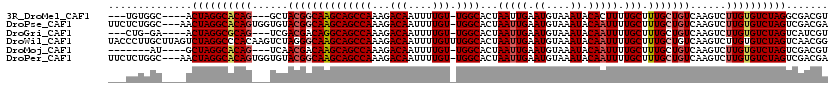
>3R_DroMel_CAF1 9451177 109 - 27905053 ---UGUGGC----ACUAGGCACAG---GCUACGGCAAGCAGCCAAAGACAAUUUUGU-UGGCACUAAUUGAAUGUAAAUACACUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGGCGACGU ---.((.((----.((((((((((---((((((((((((((((((.((.....)).)-))))..........(((....)))....))).)))))))..)).))))))))))))).)).. ( -41.40) >DroPse_CAF1 81865 116 - 1 UUCUCUGGC---AACUAGGCACAGUGGUGUACGGCAAGCAGCCAAAGACAAUUUUGU-UGGCACUAAUUGAAUGUAAAUACAAUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGUCGACGA .....((.(---.(((((((((((...((.(((((((((((((((.((.....)).)-))))...(((((.((....)).))))).))).)))))))))....))))))))))).).)). ( -34.90) >DroGri_CAF1 120722 108 - 1 ---CUG-GA----ACUAGGCGCAG---UCGACGACAGGCAGCCAAAGACAAUUUUGU-UGGCACUAAUUGAAUGUAAAUACAAUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGUCAUCGU ---...-((----.((((((((((---..(((....((((((.((((.(((..((((-..(((.........)))....))))..)))))))))))))..)))))))))))))))..... ( -34.70) >DroWil_CAF1 88975 120 - 1 UACCCUUGCUUAGUCUAGGCCCACAAGUCUAGGGCAAGCAGCCAAAGACAAUUUUGUUUGGCACUAAUUGAAUGUAAAUACAAUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGUCAACGG ............(.((((((..(((((.((..((((.(((((((((.(......).))))))...........(((((......)))))..))))))).)).))))))))))).)..... ( -32.20) >DroMoj_CAF1 135417 105 - 1 -------AU----GCUAGGCACAG---UCAACGACAAGCAGCCAAAGACAAUUUUGU-UGGCACUAAUUGAAUGUAAAUACAAUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGUCGACGU -------..----(((((((((((---.....(((..(((((.((((.(((..((((-..(((.........)))....))))..))))))))))))...))))))))))))))...... ( -31.10) >DroPer_CAF1 83532 116 - 1 UUCUCUGGC---AACUAGGCACAGUGGUGUACGGCAAGCAGCCAAAGACAAUUUUGU-UGGCACUAAUUGAAUGUAAAUACAAUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGUCGACGA .....((.(---.(((((((((((...((.(((((((((((((((.((.....)).)-))))...(((((.((....)).))))).))).)))))))))....))))))))))).).)). ( -34.90) >consensus ___UCUGGC____ACUAGGCACAG___UCUACGGCAAGCAGCCAAAGACAAUUUUGU_UGGCACUAAUUGAAUGUAAAUACAAUUUUGCUUUGCUGUCAAGUCUUGUGUCUAGUCGACGU ..............((((((((((......((((((((((((((...(((....))).))))...(((((.((....)).))))).))).)))))))......))))))))))....... (-28.53 = -29.28 + 0.75)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:02:38 2006