| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 9,356,575 – 9,356,685 |
| Length | 110 |
| Max. P | 0.906482 |

| Location | 9,356,575 – 9,356,685 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 78.20 |
| Mean single sequence MFE | -46.52 |
| Consensus MFE | -30.15 |
| Energy contribution | -32.02 |
| Covariance contribution | 1.87 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.906482 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
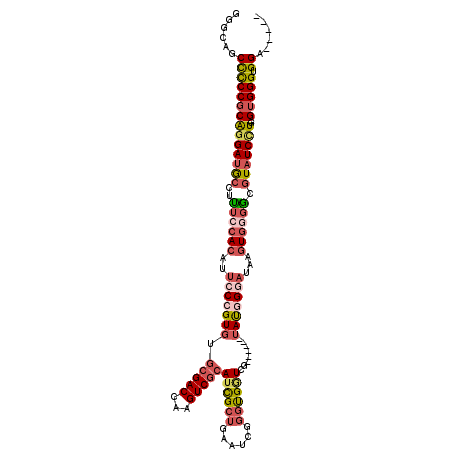
>3R_DroMel_CAF1 9356575 110 - 27905053 GGGCAGCCCCCGCAGGAUGCUUUUCCACAUUCCCGUGUGCGACCAAGUCUCAUCGCUGAAUCGGGUGGUCG------UAUGGGAUAAGUUGGGUGUAUCCUG-GUGGGUGGAUUGGG ...(((((((((((((((((...(((((..((((..(((((((((.(..(((....)))..)...))))))------)))))))...).)))).))))))).-))))).)).))).. ( -47.90) >DroSec_CAF1 126264 110 - 1 GGGCAGCCCCCGCAGGAUGCCUUUCCACAUUCCCGUGUGCGACCAAGUCGCAUCGCUGAAUCGGGUGAUCG------UAUGGGAUAAGUGGGACGUAUCCUG-GUGGGUGGCUUGGG ....((((((((((((((((..((((((..(((((((((((((...))))))(((((......)))))...------)))))))...)))))).))))))).-))))).)))).... ( -53.80) >DroSim_CAF1 135121 110 - 1 GGGCAGCCCCCGCAGGAUGCCUUUCCACAUUCCCGUGUGCGACCAAGUCGCAUCGCUGAAUCGGGUGGUUG------UAUGGGAUAAGUGGGGCGUAUCCUG-GUGGGUGGAUUGGG ...(((((((((((((((((..((((((..(((((((.(((((...)))))((((((......))))))..------)))))))...)))))).))))))).-))))).)).))).. ( -49.80) >DroEre_CAF1 126525 105 - 1 GGGCAGCCCCCGCAGGAUGCUUUUCCACAUUACCGUGUGCGACCAAGUCGCAUCGCUGAAUCGGGCGGUCG------UAUGGGAUAAGUGGGGCGUAUCUUG-GUGGGUGGA----- ......((((((((((((((..((((((.(((((.(((((((((..(......)(((......))))))))------)))))).))))))))).))))))).-))))).)).----- ( -42.40) >DroYak_CAF1 126722 105 - 1 GGGCAGCCCCCGCAGGAUGCUCUUGCACAUUACAGUGUGCGACCAAGUCGCAUCGCUGAAUCGGUUGGUCG------UACGGGAUAAGUGGGGCGUAUCCUA-GUGGGUGGA----- ......(((((((((((((((((..(..(((....(((((((((((.(((.((......))))))))))))------)))).)))..)..))).))))))).-))))).)).----- ( -44.30) >DroAna_CAF1 121558 110 - 1 AAUCACCUUCCGCG-GAAACCUCACCAC-UGCCUGUGCUCGACCAUGUCGCAUUGCACGGAAGGGUGCUCUUUGGCGGAUUGGAACGCUGUGGGAUGUCCUGUGAGGGCGGA----- ........((((((-....)(((((...-...((((((.((((...))))....)))))).(((..(..((.(((((........))))).))..)..)))))))).)))))----- ( -40.90) >consensus GGGCAGCCCCCGCAGGAUGCCUUUCCACAUUCCCGUGUGCGACCAAGUCGCAUCGCUGAAUCGGGUGGUCG______UAUGGGAUAAGUGGGGCGUAUCCUG_GUGGGUGGA_____ ......((((((((((((((..((((((..(((((((.(((((...)))))((((((......))))))........)))))))...)))))).)))))))..))))).))...... (-30.15 = -32.02 + 1.87)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:01:29 2006