
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 8,718,300 – 8,718,450 |
| Length | 150 |
| Max. P | 0.999100 |

| Location | 8,718,300 – 8,718,410 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.77 |
| Mean single sequence MFE | -29.62 |
| Consensus MFE | -23.11 |
| Energy contribution | -23.92 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.06 |
| SVM RNA-class probability | 0.907909 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
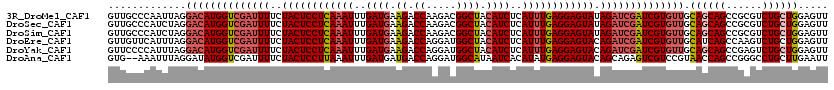
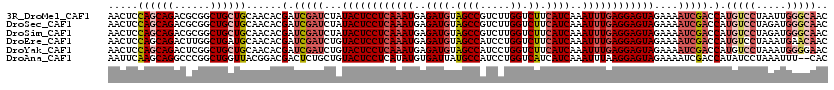
>3R_DroMel_CAF1 8718300 110 + 27905053 AUUUACCAACUU-C--AAGCCCUCAU----UCAA-UCU--GUUGCCCAAUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGU (((.((((....-.--..((...((.----....-..)--)..))((.....))...)))).))).....(((((((((((..((((.((.((.....)))).))))..))))))))))) ( -27.20) >DroSec_CAF1 144865 110 + 1 AUUAACCAACUU-C--AAGCCCUCAU----UCAA-UCU--GUUGCCCAUCUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGU ((..((((....-.--..((...((.----....-..)--)..))((.....))...))))..)).....(((((((((((..((((.((.((.....)))).))))..))))))))))) ( -28.10) >DroSim_CAF1 161797 110 + 1 AUUAACCGACUU-C--AAGCCCUCAU----UCAA-UCU--GUUGCCCAUCUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGU ......(((((.-.--...((.....----.(((-...--.)))........))....))))).......(((((((((((..((((.((.((.....)))).))))..))))))))))) ( -29.54) >DroEre_CAF1 161126 111 + 1 ACUGACCAACUU-C--AAGUGCACAU----UAAACUUU--GUUGUUCAUUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAGGAUGGCUACAUCUCAUUUGAGGAGU ..((((((..((-(--(((((.(((.----........--..))).))))).)))..)))))).......(((((((((((..((((.((.((.....)))).))))..))))))))))) ( -36.20) >DroYak_CAF1 153233 113 + 1 AUUGAACAACUU-C--AAGUCCACAU----UCAAGUUUAUGUUCCCCAUUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAGGAUGGCUACAUCUCAUUUGAGGAGU .(((((....))-)--))........----....(.((((((((........)))))))).)........(((((((((((..((((.((.((.....)))).))))..))))))))))) ( -31.10) >DroAna_CAF1 137416 116 + 1 UUUUAGCAAAUUUCUCACUUCUAUAUACACAAAAGCAU--GUG--AAAUUUAGGAUAUGGUCGAUUUUCUACUCCUUAAAUUUGAUGAUGACCAGGAUGGCAUAAUCACAUAUGAGGAGU ...(((.(((((..(((.(((((....((((......)--)))--.....)))))..)))..))))).)))(((((((.((.((((.(((.((.....))))).)))).)).))))))). ( -25.60) >consensus AUUAACCAACUU_C__AAGCCCACAU____UCAA_UCU__GUUGCCCAUUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGU ........................................((((.((((.......)))).)))).....(((((((((((..((((.((.((.....)))).))))..))))))))))) (-23.11 = -23.92 + 0.81)


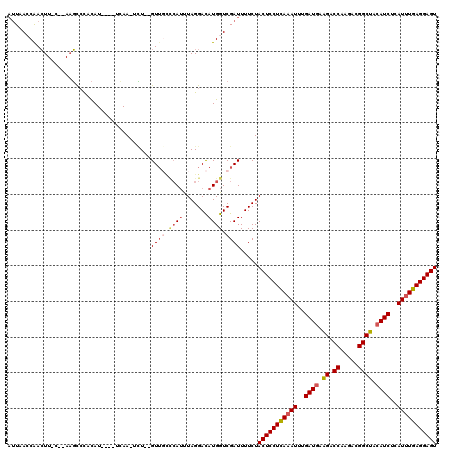
| Location | 8,718,300 – 8,718,410 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.77 |
| Mean single sequence MFE | -34.17 |
| Consensus MFE | -26.97 |
| Energy contribution | -28.50 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.40 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941965 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8718300 110 - 27905053 ACUCCUCAAAUGAGAUGUAGCCGUCUUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAUUGGGCAAC--AGA-UUGA----AUGAGGGCUU--G-AAGUUGGUAAAU (((((((((((..((((.((((.....)).)).))))..))))))))))).....((((((((.(((((((((.(....)--.))-))..----...))))).)--)-..)))))).... ( -36.10) >DroSec_CAF1 144865 110 - 1 ACUCCUCAAAUGAGAUGUAGCCGUCUUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAGAUGGGCAAC--AGA-UUGA----AUGAGGGCUU--G-AAGUUGGUUAAU (((((((((((..((((.((((.....)).)).))))..)))))))))))....(((((((((.(((((.(((.(....)--..)-))..----...))))).)--)-..)))))))... ( -37.40) >DroSim_CAF1 161797 110 - 1 ACUCCUCAAAUGAGAUGUAGCCGUCUUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAGAUGGGCAAC--AGA-UUGA----AUGAGGGCUU--G-AAGUCGGUUAAU (((((((((((..((((.((((.....)).)).))))..)))))))))))....(((((((((.(((((.(((.(....)--..)-))..----...))))).)--)-..)))))))... ( -39.50) >DroEre_CAF1 161126 111 - 1 ACUCCUCAAAUGAGAUGUAGCCAUCCUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAAUGAACAAC--AAAGUUUA----AUGUGCACUU--G-AAGUUGGUCAGU (((((((((((..((((.((((.....)).)).))))..)))))))))))........(((((((..(....(((((...--...)))))----..)..))...--.-....)))))... ( -33.20) >DroYak_CAF1 153233 113 - 1 ACUCCUCAAAUGAGAUGUAGCCAUCCUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAAUGGGGAACAUAAACUUGA----AUGUGGACUU--G-AAGUUGUUCAAU (((((((((((..((((.((((.....)).)).))))..)))))))))))..........((((.......)))).(((((...((((.(----(.(....)))--.-)))))))))... ( -34.40) >DroAna_CAF1 137416 116 - 1 ACUCCUCAUAUGUGAUUAUGCCAUCCUGGUCAUCAUCAAAUUUAAGGAGUAGAAAAUCGACCAUAUCCUAAAUUU--CAC--AUGCUUUUGUGUAUAUAGAAGUGAGAAAUUUGCUAAAA ((((((......((((.(((((.....)).))).))))......))))))..................(((((((--(.(--..((((((((....))))))))).))))))))...... ( -24.40) >consensus ACUCCUCAAAUGAGAUGUAGCCAUCCUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAAUGGGCAAC__AGA_UUGA____AUGAGGACUU__G_AAGUUGGUAAAU (((((((((((..((((.((((.....)).)).))))..)))))))))))........((((.(((((.....))))).....................(((((....)))))))))... (-26.97 = -28.50 + 1.53)


| Location | 8,718,330 – 8,718,450 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.50 |
| Mean single sequence MFE | -49.48 |
| Consensus MFE | -44.75 |
| Energy contribution | -45.75 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.10 |
| Mean z-score | -5.18 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.37 |
| SVM RNA-class probability | 0.999100 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8718330 120 + 27905053 GUUGCCCAAUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGUAUAGAUCGAUCGUGUUGCAGCAGCCGCGUCUGCUGGAGUU .............((((((((((((((..((((((((((((..((((.((.((.....)))).))))..)))))))))))).)))))))))))))).((((((......))))))..... ( -52.30) >DroSec_CAF1 144895 120 + 1 GUUGCCCAUCUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGUAUAGAUCGAUCGUGUUGCAGCAGCCGCGUCUGCUGGAGUU .............((((((((((((((..((((((((((((..((((.((.((.....)))).))))..)))))))))))).)))))))))))))).((((((......))))))..... ( -52.30) >DroSim_CAF1 161827 120 + 1 GUUGCCCAUCUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGUAUAGAUCGAUCGUGUUGCAGCAGCCGCGUCUGCUGGAGUU .............((((((((((((((..((((((((((((..((((.((.((.....)))).))))..)))))))))))).)))))))))))))).((((((......))))))..... ( -52.30) >DroEre_CAF1 161157 120 + 1 GUUGUUCAUUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAGGAUGGCUACAUCUCAUUUGAGGAGUACAGAUCGAUCGUGUUGCAUCAGCCAAGUCUGCUGGAGUU .............((((((((((((((..((((((((((((..((((.((.((.....)))).))))..)))))))))))).))))))))))))))((..((((.......))))..)). ( -49.30) >DroYak_CAF1 153266 120 + 1 GUUCCCCAUUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAGGAUGGCUACAUCUCAUUUGAGGAGUACAGAUCGAUCGUGUUGCAGCAGCCGAGUCUGCUGGAGUU .............((((((((((((((..((((((((((((..((((.((.((.....)))).))))..)))))))))))).)))))))))))))).((((((......))))))..... ( -52.40) >DroAna_CAF1 137454 118 + 1 GUG--AAAUUUAGGAUAUGGUCGAUUUUCUACUCCUUAAAUUUGAUGAUGACCAGGAUGGCAUAAUCACAUAUGAGGAGUACAGCAGAGUCGUCCGUAACCAGCCGGGCCUGCUUGAAUU ...--.......((.(((((.(((((((((((((((((.((.((((.(((.((.....))))).)))).)).))))))))..)).))))))).))))).))...((((....)))).... ( -38.30) >consensus GUUGCCCAUUUAGGACAUGGUCGAUUUUCUACUCCUCAAAUUUGAUGAAGACCAAGACGGCUACAUCUCAUUUGAGGAGUACAGAUCGAUCGUGUUGCAGCAGCCGCGUCUGCUGGAGUU .............((((((((((((((..((((((((((((..((((.((.((.....)))).))))..)))))))))))).)))))))))))))).((((((......))))))..... (-44.75 = -45.75 + 1.00)
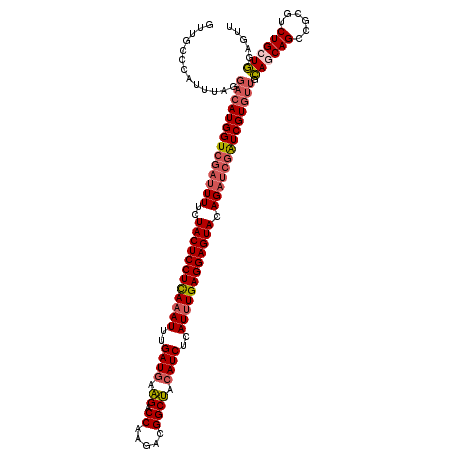
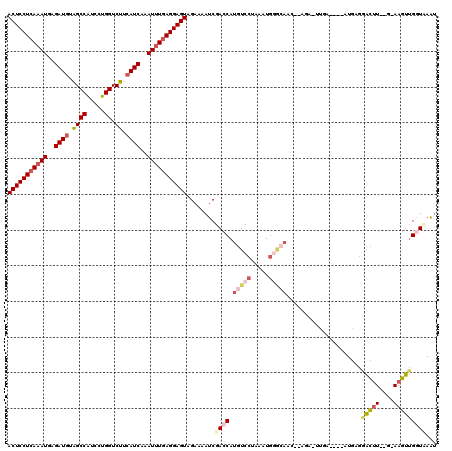
| Location | 8,718,330 – 8,718,450 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.50 |
| Mean single sequence MFE | -41.63 |
| Consensus MFE | -34.90 |
| Energy contribution | -36.77 |
| Covariance contribution | 1.86 |
| Combinations/Pair | 1.11 |
| Mean z-score | -4.19 |
| Structure conservation index | 0.84 |
| SVM decision value | 2.54 |
| SVM RNA-class probability | 0.995113 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8718330 120 - 27905053 AACUCCAGCAGACGCGGCUGCUGCAACACGAUCGAUCUAUACUCCUCAAAUGAGAUGUAGCCGUCUUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAUUGGGCAAC .....((((((......))))))......(.(((((...((((((((((((..((((.((((.....)).)).))))..))))))))))))....))))).).(((((.....))))).. ( -45.80) >DroSec_CAF1 144895 120 - 1 AACUCCAGCAGACGCGGCUGCUGCAACACGAUCGAUCUAUACUCCUCAAAUGAGAUGUAGCCGUCUUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAGAUGGGCAAC .....((((((......))))))......(.(((((...((((((((((((..((((.((((.....)).)).))))..))))))))))))....))))).).(((((.....))))).. ( -45.80) >DroSim_CAF1 161827 120 - 1 AACUCCAGCAGACGCGGCUGCUGCAACACGAUCGAUCUAUACUCCUCAAAUGAGAUGUAGCCGUCUUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAGAUGGGCAAC .....((((((......))))))......(.(((((...((((((((((((..((((.((((.....)).)).))))..))))))))))))....))))).).(((((.....))))).. ( -45.80) >DroEre_CAF1 161157 120 - 1 AACUCCAGCAGACUUGGCUGAUGCAACACGAUCGAUCUGUACUCCUCAAAUGAGAUGUAGCCAUCCUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAAUGAACAAC .....((((.......)))).....(((.(.(((((.(.((((((((((((..((((.((((.....)).)).))))..)))))))))))).)..))))).).))).............. ( -36.90) >DroYak_CAF1 153266 120 - 1 AACUCCAGCAGACUCGGCUGCUGCAACACGAUCGAUCUGUACUCCUCAAAUGAGAUGUAGCCAUCCUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAAUGGGGAAC ..(((((((((.(......))))).(((.(.(((((.(.((((((((((((..((((.((((.....)).)).))))..)))))))))))).)..))))).).)))......)))))... ( -44.70) >DroAna_CAF1 137454 118 - 1 AAUUCAAGCAGGCCCGGCUGGUUACGGACGACUCUGCUGUACUCCUCAUAUGUGAUUAUGCCAUCCUGGUCAUCAUCAAAUUUAAGGAGUAGAAAAUCGACCAUAUCCUAAAUUU--CAC ..(((.((((((..((.(((....))).))..)))))).(((((((......((((.(((((.....)).))).))))......)))))))))).....................--... ( -30.80) >consensus AACUCCAGCAGACGCGGCUGCUGCAACACGAUCGAUCUAUACUCCUCAAAUGAGAUGUAGCCAUCCUGGUCUUCAUCAAAUUUGAGGAGUAGAAAAUCGACCAUGUCCUAAAUGGGCAAC .....((((((......))))))......(.(((((...((((((((((((..((((.((((.....)).)).))))..))))))))))))....))))).).(((((.....))))).. (-34.90 = -36.77 + 1.86)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:02 2006