
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 8,466,820 – 8,466,980 |
| Length | 160 |
| Max. P | 0.812146 |

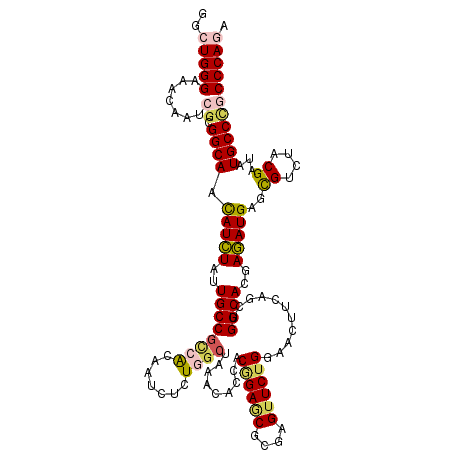
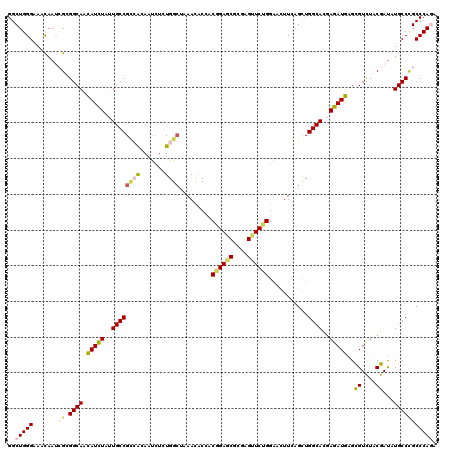
| Location | 8,466,820 – 8,466,940 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.44 |
| Mean single sequence MFE | -47.72 |
| Consensus MFE | -32.57 |
| Energy contribution | -33.08 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.97 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.812146 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
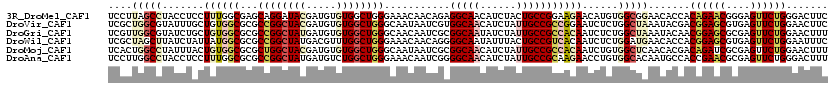
>3R_DroMel_CAF1 8466820 120 - 27905053 GGCUGGGAAACAACAGAGGCAACAUCUACUGCCGGAAGAACAUGUGGCGGAACACCACAGAACGGGAGUUCUGGGACUUCAGCUGGCACGAGAUGGGCGUCUACGAUUUGCCAGCCCAGG ((((((.(((....(((.((..(((((..((((((..(((..(((......)))((.((((((....))))))))..)))..))))))..))))).)).)))....))).)))))).... ( -50.10) >DroVir_CAF1 4003 120 - 1 GGCUGGGCAAUAAUCGUGGCAACAUCUAUUGCCGCCGGAAUCUCUGGCUAAAUACGACGGAGCGUGAGUUCUGGAACUUCAGCUGGCACGAGAUGAGCGUCUACGAUAUGCCCGCCCAGA (((.(((((...((((((((..(((((..(((((((((.....))))).........((((((....))))))...........))))..)))))...).))))))).)))))))).... ( -52.40) >DroGri_CAF1 4299 120 - 1 GGCUGGGCAACAAUCGCGGCAAUAUCUAUUGCCGCCACAAUCUCUGGCUAAAUACAACGGAGCGCGAGUUCUGGAACUUUAGCUGGCAUGAGAUGAGCGUUUACGAUAUGCCCGCCCAAA (((.(((((...(((((((((((....))))))))....(((((.(((((((.....((((((....))))))....))))))).....)))))..........))).)))))))).... ( -48.40) >DroWil_CAF1 5888 120 - 1 GGCUGGGAAACAACAGGGGCAAUAUUUACUGCCGUCACAAUCUCUGGAUGAACACCACGGAGCGUGAGUUCUGGAAUUUCAGUUGGCACGAAAUGAGUGUCUACGACAUGCCUGCCCAGA ..(((((......((((((((........))))(((............((((.....((((((....))))))....))))((.(((((.......))))).)))))...))))))))). ( -41.20) >DroMoj_CAF1 3977 120 - 1 GGCUGGGCAAUAAUCGCGGCAACAUCUAUUGCCGCCACAAUCUGUGGCUCAACACGACAGAUCGCGAGUUCUGGAACUUUAGCUGGCACGAGAUGAGCGUCUACGAUAUGCCCGCCCAGA (((.(((((...((((.(((..(((((..(((((((((.....))))).........((((.(....).))))...........))))..)))))...)))..)))).)))))))).... ( -45.60) >DroAna_CAF1 9175 120 - 1 GGCUGGGAAACAAUCGGGGCAACAUCUAUUGCCGCAAGAACCUGUGGCACAAUGCCACCGAACGCGAGUUCUGGGACUUUAGCUGGCACGAGAUGGGCGUCUACGAUCUGCCCGCCCAAG (((.(((.....((((((((..(((((..(((((((((..((.(((((.....))))).((((....)))).))..)))..)).))))..)))))...)))).))))...)))))).... ( -48.60) >consensus GGCUGGGAAACAAUCGCGGCAACAUCUAUUGCCGCCACAAUCUCUGGCUAAACACCACGGAGCGCGAGUUCUGGAACUUCAGCUGGCACGAGAUGAGCGUCUACGAUAUGCCCGCCCAGA ..(((((.......((.((((.(((((..((((((((.......)))).........((((((....))))))...........))))..)))))..((....))...))))))))))). (-32.57 = -33.08 + 0.50)

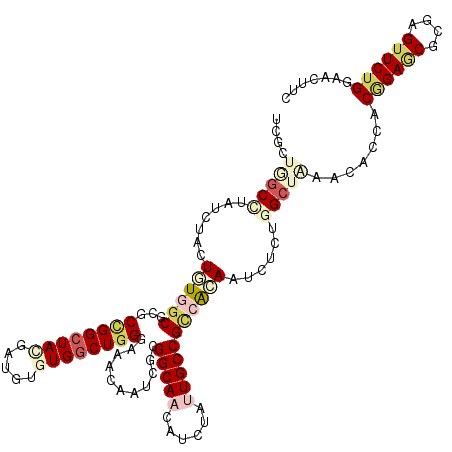
| Location | 8,466,860 – 8,466,980 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.50 |
| Mean single sequence MFE | -42.59 |
| Consensus MFE | -30.08 |
| Energy contribution | -32.14 |
| Covariance contribution | 2.06 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.619478 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8466860 120 - 27905053 UCCUUAGCCUACCUCCUUUGGCGAGCAGGAUACGAUGUGUGGCUGGGAAACAACAGAGGCAACAUCUACUGCCGGAAGAACAUGUGGCGGAACACCACAGAACGGGAGUUCUGGGACUUC ((((..(((..........)))....))))...(((((((..((((....)..)))..)).)))))..((((((.(......).))))))....((.((((((....))))))))..... ( -39.40) >DroVir_CAF1 4043 120 - 1 UCGCUGGCGUAUUUGCUGUGGCGCGCCGGCUACGAUGUGUGGCUGGGCAAUAAUCGUGGCAACAUCUAUUGCCGCCGGAAUCUCUGGCUAAAUACGACGGAGCGUGAGUUCUGGAACUUC ..(((((((....))))..)))((.((((((((.....)))))))))).....(((((((((......)))))(((((.....))))).....))))((((((....))))))....... ( -49.80) >DroGri_CAF1 4339 120 - 1 UCGUUGGCGUAUCUGCUGUGGCGCGCCGGCUAUGAUGUGUGGCUGGGCAACAAUCGCGGCAAUAUCUAUUGCCGCCACAAUCUCUGGCUAAAUACAACGGAGCGCGAGUUCUGGAACUUU ...(((((((....))((((((((.((((((((.....)))))))))).........((((((....)))))))))))).......)))))......((((((....))))))....... ( -47.10) >DroWil_CAF1 5928 120 - 1 UCGCUAGCUUAUCUAUUAUGGCGCGCCGGCUAUGACGUUUGGCUGGGAAACAACAGGGGCAAUAUUUACUGCCGUCACAAUCUCUGGAUGAACACCACGGAGCGUGAGUUCUGGAAUUUC ((.((((((......(((((((......))))))).....)))))))).......((((((........))))(((.((.....))))).....)).((((((....))))))....... ( -34.20) >DroMoj_CAF1 4017 120 - 1 UCACUGGCCUAUUUACUGUGGCGCGCUGGCUACGAUGUGUGGCUGGGCAAUAAUCGCGGCAACAUCUAUUGCCGCCACAAUCUGUGGCUCAACACGACAGAUCGCGAGUUCUGGAACUUU ....(((((.......((((((((.(..(((((.....)))))..))).........(((((......)))))))))))......))).))......((((.(....).))))....... ( -42.12) >DroAna_CAF1 9215 120 - 1 UCCUUGGCCUACCUCCUUUGGCGCGCCGGCUAUGAUGUCUGGCUGGGAAACAAUCGGGGCAACAUCUAUUGCCGCAAGAACCUGUGGCACAAUGCCACCGAACGCGAGUUCUGGGACUUU ((((.(..((.((......))(((((((((((.(....)))))))).......((((((((........(((((((......)))))))...)))).)))).))))))..).)))).... ( -42.90) >consensus UCGCUGGCCUAUCUACUGUGGCGCGCCGGCUACGAUGUGUGGCUGGGAAACAAUCGCGGCAACAUCUAUUGCCGCCACAAUCUCUGGCUAAACACCACGGAGCGCGAGUUCUGGAACUUC ....(((((.......((((((...((((((((.....))))))))...........(((((......)))))))))))......))))).......((((((....))))))....... (-30.08 = -32.14 + 2.06)
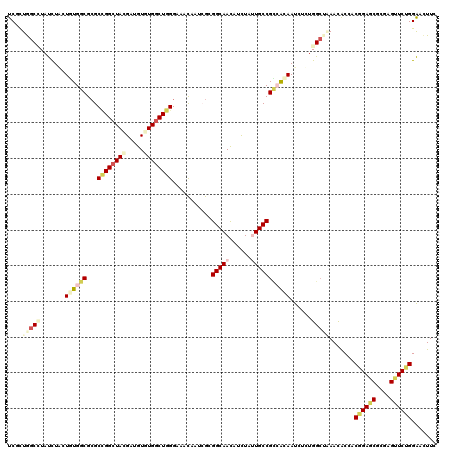
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:23 2006