
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 8,457,030 – 8,457,185 |
| Length | 155 |
| Max. P | 0.780016 |

| Location | 8,457,030 – 8,457,145 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.60 |
| Mean single sequence MFE | -50.08 |
| Consensus MFE | -31.97 |
| Energy contribution | -33.03 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.679092 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8457030 115 + 27905053 UUAACCUGGUGGCCACAGCGCCUAGCAGCAAUGCGGCCGAAGAGGCAGCUGUUGUCGGC-ACAGCACCUCCGUCAGCAGCGACGAACAGUUCAGUGGUGGCCUCACCAGG----AGGAGC ....((((((((((((.(((((..(((....)))))).(((((((..(((((.(....)-))))).))))((((......)))).....))).)).))))))..))))))----...... ( -48.90) >DroSec_CAF1 2958 115 + 1 UUAACCUGGUGGCCACAGCGCCUAGCAGCAAUGCGGCCGAAGAGGCAGCUGUUGUUGGC-ACAGCACCUCCGACGGCGGCCACCAAUAGUUCAGUGGUGGCCACACCAGG----AGGAGC ....(((((((((((((((.....(((....))).(((.....))).))))).(((((.-.........))))))))((((((((.........)))))))).)))))))----...... ( -55.60) >DroSim_CAF1 2998 115 + 1 UUAACCUGGUGGCCACAGCGCCUAGCAGCAAUGCGGCCGAAGAGGCAGCUGUUGUUGGC-ACAGCACCUCCGACGGCGGCCACCAACAGUUCAGUGGUGGCCACACCAGG----AGGAGC ....(((((((((((((((.....(((....))).(((.....))).))))).(((((.-.........))))))))((((((((.(......))))))))).)))))))----...... ( -56.20) >DroEre_CAF1 3089 115 + 1 UUAACCUGGUGGCCACAGCGCCUAGCAGCAAUUCGGCCGAAGAGGCGGCUGCUGUCGGC-ACAGCACCGACCUCGACGGCCACAAACAGUUCAGUGGCGGCAACAUCAGG----AGGAGC ....((((((.(((((.((.....))........(((((..(((((((.((((((....-))))))))).))))..)))))............)))))(....)))))))----...... ( -53.60) >DroYak_CAF1 3051 115 + 1 UUAACCUGGUUGCCACAGCGCCUAGCAGCAAUUCGGCCGAAGAGGCGGCUGUUGUCGGC-UCAGCACCCGCCACGGCGACCACGAACAGUUCUGUGGUGGCCACCCCAGG----AGGAGC ....(((((((((....((.....)).))))...((.......(((((.(((((.....-.))))).)))))..(((.((((((........)))))).))).)))))))----...... ( -45.20) >DroPer_CAF1 2453 112 + 1 UGAACCUGGUGGACAAGGCGCCGAGCAGCAAUC---CCGAUGAGGUCG----UCCCGGCGACAGCCACAACCGCCGCGGCCACGCCCAGUACAGUGGUGGCUGCC-CAGGGCUCGGAUGC .((.((((((((....(((((......))....---........((((----(....))))).)))....)))))((((((((((........)).)))))))).-..))).))...... ( -41.00) >consensus UUAACCUGGUGGCCACAGCGCCUAGCAGCAAUGCGGCCGAAGAGGCAGCUGUUGUCGGC_ACAGCACCUCCCACGGCGGCCACGAACAGUUCAGUGGUGGCCACACCAGG____AGGAGC ....(((((((......((((((((((((......(((.....))).)))))))..)))....))..........((.(((((..........))))).))..))))))).......... (-31.97 = -33.03 + 1.06)



| Location | 8,457,030 – 8,457,145 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.60 |
| Mean single sequence MFE | -53.60 |
| Consensus MFE | -32.96 |
| Energy contribution | -35.77 |
| Covariance contribution | 2.81 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.757644 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8457030 115 - 27905053 GCUCCU----CCUGGUGAGGCCACCACUGAACUGUUCGUCGCUGCUGACGGAGGUGCUGU-GCCGACAACAGCUGCCUCUUCGGCCGCAUUGCUGCUAGGCGCUGUGGCCACCAGGUUAA ......----(((((((((((.........(((.(((((((....))))))))))(((((-.......))))).)))))...(((((((.((((....)))).))))))))))))).... ( -50.50) >DroSec_CAF1 2958 115 - 1 GCUCCU----CCUGGUGUGGCCACCACUGAACUAUUGGUGGCCGCCGUCGGAGGUGCUGU-GCCAACAACAGCUGCCUCUUCGGCCGCAUUGCUGCUAGGCGCUGUGGCCACCAGGUUAA ......----((((((((((((((((.........))))))))))....(((((((((((-.......))))).))))))..(((((((.((((....)))).))))))))))))).... ( -61.00) >DroSim_CAF1 2998 115 - 1 GCUCCU----CCUGGUGUGGCCACCACUGAACUGUUGGUGGCCGCCGUCGGAGGUGCUGU-GCCAACAACAGCUGCCUCUUCGGCCGCAUUGCUGCUAGGCGCUGUGGCCACCAGGUUAA ......----(((((((((((((((((......).))))))))))....(((((((((((-.......))))).))))))..(((((((.((((....)))).))))))))))))).... ( -62.20) >DroEre_CAF1 3089 115 - 1 GCUCCU----CCUGAUGUUGCCGCCACUGAACUGUUUGUGGCCGUCGAGGUCGGUGCUGU-GCCGACAGCAGCCGCCUCUUCGGCCGAAUUGCUGCUAGGCGCUGUGGCCACCAGGUUAA ......----((((..((.(((((..............((((((..((((.(((((((((-....)))))).)))))))..))))))....(((....)))...))))).)))))).... ( -48.10) >DroYak_CAF1 3051 115 - 1 GCUCCU----CCUGGGGUGGCCACCACAGAACUGUUCGUGGUCGCCGUGGCGGGUGCUGA-GCCGACAACAGCCGCCUCUUCGGCCGAAUUGCUGCUAGGCGCUGUGGCAACCAGGUUAA ......----((((((((((((((.(((....)))..)))))))))(.(((((.((.((.-.....)).)).))))).)....((((...((((....))))...))))..))))).... ( -49.60) >DroPer_CAF1 2453 112 - 1 GCAUCCGAGCCCUG-GGCAGCCACCACUGUACUGGGCGUGGCCGCGGCGGUUGUGGCUGUCGCCGGGA----CGACCUCAUCGG---GAUUGCUGCUCGGCGCCUUGUCCACCAGGUUCA ......(((((.((-(((((((((.(((((....(((...)))...))))).))))))))(((((((.----((((((....))---).)))...))))))).........)))))))). ( -50.20) >consensus GCUCCU____CCUGGUGUGGCCACCACUGAACUGUUCGUGGCCGCCGUCGGAGGUGCUGU_GCCGACAACAGCUGCCUCUUCGGCCGCAUUGCUGCUAGGCGCUGUGGCCACCAGGUUAA ..........((((((((((((((.............)))))))).......((((((((........))))).))).....(((((((.((((....)))).))))))))))))).... (-32.96 = -35.77 + 2.81)



| Location | 8,457,070 – 8,457,185 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.57 |
| Mean single sequence MFE | -54.93 |
| Consensus MFE | -29.73 |
| Energy contribution | -30.93 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.780016 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8457070 115 + 27905053 AGAGGCAGCUGUUGUCGGC-ACAGCACCUCCGUCAGCAGCGACGAACAGUUCAGUGGUGGCCUCACCAGG----AGGAGCAGCUGUGGAGGGAGCUGUGCCCGGUCUGGGCGGUAGUCUG ..((((.(((((.((((((-(((((.((((((.((((.((..(((.....))..(((((....)))))..----.)..)).))))))))))..)))))).)))).)...))))).)))). ( -53.00) >DroSec_CAF1 2998 115 + 1 AGAGGCAGCUGUUGUUGGC-ACAGCACCUCCGACGGCGGCCACCAAUAGUUCAGUGGUGGCCACACCAGG----AGGAGCAGCUGUGGAGGGAGCUGUGCCCGGUCUGGGCGGUAGUCUG ..((((.((((((...(((-(((((.(((((.(((((((((((((.........))))))))...((...----.))....))))))))))..)))))))).......)))))).)))). ( -59.40) >DroSim_CAF1 3038 115 + 1 AGAGGCAGCUGUUGUUGGC-ACAGCACCUCCGACGGCGGCCACCAACAGUUCAGUGGUGGCCACACCAGG----AGGAGCAGCUAUGGAGGGAGCUGUGCCCGGUCUGGGCGGAAGUCUG ..(((((((....)))(((-(((((.((((((..(((((((((((.(......)))))))))...((...----.))....))).))))))..))))))))..))))((((....)))). ( -57.10) >DroEre_CAF1 3129 115 + 1 AGAGGCGGCUGCUGUCGGC-ACAGCACCGACCUCGACGGCCACAAACAGUUCAGUGGCGGCAACAUCAGG----AGGAGCAGCUGUGGAGGGAGCUGUAACCGGACUGGGCGGGAGUUUG .(((((((.((((((....-))))))))).))))(((..((.(...((((((..((..(....)..))..----.((.((((((........))))))..)))))))).).))..))).. ( -44.80) >DroYak_CAF1 3091 115 + 1 AGAGGCGGCUGUUGUCGGC-UCAGCACCCGCCACGGCGACCACGAACAGUUCUGUGGUGGCCACCCCAGG----AGGAGCAGCUGUGGAGGGAGCUGUACCCGGUCUGGGCGGUAGCUUG .(((.((((....)))).)-))(((..(((((..(((.((((((........)))))).)))....((((----.((.((((((........)))))).))...)))))))))..))).. ( -55.80) >DroPer_CAF1 2490 115 + 1 UGAGGUCG----UCCCGGCGACAGCCACAACCGCCGCGGCCACGCCCAGUACAGUGGUGGCUGCC-CAGGGCUCGGAUGCGGCGGUGGCACGCGCCGCGGGGGACACCAGUGGCAGCCUU ((..(((.----((((((((...(((...(((((((((.((..((((.(..((((....))))..-).))))..)).))))))))))))...))))).))).)))..))........... ( -59.50) >consensus AGAGGCAGCUGUUGUCGGC_ACAGCACCUCCCACGGCGGCCACGAACAGUUCAGUGGUGGCCACACCAGG____AGGAGCAGCUGUGGAGGGAGCUGUGCCCGGUCUGGGCGGUAGUCUG ..((((.((((((.((((...((((.(((.(.(((((.(((((..........))))).((..(...........)..)).))))).).))).))))...))))....)))))).)))). (-29.73 = -30.93 + 1.20)



| Location | 8,457,070 – 8,457,185 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.57 |
| Mean single sequence MFE | -48.67 |
| Consensus MFE | -24.21 |
| Energy contribution | -25.02 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.653451 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 8457070 115 - 27905053 CAGACUACCGCCCAGACCGGGCACAGCUCCCUCCACAGCUGCUCCU----CCUGGUGAGGCCACCACUGAACUGUUCGUCGCUGCUGACGGAGGUGCUGU-GCCGACAACAGCUGCCUCU (((.((..((.......))((((((((..(((((.((((.((....----..(((((....)))))..((.....))...)).))))..))))).)))))-)))......)))))..... ( -40.50) >DroSec_CAF1 2998 115 - 1 CAGACUACCGCCCAGACCGGGCACAGCUCCCUCCACAGCUGCUCCU----CCUGGUGUGGCCACCACUGAACUAUUGGUGGCCGCCGUCGGAGGUGCUGU-GCCAACAACAGCUGCCUCU ..(((.(((((((.....)))).(((((........))))).....----...)))((((((((((.........)))))))))).)))(((((((((((-.......))))).)))))) ( -52.70) >DroSim_CAF1 3038 115 - 1 CAGACUUCCGCCCAGACCGGGCACAGCUCCCUCCAUAGCUGCUCCU----CCUGGUGUGGCCACCACUGAACUGUUGGUGGCCGCCGUCGGAGGUGCUGU-GCCAACAACAGCUGCCUCU ..(((..((((((.....)))).(((((........))))).....----...)).(((((((((((......).)))))))))).)))(((((((((((-.......))))).)))))) ( -51.90) >DroEre_CAF1 3129 115 - 1 CAAACUCCCGCCCAGUCCGGUUACAGCUCCCUCCACAGCUGCUCCU----CCUGAUGUUGCCGCCACUGAACUGUUUGUGGCCGUCGAGGUCGGUGCUGU-GCCGACAGCAGCCGCCUCU .........((...(((.((...(((((........))))).....----)).)))...)).(((((..........)))))....((((.(((((((((-....)))))).))))))). ( -38.80) >DroYak_CAF1 3091 115 - 1 CAAGCUACCGCCCAGACCGGGUACAGCUCCCUCCACAGCUGCUCCU----CCUGGGGUGGCCACCACAGAACUGUUCGUGGUCGCCGUGGCGGGUGCUGA-GCCGACAACAGCCGCCUCU ..(((..((((((...(((((..(((((........))))).....----)))))(((((((((.(((....)))..)))))))))).)))))..)))((-(.((........)).))). ( -49.20) >DroPer_CAF1 2490 115 - 1 AAGGCUGCCACUGGUGUCCCCCGCGGCGCGUGCCACCGCCGCAUCCGAGCCCUG-GGCAGCCACCACUGUACUGGGCGUGGCCGCGGCGGUUGUGGCUGUCGCCGGGA----CGACCUCA ............((((((((..(((((((.(((.((((((((..(((.((((..-.((((......))))...)))).)))..)))))))).))))).))))).))))----).)))... ( -58.90) >consensus CAGACUACCGCCCAGACCGGGCACAGCUCCCUCCACAGCUGCUCCU____CCUGGUGUGGCCACCACUGAACUGUUCGUGGCCGCCGUCGGAGGUGCUGU_GCCGACAACAGCUGCCUCU .........((((.....)))).(((((........)))))...............((((((((.............)))))))).......((((((((........))))).)))... (-24.21 = -25.02 + 0.81)
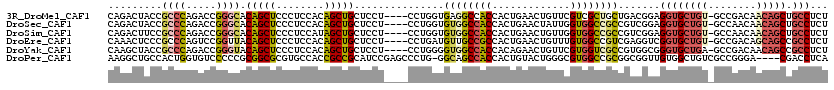
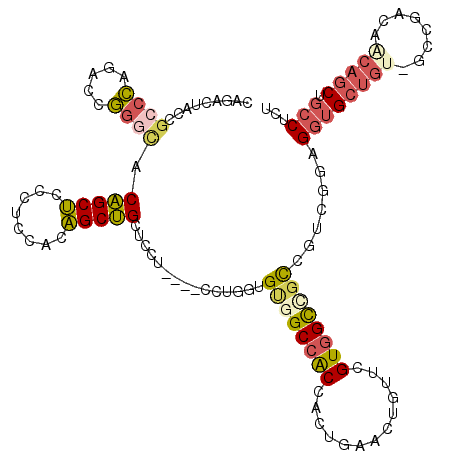
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:07 2006