
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
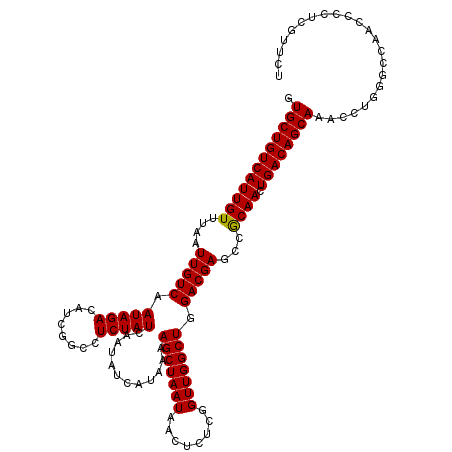
| Location | 8,097,899 – 8,098,019 |
| Length | 120 |
| Max. P | 0.906916 |

| Location | 8,097,899 – 8,098,019 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.67 |
| Mean single sequence MFE | -31.30 |
| Consensus MFE | -29.00 |
| Energy contribution | -28.84 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.906916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
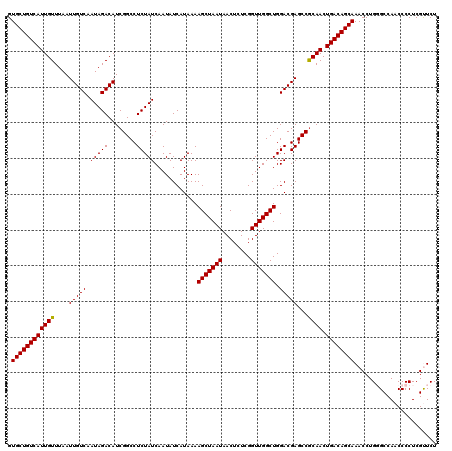
>3R_DroMel_CAF1 8097899 120 + 27905053 GUGCUGUCAUUGUUUAAUUGUCAAUAGACAUCGGCCUCUAUCAAUAUCAUAAAAGCUAAUAACUCUCGGUUGGCUGGACGAGCCACAACUGACAGCAAACCUGGGCCAACCCCUCGUUCU .((((((((((((....(((((.(((((........)))))............(((((((........))))))).)))))...)))).))))))))(((..(((.....)))..))).. ( -32.70) >DroSec_CAF1 49561 120 + 1 GUGCUGUCAUUGUUUAAUUGUCAAUAGACAUCGGCCUCUAUCAAUAUCAUAAAAGCUAAUAACUCUCGGUUGGCUGGACGAGCCGCAACUGACAGCAAACCUGGGCCAACCCCUCGUUCU .((((((((((((....(((((.(((((........)))))............(((((((........))))))).)))))...)))).))))))))(((..(((.....)))..))).. ( -33.00) >DroSim_CAF1 21645 120 + 1 GUGCUGUCAUUGUUUAAUUGUCAAUAGACAUCGGCCUCUAUCAAUAUCAUAAAAGCUAAUAACUCUCGGUUGGCUGGACGAGCCGCAACUGACAGCAAACCUGGGCCAACCCCUCGUUCU .((((((((((((....(((((.(((((........)))))............(((((((........))))))).)))))...)))).))))))))(((..(((.....)))..))).. ( -33.00) >DroEre_CAF1 24637 119 + 1 GUGCUGUCAUUGUUUAAUUGUCAAUAGACAUCGGCCUCUAUCAAUAUCAUAAAAGCUAAUAACUCUCGGUUGGCUGGACGAGCCGCAACUGACAGCAAACCUG-GCCAACCCCUCGCUCU .((((((((((((....(((((.(((((........)))))............(((((((........))))))).)))))...)))).))))))))......-................ ( -28.90) >DroYak_CAF1 18027 119 + 1 GUGCUGUCAUUGUUUAAUUGUCAAUAGACAUCGGCCUCUAUCAAUAUCAUAAAAGCUAAUAACUCUCGGUUGGCUGGACGAGCCGCAACUGACAGCAAACCUG-GCCAACCCCUCGCUCU .((((((((((((....(((((.(((((........)))))............(((((((........))))))).)))))...)))).))))))))......-................ ( -28.90) >consensus GUGCUGUCAUUGUUUAAUUGUCAAUAGACAUCGGCCUCUAUCAAUAUCAUAAAAGCUAAUAACUCUCGGUUGGCUGGACGAGCCGCAACUGACAGCAAACCUGGGCCAACCCCUCGUUCU .((((((((((((....(((((.(((((........)))))............(((((((........))))))).)))))...)))).))))))))....................... (-29.00 = -28.84 + -0.16)

| Location | 8,097,899 – 8,098,019 |
|---|---|
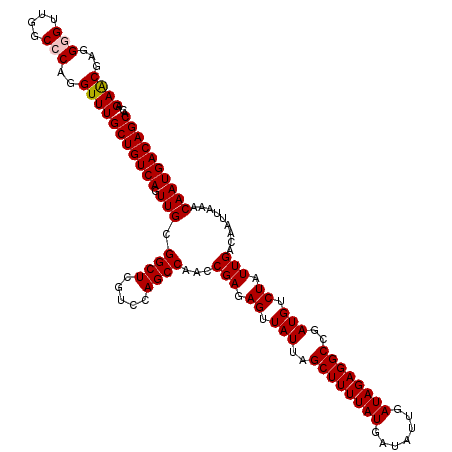
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.67 |
| Mean single sequence MFE | -37.10 |
| Consensus MFE | -33.58 |
| Energy contribution | -33.74 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.764146 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
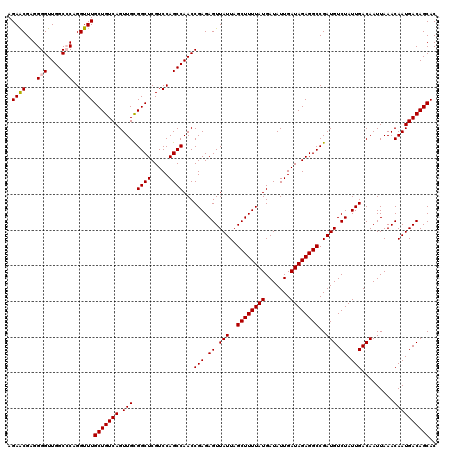
>3R_DroMel_CAF1 8097899 120 - 27905053 AGAACGAGGGGUUGGCCCAGGUUUGCUGUCAGUUGUGGCUCGUCCAGCCAACCGAGAGUUAUUAGCUUUUAUGAUAUUGAUAGAGGCCGAUGUCUAUUGACAAUUAAACAAUGACAGCAC .((((...(((....)))..))))(((((((.((((((((.....))))...(((.((.(((..((((((((.......))))))))..))).)).)))........))))))))))).. ( -37.90) >DroSec_CAF1 49561 120 - 1 AGAACGAGGGGUUGGCCCAGGUUUGCUGUCAGUUGCGGCUCGUCCAGCCAACCGAGAGUUAUUAGCUUUUAUGAUAUUGAUAGAGGCCGAUGUCUAUUGACAAUUAAACAAUGACAGCAC .((((...(((....)))..))))(((((((.(((.((((.....))))...(((.((.(((..((((((((.......))))))))..))).)).))).........)))))))))).. ( -36.40) >DroSim_CAF1 21645 120 - 1 AGAACGAGGGGUUGGCCCAGGUUUGCUGUCAGUUGCGGCUCGUCCAGCCAACCGAGAGUUAUUAGCUUUUAUGAUAUUGAUAGAGGCCGAUGUCUAUUGACAAUUAAACAAUGACAGCAC .((((...(((....)))..))))(((((((.(((.((((.....))))...(((.((.(((..((((((((.......))))))))..))).)).))).........)))))))))).. ( -36.40) >DroEre_CAF1 24637 119 - 1 AGAGCGAGGGGUUGGC-CAGGUUUGCUGUCAGUUGCGGCUCGUCCAGCCAACCGAGAGUUAUUAGCUUUUAUGAUAUUGAUAGAGGCCGAUGUCUAUUGACAAUUAAACAAUGACAGCAC ...((....(((((((-..((...(((((.....)))))....)).)))))))....((((((.((((((((.......))))))))...((((....)))).......)))))).)).. ( -37.40) >DroYak_CAF1 18027 119 - 1 AGAGCGAGGGGUUGGC-CAGGUUUGCUGUCAGUUGCGGCUCGUCCAGCCAACCGAGAGUUAUUAGCUUUUAUGAUAUUGAUAGAGGCCGAUGUCUAUUGACAAUUAAACAAUGACAGCAC ...((....(((((((-..((...(((((.....)))))....)).)))))))....((((((.((((((((.......))))))))...((((....)))).......)))))).)).. ( -37.40) >consensus AGAACGAGGGGUUGGCCCAGGUUUGCUGUCAGUUGCGGCUCGUCCAGCCAACCGAGAGUUAUUAGCUUUUAUGAUAUUGAUAGAGGCCGAUGUCUAUUGACAAUUAAACAAUGACAGCAC .((((...(((....)))..))))(((((((.(((.((((.....))))...(((.((.(((..((((((((.......))))))))..))).)).))).........)))))))))).. (-33.58 = -33.74 + 0.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:51:52 2006