
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 7,839,231 – 7,839,391 |
| Length | 160 |
| Max. P | 0.769229 |

| Location | 7,839,231 – 7,839,351 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.39 |
| Mean single sequence MFE | -39.33 |
| Consensus MFE | -23.79 |
| Energy contribution | -24.22 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.648255 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7839231 120 - 27905053 AUGCUGCUGCAUCAGGUCGUGUUGGAAUUGCAACUGGCGUUGGUUUUCUAGCGACAGCAGGUCGUUUUGCUGACGCAGUUCGUCAACUAGAGAGACGAGCUCUUUGCUACGCUUUUGCAG ......(((((..(((.((((((((((((((......((((((....)))))).(((((((....)))))))..)))))))..))))(((((((.....)))))))..)))))).))))) ( -40.20) >DroPse_CAF1 2491 120 - 1 AUGCUGCUGCAUAAGGUCGUGCUGGAAUUGCACCUGGCGUUGGUUUUCCAGCGUUACGGUGUCGUUCUGCUGACGCAGAUCGUCCACCAGUAUUAGGAGUUCUCUGCUACGUUUCUGCAG ((((....))))...((((.((.(((((.((((((((((((((....))))))))).))))).)))))))))))(((((.(((...((.......))(((.....))))))..))))).. ( -45.20) >DroWil_CAF1 2398 120 - 1 AUGUUGUUGCAUUAGCUCCUGUUGGAAUUUCAAUUGACUUUGGUUCUCUAUAGUGAGUAUAUCGUUUUGCUGACGUAGUUCCUCUACUAGUUUCAGGAAGUCCUUGCUGCGUUUUUGUAG .....((.(((...(((((((.((((...((((......))))...))))..((.((((........)))).))((((.....))))......)))).)))...))).)).......... ( -24.20) >DroYak_CAF1 2482 120 - 1 AUGCUGCUGCAUCAGGUCGUGUUGGAAUUGCAACUGGCGUUGGUUUUCUAGCGUCAGCAGGUCAUUUUGCUGACGCAGUUCGUCAACUAAUGAAAGAAGCUCUUUGCUACGCUUUUGCAG ......(((((..(((.(((((((......)))).((((..(..(((((.(((((((((((....)))))))))))..(((((......))))))))))..)..)))))))))).))))) ( -40.80) >DroAna_CAF1 2463 120 - 1 AUGCUUCUGCAUCAGAUCCUGCUGGAAUUGCAUUUGGCGUUGAUUCUCUAGCGUGAGUAGGUCAUUCUGUUGACGCAGUUCGUCAACCAACGCCGAGAGUUCCUUGCUUCGCUUCUGCAG ......(((((..((.....((.((((((...((((((((((.....((.((....)).)).......(((((((.....)))))))))))))))))))))))..))....))..))))) ( -40.40) >DroPer_CAF1 2491 120 - 1 AUGCUGCUGCAUAAGGUCGUGCUGGAAUUGCACCUGGCGUUGGUUUUCCAGCGUUACGGUGUCGUUCUGCUGACGCAGAUCGUCCACCAGUAUUAGGAGUUCUCUGCUACGUUUCUGCAG ((((....))))...((((.((.(((((.((((((((((((((....))))))))).))))).)))))))))))(((((.(((...((.......))(((.....))))))..))))).. ( -45.20) >consensus AUGCUGCUGCAUCAGGUCGUGCUGGAAUUGCAACUGGCGUUGGUUUUCUAGCGUCAGCAGGUCGUUCUGCUGACGCAGUUCGUCAACCAGUGUCAGGAGUUCUUUGCUACGCUUCUGCAG ......(((((..((.........(((((((..((((((((((....))))))))))...((((......)))))))))))................(((.....)))...))..))))) (-23.79 = -24.22 + 0.42)



| Location | 7,839,271 – 7,839,391 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.39 |
| Mean single sequence MFE | -45.55 |
| Consensus MFE | -27.80 |
| Energy contribution | -27.72 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.769229 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7839271 120 - 27905053 GCAACUGGUGCCCGGCGUCCAGCUCAUCAACUUGCUGGCGAUGCUGCUGCAUCAGGUCGUGUUGGAAUUGCAACUGGCGUUGGUUUUCUAGCGACAGCAGGUCGUUUUGCUGACGCAGUU (((((((((((.(((((((((((..........)))))..))))))..))))))).((......)).))))(((((.(((..((.....((((((.....))))))..))..)))))))) ( -47.40) >DroPse_CAF1 2531 120 - 1 GUGCUUGGUAGCCGACAUCCAUUUGGUCCAUUUGUUGGCGAUGCUGCUGCAUAAGGUCGUGCUGGAAUUGCACCUGGCGUUGGUUUUCCAGCGUUACGGUGUCGUUCUGCUGACGCAGAU .(((......(((((((.((....))......))))))).((((....))))...((((.((.(((((.((((((((((((((....))))))))).))))).))))))))))))))... ( -48.50) >DroEre_CAF1 2518 120 - 1 GCAACUGGUGCCCGGCGUCCAGCUCAUCAACUUGUUGGCGAUGCUGCUGCAUCAGAUCGUGUUGGAAUUGCAACUGGCGUUUGUUUUCUAACGACAGCAGGUCGUUUUGCUGACGCAAUU ((..(((((((.(((((((((((..........)))))..))))))..)))))))...(((((((((..((((.......)))).)))))))..(((((((....))))))))))).... ( -41.30) >DroYak_CAF1 2522 120 - 1 GCAACUGGUGCGCGGCGUCUAGUUCAUCUACUUGUUGGCGAUGCUGCUGCAUCAGGUCGUGUUGGAAUUGCAACUGGCGUUGGUUUUCUAGCGUCAGCAGGUCAUUUUGCUGACGCAGUU .((((((((((((((((((.....((......)).....)))))))).)))))).((((.((((......))))))))))))........(((((((((((....))))))))))).... ( -49.80) >DroAna_CAF1 2503 120 - 1 UCGUUUGGUGCGUGGCUUCUAAUUGGUCCACCUGCUGGCGAUGCUUCUGCAUCAGAUCCUGCUGGAAUUGCAUUUGGCGUUGAUUCUCUAGCGUGAGUAGGUCAUUCUGUUGACGCAGUU ........(((((((((.......)).))))(((((.(((((((....))))).......((((((....((........))....)))))))).)))))((((......)))))))... ( -37.80) >DroPer_CAF1 2531 120 - 1 GUGCUUGGUAGCCGACAUCCAUUUGGUCCAUUUGUUGGCGAUGCUGCUGCAUAAGGUCGUGCUGGAAUUGCACCUGGCGUUGGUUUUCCAGCGUUACGGUGUCGUUCUGCUGACGCAGAU .(((......(((((((.((....))......))))))).((((....))))...((((.((.(((((.((((((((((((((....))))))))).))))).))))))))))))))... ( -48.50) >consensus GCAACUGGUGCCCGGCGUCCAGUUCAUCCACUUGUUGGCGAUGCUGCUGCAUCAGGUCGUGCUGGAAUUGCAACUGGCGUUGGUUUUCUAGCGUCAGCAGGUCGUUCUGCUGACGCAGUU ....((((..........)))).......(((((((((((((((....))))).......(((((((.(((.....)))......)))))))))))))))))....((((....)))).. (-27.80 = -27.72 + -0.08)

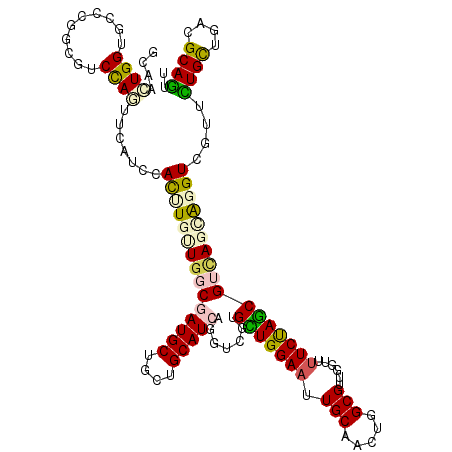
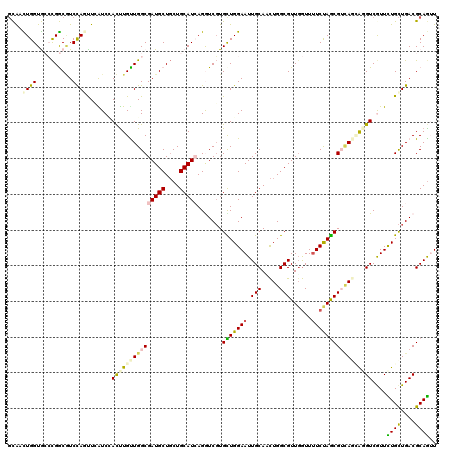
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:47:50 2006