| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 1,118,335 – 1,118,445 |
| Length | 110 |
| Max. P | 0.951092 |

| Location | 1,118,335 – 1,118,445 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.09 |
| Mean single sequence MFE | -40.05 |
| Consensus MFE | -28.79 |
| Energy contribution | -29.52 |
| Covariance contribution | 0.73 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.41 |
| SVM RNA-class probability | 0.951092 |
| Prediction | RNA |
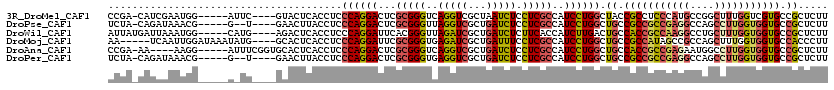
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1118335 110 + 27905053 CCGA-CAUCGAAUGG-----AUUC----GUACUCACCUCCCAGGACUCGCGGGUCAGGUCGCUAAUCUCCUCGCCAUCCUGGCUACCGCCUCCCAUGCCGGCUUUGGUCGUGCCGCUCUU ..((-.(((.....)-----))))----((((..(((..((((((...(((((..((((.....)))).)))))..)))))).....(((.........)))...))).))))....... ( -27.60) >DroPse_CAF1 58584 108 + 1 UCUA-CAGAUAAACG-----G--U----GAACUUACCUCCCAGGACUCGCGGGUUAGGUCGCUGAUCUCCUCGCCAUCCUGGCUGCCGCCGCCGAGGCCAGCCUUGGUGGUGCCGCUCUU ....-(((......(-----(--(----(....))))..((((((...(((((..(((((...))))).)))))..))))))))).(((((((((((....)))))))))))........ ( -43.50) >DroWil_CAF1 55171 111 + 1 AUUAUGAUUAAAUGG-----CAUG----AGACUCACCUCCCAGGAUUCACGGGUUAGAUCGCUGAUCUCUUCACCAUCUUGACUGCCACCGCCAAGGCCUGCUUUGGUGGUGCCGCUCUU .............((-----((.(----((......))).((((((....(((((((....))))))).......))))))..))))((((((((((....))))))))))......... ( -32.50) >DroMoj_CAF1 47108 111 + 1 AA-----UCAAUUGGAUAAAUAUG----GCACUCACCUCCCAGGAUUCGCGGGUGAGAUCGCUGAUUUCCUCGCCAUCCUGGCUGCCGCCAUAGCCGCCAGCUUUGGUGGUGCCACCCUU ..-----...............((----((((.((((...((((((..(((((.((((((...))))))))))).))))))(((((.((....)).).))))...))))))))))..... ( -44.80) >DroAna_CAF1 49803 110 + 1 CCGA-AA----AAGG-----AUUUCGGUGCACUCACCUCCCAGGACUCGCGGGUCAGGUCGCUGAUCUCCUCGCCAUCCUGGCUGCCACCGCCGAGAAUGGCCUUGGUGGUGCCGCUCUU ((((-((----....-----.)))))).((.........((((((...(((((..(((((...))))).)))))..))))))..(.((((((((((......)))))))))).))).... ( -43.50) >DroPer_CAF1 57960 108 + 1 UCUA-CAGAUAAACG-----G--U----GAACUUACCUCCCAGGACUCGCGGGUGAGGUCGCUGAUCUCCUCGCCAUCCUGGCUGCCGCCGCCGAGGCCAGCCUUGGUGGUGCCGCUCUU ....-(((......(-----(--(----(....))))..((((((...(((((.((((((...)))))))))))..))))))))).(((((((((((....)))))))))))........ ( -48.40) >consensus ACUA_CAUAAAAAGG_____A_UU____GAACUCACCUCCCAGGACUCGCGGGUCAGGUCGCUGAUCUCCUCGCCAUCCUGGCUGCCGCCGCCGAGGCCAGCCUUGGUGGUGCCGCUCUU .......................................((((((...(((((..(((((...))))).)))))..)))))).((.(((((((((((....))))))))))).))..... (-28.79 = -29.52 + 0.73)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:39:34 2006