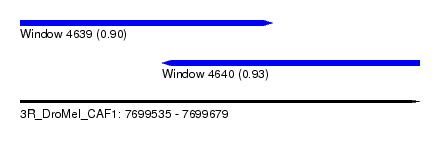
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 7,699,535 – 7,699,679 |
| Length | 144 |
| Max. P | 0.927816 |

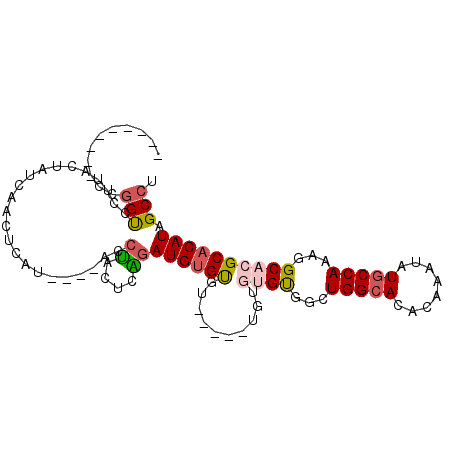
| Location | 7,699,535 – 7,699,626 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 76.66 |
| Mean single sequence MFE | -26.31 |
| Consensus MFE | -10.89 |
| Energy contribution | -11.37 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.41 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.903012 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7699535 91 + 27905053 ---------CCGGUCCUUACUAUCAACACAUACAUACCUAGUCAGAUGUGUGUA----UAUGUGUGGCUGGCACACAAAUAUGCCAAAGGCACGCACAUAGCCU ---------..(((.............((((((((..........)))))))).----(((((((((((((((........))))...))).))))))))))). ( -27.20) >DroSec_CAF1 49343 91 + 1 ---------CCGGUCCUUACUAUCAACUCAU----ACCUACUCAGAUGUGUGUGUGUUUGUGUGUGGCUGGCACACAAAUAUGCCAAAGGCACGCACAUAGCCA ---------..(((..............(((----((.(((......))).)))))..((((((((.((((((........))))..)).))))))))..))). ( -28.30) >DroSim_CAF1 49441 91 + 1 ---------CCGGUCCUUACUAUCAACUCAU----ACCUACUCAGAUGUGUGUGUUUGUGUGUGUGGCUGGCACACAAAUAUGCCAAAGGCACGCACAUAGCCU ---------..(((..........(((.(((----((..........))))).)))((((((((((.((((((........))))..)).))))))))))))). ( -28.60) >DroEre_CAF1 49830 83 + 1 ----CCUGGCUGGUCCUUACUAUC-ACUCAU----ACCUACUCAGAUGUGU------------GCUGUUGGCACACAAAUAUGCCAAAGGCACGCACAUAGCCU ----...(((((((..........-......----)))........(((((------------(((.((((((........)))))).))))))))...)))). ( -26.19) >DroYak_CAF1 51066 88 + 1 CUUCCCUGGCCGGUCCUUACUUUCAACUCAU----ACCCACUCGGAUGUGU------------GCGGUUGGCACACAAAUAUGCCAAAGGCACGCACAUAGCCU .......(((..((((...............----........))))((((------------((..((((((........))))))..)))))).....))). ( -25.10) >DroAna_CAF1 51340 87 + 1 ---------ACAGCCCUUCCUCUGAAUGCAU----ACCUCUUCGCAUGAGCGUU----UGCGUGUGUUUGGAACACAAAUAUGCCAAAGGCACGCACAUUGCCU ---------.(((........))).((((..----........))))..(((..----(((((((.(((((............))))).)))))))...))).. ( -22.50) >consensus _________CCGGUCCUUACUAUCAACUCAU____ACCUACUCAGAUGUGUGU_____UGUGUGUGGCUGGCACACAAAUAUGCCAAAGGCACGCACAUAGCCU ...........(((.......................((....))((((((..........((((...(((((........)))))...)))))))))).))). (-10.89 = -11.37 + 0.48)

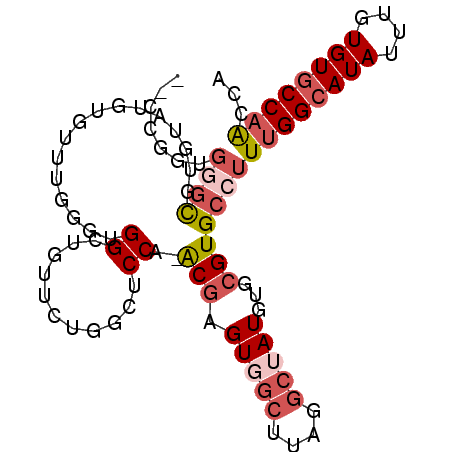
| Location | 7,699,586 – 7,699,679 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 72.64 |
| Mean single sequence MFE | -35.25 |
| Consensus MFE | -11.43 |
| Energy contribution | -12.73 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.32 |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.927816 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7699586 93 - 27905053 --CCGGUGGGCGUGUAUGUGUUUGGGUGGCUGUUUUGUCUCCAAACGAGUGGCUUAGGCUAUGUGCGUGCCUUUGGCAUAUUUGUGUGCCAGCCA --..(((((((((((((.((((((((.(((......))))))))))).(((((....))))))))))))))).(((((((....)))))))))). ( -39.60) >DroVir_CAF1 64792 75 - 1 --CCAGGUGACAUAGAUGUGUAUGCGU-----------------GCGCGUGG-UUAGGCAAUGCGCGUGCAUUUGGCAUAUUUGUGUGCCUACCC --..((((.((((((((((((((((((-----------------((((((.(-.....).))))))))))))...)))))))))))))))).... ( -33.70) >DroSec_CAF1 49394 93 - 1 --CCGGUGGGCGAGUGUGUGUUUGGAUGGCUGUUCUGGCUCCAAACGAGUGGCUUUGGCUAUGUGCGUGCCUUUGGCAUAUUUGUGUGCCAGCCA --..((((((((.(..(.((((((((..(((.....))))))))))).(((((....))))))..).))))).(((((((....)))))))))). ( -36.30) >DroSim_CAF1 49492 93 - 1 --CCGGUGGGCGAGUGUGUGUUUGGAUGGCUGUUCUGGCUCCAAACGAGUGGCUUAGGCUAUGUGCGUGCCUUUGGCAUAUUUGUGUGCCAGCCA --..((((((((.(..(.((((((((..(((.....))))))))))).(((((....))))))..).))))).(((((((....)))))))))). ( -36.30) >DroEre_CAF1 49873 88 - 1 --GCGGUGGGCGUGUGGGUGGGU----GGCUGUUCUGGCUCCC-ACGAGUGACUUAGGCUAUGUGCGUGCCUUUGGCAUAUUUGUGUGCCAACAG --..((((.(((..((((((((.----((((.....)))))))-))(((...)))...)))..))).)))).((((((((....))))))))... ( -35.50) >DroAna_CAF1 51387 88 - 1 AGCCUGGGAGUGGGAAUGGGCGUGGGCGGGA------GCGCCU-GCAAGUGGCUCAGGCAAUGUGCGUGCCUUUGGCAUAUUUGUGUUCCAAACA ......(((..(.(((((.(((..((((...------.)))).-.)......(..(((((.......)))))..))).))))).)..)))..... ( -30.10) >consensus __CCGGUGGGCGUGUAUGUGUUUGGGUGGCUGUUCUGGCUCCA_ACGAGUGGCUUAGGCUAUGUGCGUGCCUUUGGCAUAUUUGUGUGCCAACCA .......((((................((...........))..(((.(((((....)))))...)))))))((((((((....))))))))... (-11.43 = -12.73 + 1.31)

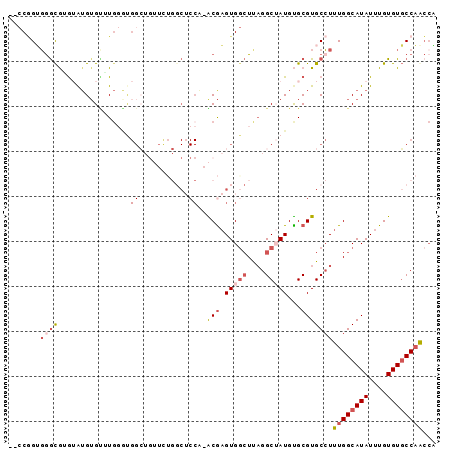
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:47 2006