
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
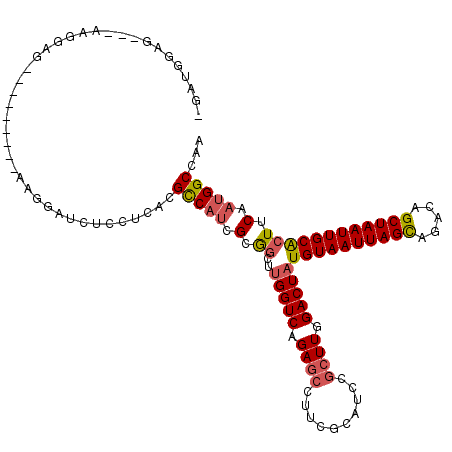
| Location | 7,655,515 – 7,655,629 |
| Length | 114 |
| Max. P | 0.868453 |

| Location | 7,655,515 – 7,655,611 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.72 |
| Mean single sequence MFE | -36.43 |
| Consensus MFE | -22.99 |
| Energy contribution | -23.43 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.868453 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7655515 96 + 27905053 -GAUGGAG---AAGGAGAA--------------------CGCCAUAGCGGCUUGGUCAGAGCGUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAA -..(((..---..((((..--------------------.(((.....))).(((((.(((((........))))).)))))((((((((((.....)))))))))))))).....))). ( -32.80) >DroSec_CAF1 10524 116 + 1 -GAUGGAG---AAGGAGAACGAGGGAAAGGAUCUCCUCACGCCACCGCGGCUUGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAA -..(((.(---(((.((..(((((((((((.(((..(((.(((.....))).)))..))).))))....))).))))..)).((((((((((.....)))))))))))))).....))). ( -38.10) >DroSim_CAF1 10203 116 + 1 -GAUGGAG---AAGGAGAACGAGGGAAAGGAUCUCCUCACGCCAUCGCGGCUUGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAA -(((((..---.((((((.(........)..))))))....)))))..(((((((((.((((..........)))).)))))((((((((((.....)))))))))).......)))).. ( -41.00) >DroEre_CAF1 10309 110 + 1 -GAUGGAG---AAGGAG------UCGAAGGAUCUGCUCACGCCAUCGUGGCUCGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAA -..(((((---..(((.------.((((((.((((..(..(((.....)))..)..)))).))))))..)))..........((((((((((.....)))))))))))))))........ ( -40.30) >DroYak_CAF1 10083 114 + 1 UGAUGGAGAUGAAGGAG------ACGAGGGAUCUCCUCACGCCAUCGUAGCUUGGUCAGAGCCCUCGCAUCCGCUUGGACUAUGUAAUUAGCAGGCAGCUAAUUGCACUUCAAUGGCCAA .(((((.(.(((.((((------((....).))))))))).)))))......((((((.((.((..((....))..)).)).((((((((((.....))))))))))......)))))). ( -40.40) >DroAna_CAF1 14965 87 + 1 ---UGGCG---CACUCG----------------UCCUCACGUCCUCGCGUCUUGGUCAGAGCC-----------UUGGACUAUGUAAUUAGUAGUCAGCUAAUUGCGCUUCAAUGGCCGA ---.((((---(...((----------------(....))).....)))))(((((((((((.-----------...((((((.......)))))).((.....))))))...))))))) ( -26.00) >consensus _GAUGGAG___AAGGAG_________AAGGAUCUCCUCACGCCAUCGCGGCUUGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAA ........................................(((((.(.((..(((((.((((..........)))).)))))((((((((((.....)))))))))))).).)))))... (-22.99 = -23.43 + 0.45)


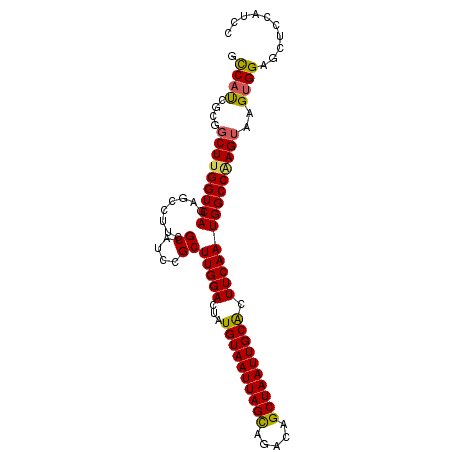
| Location | 7,655,531 – 7,655,629 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | forward |
| Mean pairwise identity | 88.64 |
| Mean single sequence MFE | -34.60 |
| Consensus MFE | -28.11 |
| Energy contribution | -28.03 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.60 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.848764 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7655531 98 + 27905053 GCCAUAGCGGCUUGGUCAGAGCGUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAAGUAAGUGGAGCUCCAUUC ((....)).((((((((((((((........))))).((...((((((((((.....))))))))))..))..))))))))).(((((....))))). ( -34.60) >DroSec_CAF1 10560 98 + 1 GCCACCGCGGCUUGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAAGUAAGUGGGGCUCCAUCC (((.((((.(((((((((.((((...((....))..)).)).((((((((((.....))))))))))......)))))))))..)))))))....... ( -39.90) >DroSim_CAF1 10239 98 + 1 GCCAUCGCGGCUUGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAAGUAAGUGGGGCUCCAUCC (((.((((.(((((((((.((((...((....))..)).)).((((((((((.....))))))))))......)))))))))..)))))))....... ( -36.90) >DroEre_CAF1 10339 98 + 1 GCCAUCGUGGCUCGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAAGUAAGUGGAGCUCCAUGC (((.....))).......((((.(((((....((((((.(((((((((((((.....))))))))))......)))))))))..)))))))))..... ( -33.70) >DroYak_CAF1 10117 98 + 1 GCCAUCGUAGCUUGGUCAGAGCCCUCGCAUCCGCUUGGACUAUGUAAUUAGCAGGCAGCUAAUUGCACUUCAAUGGCCAAGUAAGUGGAGUUCCAUCC .((((....(((((((((.((.((..((....))..)).)).((((((((((.....))))))))))......)))))))))..)))).......... ( -32.60) >DroAna_CAF1 14983 85 + 1 GUCCUCGCGUCUUGGUCAGAGCC-----------UUGGACUAUGUAAUUAGUAGUCAGCUAAUUGCGCUUCAAUGGCCGAGUAAGUGGAGAU--AUCC (((..(((..((((((((((((.-----------...((((((.......)))))).((.....))))))...))))))))...)))..)))--.... ( -29.90) >consensus GCCAUCGCGGCUUGGUCAGAGCCUUCGCAUCCGCUUGGACUAUGUAAUUAGCAGACAGCUAAUUGCACUUCAAUGGCCAAGUAAGUGGAGCUCCAUCC .((((....(((((((((........((....))(((((...((((((((((.....)))))))))).))))))))))))))..)))).......... (-28.11 = -28.03 + -0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:11 2006