
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 7,411,863 – 7,411,999 |
| Length | 136 |
| Max. P | 0.854104 |

| Location | 7,411,863 – 7,411,963 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 78.29 |
| Mean single sequence MFE | -34.47 |
| Consensus MFE | -24.88 |
| Energy contribution | -24.69 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.854104 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
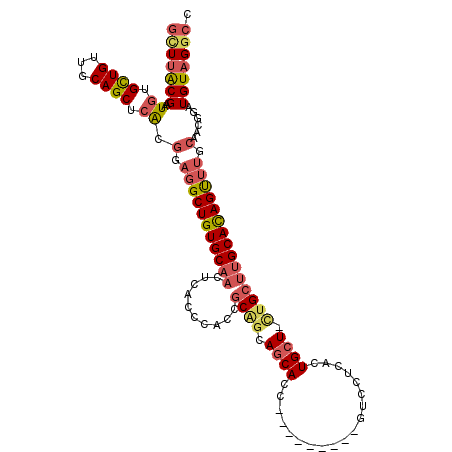
>3R_DroMel_CAF1 7411863 100 + 27905053 UAUCAUCUCGGCAU------CCCCCAGGCCUGGAUGGUGUUUACGAAUGUGCUGUUGCAGCUCGCGGAGGCUGUGCAACUCACCCACCGCAUCAGCACC---------GUCCGCA .........(((..------.......))).((((((((((.......(.((((...)))).)((((.((.((.......)).)).))))...))))))---------))))... ( -33.90) >DroSec_CAF1 3214 100 + 1 UAUCAUCUCGGCAU------CCCCCAGGCCCGGAUGGUGCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCACCGCAGCAGCAUC---------GUCCUCA ((((((((.(((..------.......))).)))))))).....((..((((((((((((((......))))(((.....))).....)))))))))).---------.)).... ( -33.50) >DroSim_CAF1 14039 100 + 1 UAUCAUCUCGGCAU------CCCCCAGGCCCGGAUGGUGCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCACCGCAGCAGCAUC---------GUCCUCA ((((((((.(((..------.......))).)))))))).....((..((((((((((((((......))))(((.....))).....)))))))))).---------.)).... ( -33.50) >DroEre_CAF1 7643 100 + 1 UAUCAUCUCGGCAU------CCCCCAUACCCGGAUGGUGCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCGCCGCGGCAGCACC---------GUCUUCA (((((((.(((...------.........)))))))))).....((..((((((((((.((.....(((.........)))....)).)))))))))).---------.)).... ( -32.20) >DroYak_CAF1 7635 100 + 1 UAUCAUCUCGGCAU------CUCCCAAACUCGGAUGGUGCUUACGAAUGUGCUGCUGCAGCUCACGGAGGCUGUGCAACUCAGCCACCGCUGCAGCACC---------GUCUUCA .........(((((------(..((......))..))))))...(((..((.(((((((((....((.(((((.......))))).))))))))))).)---------)..))). ( -40.60) >DroMoj_CAF1 11508 114 + 1 UUGCAUUUGCUUGUCCUGCGCCACCACAUCCGGAUGUACGUCGCGUAUGUGCUGUUGCAGUUCACGCAUACUGUGUAAUCCGGCAGCCGCCACACCAUGACUUACCCCAUCCG-G ((((((.(((.((..(((((.((.(((((.(((((....))).)).))))).)).)))))..)).)))....))))))...(((....)))......................-. ( -33.10) >consensus UAUCAUCUCGGCAU______CCCCCAGACCCGGAUGGUGCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCACCGCAGCAGCACC_________GUCCUCA ((((((((.(((...............))).)))))))).........((((((((((((((......))))(((.........))).))))))))))................. (-24.88 = -24.69 + -0.19)



| Location | 7,411,895 – 7,411,999 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 82.24 |
| Mean single sequence MFE | -38.63 |
| Consensus MFE | -25.70 |
| Energy contribution | -26.68 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.543499 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7411895 104 + 27905053 GUUUACGAAUGUGCUGUUGCAGCUCGCGGAGGCUGUGCAACUCACCCACCGCAUCAGCACC---------GUCCGCACUGCU-CUGCUUGCAUAGUUUGCAACGGAUGUAGGCC ......((..((((((.(((.(....)((.((.((.......)).)).))))).)))))).---------.)).((.(((((-(((.(((((.....))))))))).)))))). ( -35.20) >DroGri_CAF1 15239 113 + 1 CGUCGCGCAUAUGUUGCUGCAGCUCACACAUGCUGUGCAUCCCGGCUGCAACGGGACCAUGACCCAGUUCAUCCG-GCUGCUGUUGCUCGCACAGCUGGCAACGUAUGUAGGCG ((((..(((((((((((((((((........)))((((...((((.((.((((((.......))).))))).)))-)..((....))..)))).)).)))))))))))).)))) ( -46.80) >DroSec_CAF1 3246 104 + 1 GCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCACCGCAGCAGCAUC---------GUCCUCACUGCU-CUGCUUGCAUAGUUUGCAACGGAUGUAGGCC ((((((((..((((((((((((((......))))(((.....))).....)))))))))).---------.))........(-(((.(((((.....))))))))).)))))). ( -37.30) >DroSim_CAF1 14071 104 + 1 GCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCACCGCAGCAGCAUC---------GUCCUCACUGCU-CUGCUUGCAUAGUUUGCAACGGAUGUAGGCC ((((((((..((((((((((((((......))))(((.....))).....)))))))))).---------.))........(-(((.(((((.....))))))))).)))))). ( -37.30) >DroEre_CAF1 7675 104 + 1 GCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCGCCGCGGCAGCACC---------GUCUUCACUGCU-CUGCUUGCACAGUUUACAACGGAUGUAGGCC (((((((..((.((((...)))).))((.(((((((((((..........((((.((((..---------(....)..))))-)))))))))))))))....))..))))))). ( -34.80) >DroYak_CAF1 7667 104 + 1 GCUUACGAAUGUGCUGCUGCAGCUCACGGAGGCUGUGCAACUCAGCCACCGCUGCAGCACC---------GUCUUCACUGCU-CUGCUUGCACAGUUUACAUCGGAUGUAGGCC (((((((.......(((((((((....((.(((((.......))))).)))))))))))((---------(.....((((..-(.....)..))))......))).))))))). ( -40.40) >consensus GCUUACGAAUGUGCUGUUGCAGCUCACGGAGGCUGUGCAACUCACCCACCGCAGCAGCACC_________GUCCUCACUGCU_CUGCUUGCACAGUUUGCAACGGAUGUAGGCC (((((((..((.((((...)))).)).(.(((((((((((..........((((.((((...................)))).))))))))))))))).)......))))))). (-25.70 = -26.68 + 0.98)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:44:21 2006