
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
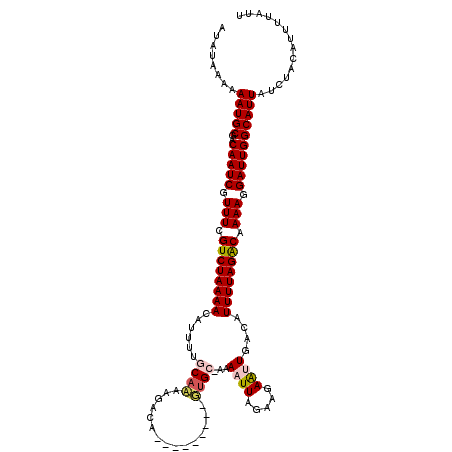
| Location | 7,073,835 – 7,073,944 |
| Length | 109 |
| Max. P | 0.979477 |

| Location | 7,073,835 – 7,073,944 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 81.50 |
| Mean single sequence MFE | -27.27 |
| Consensus MFE | -16.35 |
| Energy contribution | -16.58 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.77 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.961137 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
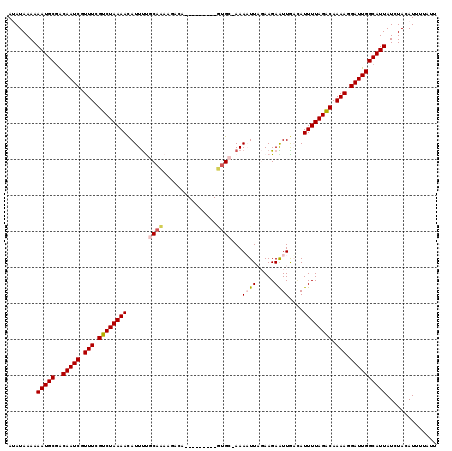
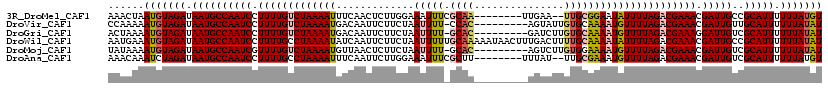
>3R_DroMel_CAF1 7073835 109 + 27905053 ACAUAAAAAAUGCGGCAAUCGUUUCGUCUAAAAUAUUCCGCAA--UUCAA--------UUGCGAAAUUUCCAAGAGUUGAAAUUUUAGACAAAAGGAUUGGCAUUAUCUACAUUAGUUU ........(((((..(((((.(((.(((((((((.((((((((--(...)--------))))).(((((....))))))))))))))))).))).)))))))))).............. ( -26.20) >DroVir_CAF1 21267 109 + 1 AUAUAAAAAAUGCAACAAUCGUUUCGUCUAAAACAUUUUGCACAAUACU---------GUGG-AAAAUUAGAAGAAUUGUCAUUUUAGACAAAAGGAUUGGCAUUAUCUACAUUUUUGG ........(((((..(((((.(((.((((((((.(((((.((((....)---------))).-))))).....((....)).)))))))).))).)))))))))).............. ( -24.80) >DroGri_CAF1 11167 109 + 1 AUAUAAAAAAUGCGACAAUCCUUUCGUCUAAAACAUUUUGCACAAGAUC---------GUGC-AAAAUUAGAAGAAUUGUCAUUUUAGACAAAAGGAUUGGCAUUAUCUACAUUUUAGU ........(((((..(((((((((.((((((((.(((((((((......---------))))-))))).....((....)).)))))))).)))))))))))))).............. ( -32.80) >DroWil_CAF1 4212 119 + 1 AUAUAAAAAAUGCGGCAAUCGUUUCGUCUAAAAUAUUUUGCAAAAGUCAAAGUUAUUUUUGCAAAAAUUAGAAGAAUUGAUAUUUUAGGCAAAAGGAUUGGCAUUAUCUACAUUUCAUU ........(((((..(((((.(((.((((((((((((((((((((((.......)))))))))).((((.....)))))))))))))))).))).)))))))))).............. ( -29.70) >DroMoj_CAF1 9847 109 + 1 AUAUAAAAAAUGCGACAAUCGUUUCGUCUAAAACAUUUUCCACAAGACU---------GUGC-AAAAUUAGAAGAGUUAACAUUUUAGACAAAACGAUUGGCAUUAUCUACAUUUUAUA .((((((((((((..(((((((((.((((((((.(((((.((((....)---------))).-))))).......(....).)))))))).)))))))))))))).......))))))) ( -25.61) >DroAna_CAF1 8744 109 + 1 ACAUAAAAAAUGCGACAAUCGUUUCGUCUAAAACAUUUCGCAA--AUAAA--------AAGCGAAAUUUCCAAGAAUUGAAAUUUUAGGCAAAAGGAUUGGCAUUAUCUAGAUUUGUUU ........(((((..(((((.(((.((((((((..((((((..--.....--------..))))))((((........)))))))))))).))).)))))))))).............. ( -24.50) >consensus AUAUAAAAAAUGCGACAAUCGUUUCGUCUAAAACAUUUUGCAAAAGACA_________GUGC_AAAAUUAGAAGAAUUGACAUUUUAGACAAAAGGAUUGGCAUUAUCUACAUUUUAUU ........(((((..(((((.(((.((((((((......((((...............))))...((((.....))))....)))))))).))).)))))))))).............. (-16.35 = -16.58 + 0.22)

| Location | 7,073,835 – 7,073,944 |
|---|---|
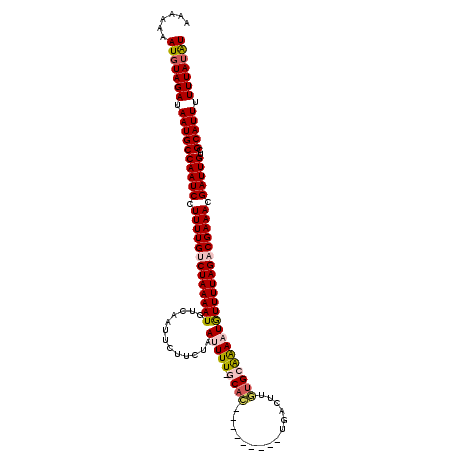
| Length | 109 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.50 |
| Mean single sequence MFE | -33.11 |
| Consensus MFE | -22.09 |
| Energy contribution | -22.45 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.13 |
| Mean z-score | -5.60 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979477 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7073835 109 - 27905053 AAACUAAUGUAGAUAAUGCCAAUCCUUUUGUCUAAAAUUUCAACUCUUGGAAAUUUCGCAA--------UUGAA--UUGCGGAAUAUUUUAGACGAAACGAUUGCCGCAUUUUUUAUGU ......(((((((.((((((((((.(((((((((((((((((.....)))))(((((((((--------(...)--))))))))).)))))))))))).)))))..))))).))))))) ( -35.20) >DroVir_CAF1 21267 109 - 1 CCAAAAAUGUAGAUAAUGCCAAUCCUUUUGUCUAAAAUGACAAUUCUUCUAAUUUU-CCAC---------AGUAUUGUGCAAAAUGUUUUAGACGAAACGAUUGUUGCAUUUUUUAUAU ......(((((((.((((((((((.(((((((((((((((....)).....(((((-.(((---------......))).)))))))))))))))))).)))))..))))).))))))) ( -29.60) >DroGri_CAF1 11167 109 - 1 ACUAAAAUGUAGAUAAUGCCAAUCCUUUUGUCUAAAAUGACAAUUCUUCUAAUUUU-GCAC---------GAUCUUGUGCAAAAUGUUUUAGACGAAAGGAUUGUCGCAUUUUUUAUAU ......(((((((.((((((((((((((((((((((((((....)).....(((((-((((---------(....)))))))))))))))))))))))))))))..))))).))))))) ( -42.40) >DroWil_CAF1 4212 119 - 1 AAUGAAAUGUAGAUAAUGCCAAUCCUUUUGCCUAAAAUAUCAAUUCUUCUAAUUUUUGCAAAAAUAACUUUGACUUUUGCAAAAUAUUUUAGACGAAACGAUUGCCGCAUUUUUUAUAU ......(((((((.((((((((((.(((((.(((((((((..............(((((((((...........)))))))))))))))))).))))).)))))..))))).))))))) ( -28.53) >DroMoj_CAF1 9847 109 - 1 UAUAAAAUGUAGAUAAUGCCAAUCGUUUUGUCUAAAAUGUUAACUCUUCUAAUUUU-GCAC---------AGUCUUGUGGAAAAUGUUUUAGACGAAACGAUUGUCGCAUUUUUUAUAU ......(((((((.((((((((((((((((((((((((.............(((((-.(((---------......))).))))))))))))))))))))))))..))))).))))))) ( -36.71) >DroAna_CAF1 8744 109 - 1 AAACAAAUCUAGAUAAUGCCAAUCCUUUUGCCUAAAAUUUCAAUUCUUGGAAAUUUCGCUU--------UUUAU--UUGCGAAAUGUUUUAGACGAAACGAUUGUCGCAUUUUUUAUGU .........((((.((((((((((.(((((.(((((((((((.....)))))(((((((..--------.....--..))))))).)))))).))))).)))))..))))).))))... ( -26.20) >consensus AAAAAAAUGUAGAUAAUGCCAAUCCUUUUGUCUAAAAUGUCAAUUCUUCUAAUUUU_GCAC_________UGACUUGUGCAAAAUGUUUUAGACGAAACGAUUGUCGCAUUUUUUAUAU ......(((((((.((((((((((.(((((((((((((.............(((((.((((...............)))))))))))))))))))))).)))))..))))).))))))) (-22.09 = -22.45 + 0.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:40:37 2006