
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 7,029,091 – 7,029,232 |
| Length | 141 |
| Max. P | 0.889165 |

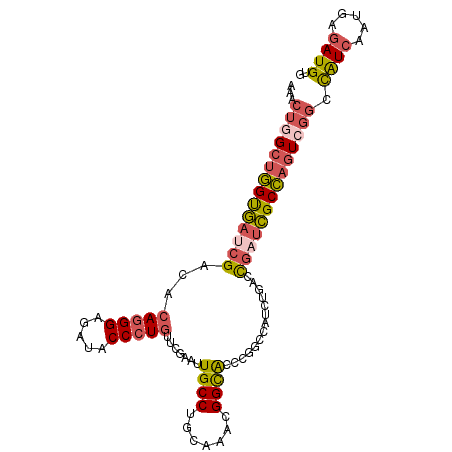
| Location | 7,029,091 – 7,029,192 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 83.30 |
| Mean single sequence MFE | -39.35 |
| Consensus MFE | -30.48 |
| Energy contribution | -31.27 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.889165 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7029091 101 + 27905053 AAACUGGCUGGUGAACGGCACAGGGAGAUACCCUGUUCGAAUUGCCUGAAAACGGCACCUGGCCAUCUGAUCGAUCGCCAGUCGGCCAUCAAUGAGAUGUG ...(((((((((((..(((((((((.....))))))......(((((....).))))....))).((.....))))))))))))).((((.....)))).. ( -38.00) >DroSec_CAF1 28560 101 + 1 AAACUGGCUGGUGAUCGACACAGGGAGAUACCCUGUUCGAAUUGCCUCCAAACGGCACCCGGCCAUCUGACCGAUCGCCAGUCGGCUAUCAAUGAGAUGUG ...((((((((((((((..((((((.....))))))(((...((((.......))))..))).........)))))))))))))).((((.....)))).. ( -39.30) >DroSim_CAF1 43263 101 + 1 AAACUGGCUGGUGAUCGACACAGGGAGAUACCCUGUUCGAAUUGCCUGCAAACGGCACCCGGCCAUCUGACCGAUCGCCAGUCGGCCAUCAAUGAGAUGUG ...((((((((((((((..((((((.....))))))(((.((.((((((.....)))...))).)).))).)))))))))))))).((((.....)))).. ( -42.30) >DroEre_CAF1 26798 101 + 1 GAGCUAGCUGGUGAUCGACACAGGGAUAUACCCUGCUCGAAUUGCCUGCAAACGGCAGCCGGUCAUCUGACCGAUCGCCAGUCGGCCAUCAAUGAGAUGUG ..((..((((((((((((..(((((.....))))).)))).(((((.......))))).(((((....))))).))))))))..))((((.....)))).. ( -43.80) >DroYak_CAF1 28680 101 + 1 AAACUGGCUGGCGAUCGGCACAGGGAUAUACCCUGCUCGAAUUGCCUGCAAACGGCACCCGAUCAUCUGACCGAUCGCCAGUCGGCCAUCAAUGAGAUGUG ...((((((((((((((((((((((.....))))).(((...((((.......))))..))).....)).))))))))))))))).((((.....)))).. ( -46.40) >DroAna_CAF1 31881 98 + 1 AAAAAAGAGAGUUCCGG---AAUGGACAAGCCCUGUCUGAAUUGCCUGCAACUGGUGCCGGAUCACCUGGCUGAUAGCUACUCGGCCGUGAAUGAGAUGUG ............(((((---...(((((.....))))).....(((.......))).)))))((((..((((((.......)))))))))).......... ( -26.30) >consensus AAACUGGCUGGUGAUCGACACAGGGAGAUACCCUGUUCGAAUUGCCUGCAAACGGCACCCGGCCAUCUGACCGAUCGCCAGUCGGCCAUCAAUGAGAUGUG ...((((((((((((((...(((((.....))))).......((((.......))))..............)))))))))))))).((((.....)))).. (-30.48 = -31.27 + 0.78)

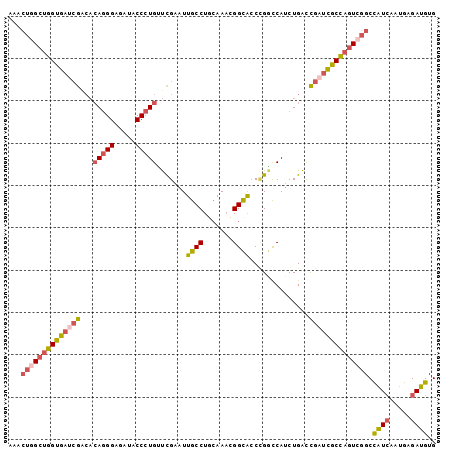
| Location | 7,029,091 – 7,029,192 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 83.30 |
| Mean single sequence MFE | -39.03 |
| Consensus MFE | -26.81 |
| Energy contribution | -27.65 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.732204 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7029091 101 - 27905053 CACAUCUCAUUGAUGGCCGACUGGCGAUCGAUCAGAUGGCCAGGUGCCGUUUUCAGGCAAUUCGAACAGGGUAUCUCCCUGUGCCGUUCACCAGCCAGUUU ...((((.((((((.(((....))).)))))).))))(((..((((((.......)))....((.((((((.....))))))..))...))).)))..... ( -37.90) >DroSec_CAF1 28560 101 - 1 CACAUCUCAUUGAUAGCCGACUGGCGAUCGGUCAGAUGGCCGGGUGCCGUUUGGAGGCAAUUCGAACAGGGUAUCUCCCUGUGUCGAUCACCAGCCAGUUU ...(((.....)))....(((((((...(((((....)))))((((((.......)))...((((((((((.....)))))).))))..))).))))))). ( -40.60) >DroSim_CAF1 43263 101 - 1 CACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGGCCGGGUGCCGUUUGCAGGCAAUUCGAACAGGGUAUCUCCCUGUGUCGAUCACCAGCCAGUUU ..((((.....))))...(((((((...(((((....)))))((((((.......)))...((((((((((.....)))))).))))..))).))))))). ( -44.00) >DroEre_CAF1 26798 101 - 1 CACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGACCGGCUGCCGUUUGCAGGCAAUUCGAGCAGGGUAUAUCCCUGUGUCGAUCACCAGCUAGCUC ..((((.....))))((...((((.((((((((....))))(((((((.......))))......((((((.....))))))))))))).))))...)).. ( -41.50) >DroYak_CAF1 28680 101 - 1 CACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGAUCGGGUGCCGUUUGCAGGCAAUUCGAGCAGGGUAUAUCCCUGUGCCGAUCGCCAGCCAGUUU ..((((.....))))((.(.((((((((((((.......(((((((((.......))).))))))((((((.....)))))))))))))))))).).)).. ( -47.90) >DroAna_CAF1 31881 98 - 1 CACAUCUCAUUCACGGCCGAGUAGCUAUCAGCCAGGUGAUCCGGCACCAGUUGCAGGCAAUUCAGACAGGGCUUGUCCAUU---CCGGAACUCUCUUUUUU .........(((.(((((..((((((....(((.((....)))))...)))))).)))..........(((....)))...---.)))))........... ( -22.30) >consensus CACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGACCGGGUGCCGUUUGCAGGCAAUUCGAACAGGGUAUCUCCCUGUGUCGAUCACCAGCCAGUUU ..(((((.((((((.(((....))).)))))).)))))....((((((.......))).......((((((.....)))))).......)))......... (-26.81 = -27.65 + 0.84)



| Location | 7,029,127 – 7,029,232 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 89.46 |
| Mean single sequence MFE | -43.59 |
| Consensus MFE | -35.80 |
| Energy contribution | -36.88 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.584625 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 7029127 105 - 27905053 ACCUUGUGGACGUCUGCAUUCGGGACCACACAGGCGCUGGCACAUCUCAUUGAUGGCCGACUGGCGAUCGAUCAGAUGGCCAGGUGCCGUUUUCAGGCAAUUCGA .((((((((...((((....)))).)))))..(((((((((.(((((.((((((.(((....))).)))))).))))))))).)))))......)))........ ( -44.70) >DroSec_CAF1 28596 105 - 1 ACCUUGUGGACGUCUGCAUUCGGGACCACACAGGCGCUGGCACAUCUCAUUGAUAGCCGACUGGCGAUCGGUCAGAUGGCCGGGUGCCGUUUGGAGGCAAUUCGA .((((((((...((((....)))).))))...(((((((((.(((((.((((((.(((....))).)))))).)))))))).)))))).....))))........ ( -42.80) >DroSim_CAF1 43299 105 - 1 ACCUUGUGGACGUCUGCAUUCGGGACCACACAGGCGCUGGCACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGGCCGGGUGCCGUUUGCAGGCAAUUCGA .((....))..(((((((..((....).....(((((((((.(((((.((((((.(((....))).)))))).)))))))).)))))))..)))))))....... ( -48.10) >DroEre_CAF1 26834 105 - 1 ACCUUGGGGACGUCUGCAUUCGGGACCACACAGACGCUGGCACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGACCGGCUGCCGUUUGCAGGCAAUUCGA .....(....)(((((((..(((.(((.....(((((((((.((((.....)))))))....)))).))((((....)))))).).)))..)))))))....... ( -43.80) >DroYak_CAF1 28716 105 - 1 ACCUUGGGGCCGUCUGCACUCGGGACCACACAGGCGCUGGCACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGAUCGGGUGCCGUUUGCAGGCAAUUCGA .((....))..(((((((..(((.(((...(((....((((.((((.....)))))))).))).((((((......))))))))).)))..)))))))....... ( -44.00) >DroAna_CAF1 31914 105 - 1 UCCUUGGGGCUGUCUGCACUGGGGACCACACAGGCGCUGGCACAUCUCAUUCACGGCCGAGUAGCUAUCAGCCAGGUGAUCCGGCACCAGUUGCAGGCAAUUCAG ....((((..((((((((((((.(.((.....)))(((((..(((((.......(((.((.......)).))))))))..))))).)))).)))))))).)))). ( -38.11) >consensus ACCUUGGGGACGUCUGCAUUCGGGACCACACAGGCGCUGGCACAUCUCAUUGAUGGCCGACUGGCGAUCGGUCAGAUGACCGGGUGCCGUUUGCAGGCAAUUCGA .((....))..(((((((..(((.(((...(((...)))...(((((.((((((.(((....))).)))))).)))))....))).)))..)))))))....... (-35.80 = -36.88 + 1.09)

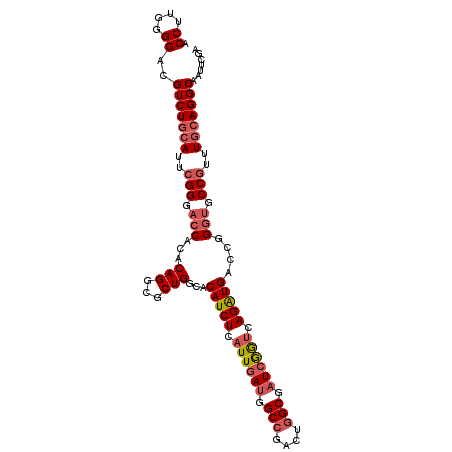

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:40:11 2006