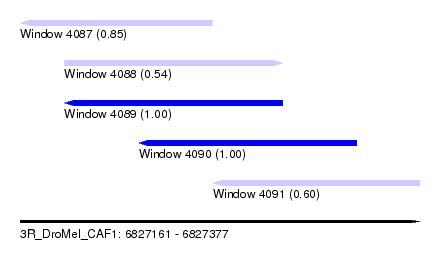
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 6,827,161 – 6,827,377 |
| Length | 216 |
| Max. P | 0.999686 |

| Location | 6,827,161 – 6,827,265 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.10 |
| Mean single sequence MFE | -27.30 |
| Consensus MFE | -25.50 |
| Energy contribution | -25.53 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.24 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.847882 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6827161 104 - 27905053 AGCGCCAUGAAGCCUUGGCUAUAUCUCUGCGCCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUUUUCUGUGCCAGCUCAACUGCUCAA---------------- .((((...(((((....)))...))...))))((((...(((((....)))))((((((((........)))))(((((.....))))).)))..)))).....---------------- ( -26.20) >DroSec_CAF1 86253 102 - 1 AGCGCCAUGAAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUUUUCUGUGCCAGCUCAACUGCUCAA---------------- ...((((........))))........--.((((((...(((((....)))))((((((((........)))))(((((.....))))).)))..))))))...---------------- ( -26.80) >DroSim_CAF1 92118 102 - 1 AGCGCCAUGAAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUUUUCUGUGCCAGCUCAACUGCUCAA---------------- ...((((........))))........--.((((((...(((((....)))))((((((((........)))))(((((.....))))).)))..))))))...---------------- ( -26.80) >DroEre_CAF1 71191 102 - 1 AGCGCCAUGGAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUUUUCUGUGCCAGCUCAACUGCUCAA---------------- ...((((.(....).))))........--.((((((...(((((....)))))((((((((........)))))(((((.....))))).)))..))))))...---------------- ( -27.50) >DroYak_CAF1 84501 118 - 1 AGCGCCAUGGAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUUUUCUGUGCCAGCUCAACUGCUCAACUGCUCAACUGGUUCA .(.((((..((((...((........)--)((((((...(((((....)))))((((((((........)))))(((((.....))))).)))..)))))).....))))...)))).). ( -31.80) >DroAna_CAF1 42751 100 - 1 AGCGCCAUGAAGCCUUGGCUAUAUCUC--UGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGUAUUUUCUGUGCCAGCUCAACUGCUU------------------ ...((((........))))........--.((((((...(((((....)))))((((((((........)))))(((((.....))))).)))..)))))).------------------ ( -24.70) >consensus AGCGCCAUGAAGCCUUGGCUAUAUCUC__CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUUUUCUGUGCCAGCUCAACUGCUCAA________________ ...((((........))))...........((((((...(((((....)))))((((((((........)))))(((((.....))))).)))..))))))................... (-25.50 = -25.53 + 0.03)



| Location | 6,827,185 – 6,827,303 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.27 |
| Mean single sequence MFE | -28.07 |
| Consensus MFE | -25.47 |
| Energy contribution | -25.63 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.04 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.535897 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6827185 118 + 27905053 AAUGCCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGGCGCAGAGAUAUAGCCAAGGCUUCAUGGCGCUUGGCGCAGCAG--AACUAAGCCAAAAAUGCCACAAAACAU ...(((...(((((((.(((.......)))))))))).........((((...........)))).)))....(((((.(((((..((...--..))..)))))...)))))........ ( -30.40) >DroSec_CAF1 86277 116 + 1 AAUGCCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGCCG--GAGAUAUAGCCAAGGCUUCAUGGCGCUUGGCGCAGCAG--AACUAAGCCAAAAAUGCCACAAAACAU ..........(((((..(((((.((((.((..((.(((........)))))--........(((((((((....))).))))))))))))(--........)...))))).))))).... ( -27.70) >DroSim_CAF1 92142 116 + 1 AAUGCCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGCCG--GAGAUAUAGCCAAGGCUUCAUGGCGCUUGGCGCAGCAG--AACUAAGCCAAAAAUGCCACAAAACAU ..........(((((..(((((.((((.((..((.(((........)))))--........(((((((((....))).))))))))))))(--........)...))))).))))).... ( -27.70) >DroEre_CAF1 71215 116 + 1 AAUGCCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGCCG--GAGAUAUAGCCAAGGCUCCAUGGCGCUUGGCGCAGCAG--AACUAAGCCAAAAAUGCCACAAAACAU ..........(((((..(((((.((((.((..((.(((........)))))--........(((((((((....))).))))))))))))(--........)...))))).))))).... ( -26.90) >DroYak_CAF1 84541 118 + 1 AAUGCCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGCCG--GAGAUAUAGCCAAGGCUCCAUGGCGCUUGGCGCAGCAGAGAACUAAGCCAAAAAUGCCACAAAACAU ..........(((((..(((((.((((.((..((.(((........)))))--........(((((((((....))).))))))))))))..(........)...))))).))))).... ( -26.90) >DroAna_CAF1 42773 116 + 1 AAUACCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGCCA--GAGAUAUAGCCAAGGCUUCAUGGCGCUUGGCGCUGCAG--AACUAAGCCAAAAAUGCCACAAAACAU ..........(((((..(((((.((((((...)))))).......((((.(--(.......(((((((((....))).)))))).))))))--............))))).))))).... ( -28.80) >consensus AAUGCCACAAUUUGUAUGCAUUAUGCUUGCUACGAGUAACAGAUACUGCCG__GAGAUAUAGCCAAGGCUUCAUGGCGCUUGGCGCAGCAG__AACUAAGCCAAAAAUGCCACAAAACAU ..........(((((..(((((.((((((...)))))).......(((((.....).....(((((((((....))).))))))...))))..............))))).))))).... (-25.47 = -25.63 + 0.17)


| Location | 6,827,185 – 6,827,303 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.27 |
| Mean single sequence MFE | -34.74 |
| Consensus MFE | -35.30 |
| Energy contribution | -35.27 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.75 |
| Structure conservation index | 1.02 |
| SVM decision value | 3.89 |
| SVM RNA-class probability | 0.999686 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6827185 118 - 27905053 AUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUCUGCGCCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUU ..((((..((((...((((((.(((..--...))).)))))).))))..))))....((((((...(((((..((((.....)))))))))...((.....))........))))))... ( -35.70) >DroSec_CAF1 86277 116 - 1 AUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUU ..((((..((((...((((((.(((..--...))).)))))).))))..))))....((((((....--..(((.(((((((((....)))).)).))).)))........))))))... ( -34.94) >DroSim_CAF1 92142 116 - 1 AUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUU ..((((..((((...((((((.(((..--...))).)))))).))))..))))....((((((....--..(((.(((((((((....)))).)).))).)))........))))))... ( -34.94) >DroEre_CAF1 71215 116 - 1 AUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGGAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUU ..((((..((((...((((((.(((..--...))).)))))).))))..))))....((((((....--..(((.(((((((((....)))).)).))).)))........))))))... ( -35.34) >DroYak_CAF1 84541 118 - 1 AUGUUUUGUGGCAUUUUUGGCUUAGUUCUCUGCUGCGCCAAGCGCCAUGGAGCCUUGGCUAUAUCUC--CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUU ..((((..((((...((((((.((((.....)))).)))))).))))..))))....((((((....--..(((.(((((((((....)))).)).))).)))........))))))... ( -36.44) >DroAna_CAF1 42773 116 - 1 AUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCAGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC--UGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGUAUU ....(((((((((((....((((....--((((...((((((.((......))))))))........--((((((...))))))...)))).))))..))))).)))))).......... ( -31.10) >consensus AUGUUUUGUGGCAUUUUUGGCUUAGUU__CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC__CGGCAGUAUCUGUUACUCGUAGCAAGCAUAAUGCAUACAAAUUGUGGCAUU ..((((..((((...((((((.((((.....)))).)))))).))))..))))....((((((........(((.(((((((((....)))).)).))).)))........))))))... (-35.30 = -35.27 + -0.03)

| Location | 6,827,225 – 6,827,343 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.27 |
| Mean single sequence MFE | -35.14 |
| Consensus MFE | -33.67 |
| Energy contribution | -33.78 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.80 |
| SVM RNA-class probability | 0.997129 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6827225 118 - 27905053 GUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUCUGCGCCAGUAUCU .((((.....(....)........(((.((.(((((((.((.((((..((((...((((((.(((..--...))).)))))).))))..)))).)).))))))).)))))))))...... ( -38.60) >DroSec_CAF1 86317 116 - 1 GUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC--CGGCAGUAUCU ...(((....(((((........)))))((.(((((((.((.((((..((((...((((((.(((..--...))).)))))).))))..)))).)).))))))).))--.)))....... ( -34.60) >DroSim_CAF1 92182 116 - 1 GUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC--CGGCAGUAUCU ...(((....(((((........)))))((.(((((((.((.((((..((((...((((((.(((..--...))).)))))).))))..)))).)).))))))).))--.)))....... ( -34.60) >DroEre_CAF1 71255 116 - 1 GUGGCUAAGAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCAAGCGCCAUGGAGCCUUGGCUAUAUCUC--CGGCAGUAUCU (((((((((.(((((........)))))..............((((..((((...((((((.(((..--...))).)))))).))))..))))))))))))).....--........... ( -35.00) >DroYak_CAF1 84581 118 - 1 GUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUUCUCUGCUGCGCCAAGCGCCAUGGAGCCUUGGCUAUAUCUC--CGGCAGUAUCU ...(((....(((((........)))))((.(((((((.((.((((..((((...((((((.((((.....)))).)))))).))))..)))).)).))))))).))--.)))....... ( -36.10) >DroAna_CAF1 42813 116 - 1 GUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCAGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC--UGGCAGUAUCU ...((((...(((((........)))))((.(((((((.((.((((..((((...((((((......--.......)))))).))))..)))).)).))))))).))--))))....... ( -31.92) >consensus GUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU__CUGCUGCGCCAAGCGCCAUGAAGCCUUGGCUAUAUCUC__CGGCAGUAUCU ...(((....(((((........)))))((.(((((((.((.((((..((((...((((((.((((.....)))).)))))).))))..)))).)).))))))).))...)))....... (-33.67 = -33.78 + 0.11)

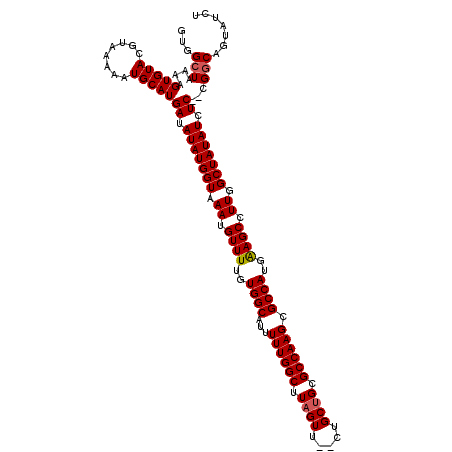

| Location | 6,827,265 – 6,827,377 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.07 |
| Mean single sequence MFE | -32.47 |
| Consensus MFE | -26.37 |
| Energy contribution | -26.90 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.598092 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6827265 112 - 27905053 GGGAGAGAUAGUGGCGCAGGAAGUGAGACA------GCAAGUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCA ...........(((((((((((.((((.((------((.....)))...........((((((((((..(.((((.....)))).)..).)))))))))))))).))--)..)))))))) ( -32.60) >DroSec_CAF1 86355 112 - 1 GGGAGAGAUAGUGGCGCAGGGAGUGAGACA------GCAAGUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCA ...........(((((((((((((......------))....((((((((((((.(((....)))((..(.((((.....)))).)..))))))))))))))....)--)).)))))))) ( -32.60) >DroSim_CAF1 92220 112 - 1 GAGAGAGAUAGUGGCGCAGGGAGUGAGACA------GCAAGUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCA ...........(((((((((((((......------))....((((((((((((.(((....)))((..(.((((.....)))).)..))))))))))))))....)--)).)))))))) ( -32.60) >DroEre_CAF1 71293 118 - 1 GGGAGAGAUAGUGGCCCAGAGAGUGAGACAGCAAGAGCAAGUGGCUAAGAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCUGCGCCA (((...........)))...(.(((...(((((.((((....((((((((((((.(((....)))((..(.((((.....)))).)..))))))))))))))..)))--)))))))))). ( -32.10) >DroYak_CAF1 84619 120 - 1 GGGAGAGAUAGUGGCGCAGAGAGUGAGACGGCAACAGCAAGUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUUCUCUGCUGCGCCA ...........((((((((((((...((((....).......((((((((((((.(((....)))((..(.((((.....)))).)..))))))))))))))..))))))).)))))))) ( -38.30) >DroAna_CAF1 42851 113 - 1 GGGAGAUAGAAAGACGCACACACU-CGCUUGCA----AAAGUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU--CUGCAGCGCCA (((((.................))-)(((.(((----.((.(((((((((((((.(((....)))((..(.((((.....)))).)..))))))))))))).)).))--.)))))).)). ( -26.63) >consensus GGGAGAGAUAGUGGCGCAGAGAGUGAGACA______GCAAGUGGCUAAAAGUGUACGUAAAAAUGCAUGAUAUAUGGUAAAUGUUUUGUGGCAUUUUUGGCUUAGUU__CUGCUGCGCCA ............(((((((.......................((((((((((((.(((....)))((..(.((((.....)))).)..))))))))))))))(((....)))))))))). (-26.37 = -26.90 + 0.53)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:37:54 2006