
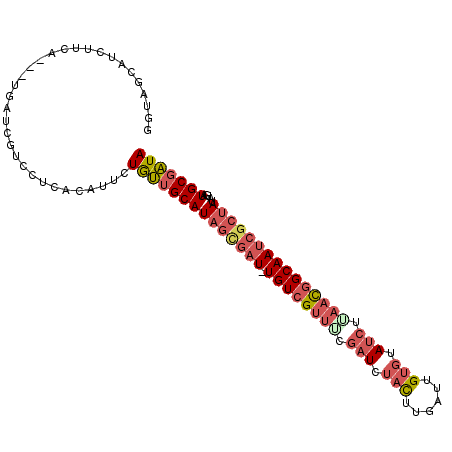
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 6,541,234 – 6,541,335 |
| Length | 101 |
| Max. P | 0.984931 |

| Location | 6,541,234 – 6,541,335 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 79.41 |
| Mean single sequence MFE | -32.73 |
| Consensus MFE | -20.63 |
| Energy contribution | -22.05 |
| Covariance contribution | 1.42 |
| Combinations/Pair | 1.25 |
| Mean z-score | -3.55 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.99 |
| SVM RNA-class probability | 0.984931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
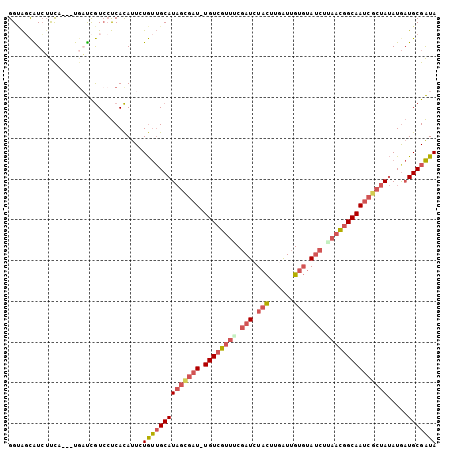
>3R_DroMel_CAF1 6541234 101 + 27905053 GGUAGCAUCUUCA---UGAUCGUCCUCACAUUCUGUUGCAUAGCGAU-UGUCGUUUCGAUCUACUUGAUUGUGUAUCUAAACGGCAAUCGCUAUAUAAUGCGAUA ((..(.(((....---.))))..))........((((((((((((((-((((((((.(((.(((......))).))).))))))))))))))).....))))))) ( -33.90) >DroSec_CAF1 18130 101 + 1 GGUAGCAUCUUUA---UGAUCGUCCUCACAUUCUGUUGCAUAGCGAU-UGUCGUUUCGAUCUACUUGGUUGUGUAUCUUAACGGCAAUCGCUAUAUGAUGCGAUA ....(((((....---(((......)))...........((((((((-(((((((..(((.(((......))).)))..)))))))))))))))..))))).... ( -34.40) >DroSim_CAF1 17563 101 + 1 GGCAGCAUCUUUA---UGAUCGUCCUCACAUUCUGUUGCAUAGCGAU-UGUCGUUUCGAUCUACUUGGUUGUGUAUCUUAACGGCAAUCGCUAUAUGAUGCGAUA .((((((......---(((......))).....))))))((((((((-(((((((..(((.(((......))).)))..)))))))))))))))........... ( -34.50) >DroEre_CAF1 20356 101 + 1 GGCACCAUCUUCA---UCCUCGUCCUCACAUUGUGUUGCAUAGUGAU-UGUCGUUUCGAUCUACUUGAUUGUGUAUCUGAACGGCAAUCGCUAUAUGAUGCGAUA .............---.............((((((((..((((((((-((((((((.(((.(((......))).))).))))))))))))))))..)))))))). ( -34.20) >DroYak_CAF1 18555 102 + 1 GGUAGCGUCGUCA---UGCUCGUCCUCACAUUGUGUUGCAUAGUGAUUUGUCGUUUCGAUAUACUUGAUUGUGUAUCUGAACGGCAAUCGCUAUAUGAUGCGAUA ((.(((((....)---))))...))....((((((((..(((((((((.(((((((.(((((((......))))))).))))))))))))))))..)))))))). ( -39.40) >DroAna_CAF1 23853 101 + 1 GGAGCUGUGUCCGUAUUGACUAUAACCAUACUGUACAGCAU---CAU-UGUGGGCACCAUUUUUAUUAUCGUCUACCUUAAUGGCAACAUCUACAUGCUGCGGUA .((((((((.(.((((.(........))))).))))))).)---)..-.((((((...............))))))....((.(((.(((....))).))).)). ( -19.96) >consensus GGUAGCAUCUUCA___UGAUCGUCCUCACAUUCUGUUGCAUAGCGAU_UGUCGUUUCGAUCUACUUGAUUGUGUAUCUUAACGGCAAUCGCUAUAUGAUGCGAUA .................................((((((((((((((.((((((((.(((.(((......))).))).))))))))))))))).....))))))) (-20.63 = -22.05 + 1.42)

| Location | 6,541,234 – 6,541,335 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 79.41 |
| Mean single sequence MFE | -27.40 |
| Consensus MFE | -10.64 |
| Energy contribution | -11.20 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.39 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.661707 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
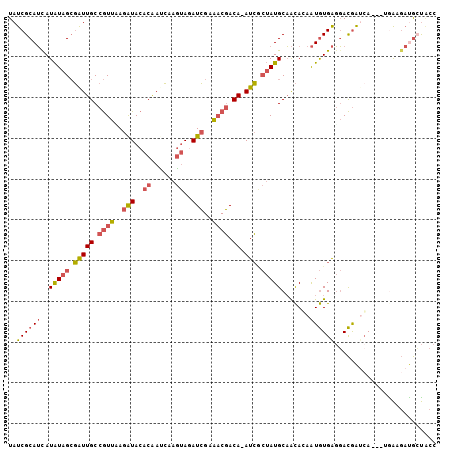
>3R_DroMel_CAF1 6541234 101 - 27905053 UAUCGCAUUAUAUAGCGAUUGCCGUUUAGAUACACAAUCAAGUAGAUCGAAACGACA-AUCGCUAUGCAACAGAAUGUGAGGACGAUCA---UGAAGAUGCUACC ....(((((.(((((((((((.(((((.(((..((......))..))).))))).))-))))))))).........((((......)))---)...))))).... ( -30.40) >DroSec_CAF1 18130 101 - 1 UAUCGCAUCAUAUAGCGAUUGCCGUUAAGAUACACAACCAAGUAGAUCGAAACGACA-AUCGCUAUGCAACAGAAUGUGAGGACGAUCA---UAAAGAUGCUACC ....(((((.(((((((((((.((((..(((..((......))..)))..)))).))-)))))))))........(((((......)))---))..))))).... ( -33.30) >DroSim_CAF1 17563 101 - 1 UAUCGCAUCAUAUAGCGAUUGCCGUUAAGAUACACAACCAAGUAGAUCGAAACGACA-AUCGCUAUGCAACAGAAUGUGAGGACGAUCA---UAAAGAUGCUGCC ....(((((.(((((((((((.((((..(((..((......))..)))..)))).))-)))))))))........(((((......)))---))..))))).... ( -33.30) >DroEre_CAF1 20356 101 - 1 UAUCGCAUCAUAUAGCGAUUGCCGUUCAGAUACACAAUCAAGUAGAUCGAAACGACA-AUCACUAUGCAACACAAUGUGAGGACGAGGA---UGAAGAUGGUGCC ....(((((((((((.(((((.((((..(((..((......))..)))..)))).))-))).))))....(((...)))..........---.....))))))). ( -25.30) >DroYak_CAF1 18555 102 - 1 UAUCGCAUCAUAUAGCGAUUGCCGUUCAGAUACACAAUCAAGUAUAUCGAAACGACAAAUCACUAUGCAACACAAUGUGAGGACGAGCA---UGACGACGCUACC ..((((((..(((((.((((..((((..((((.((......)).))))..))))...)))).))))).......))))))((...(((.---.......))).)) ( -21.10) >DroAna_CAF1 23853 101 - 1 UACCGCAGCAUGUAGAUGUUGCCAUUAAGGUAGACGAUAAUAAAAAUGGUGCCCACA-AUG---AUGCUGUACAGUAUGGUUAUAGUCAAUACGGACACAGCUCC (((((((((((....)))))))......))))...............((.((....(-((.---(((((....))))).)))...(((......)))...)).)) ( -21.00) >consensus UAUCGCAUCAUAUAGCGAUUGCCGUUAAGAUACACAAUCAAGUAGAUCGAAACGACA_AUCGCUAUGCAACACAAUGUGAGGACGAUCA___UGAAGAUGCUACC ..((((((..(((((.(((((.((((..(((..((......))..)))..)))).)).))).))))).......))))))......................... (-10.64 = -11.20 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:31:57 2006