
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 6,021,553 – 6,021,698 |
| Length | 145 |
| Max. P | 0.880736 |

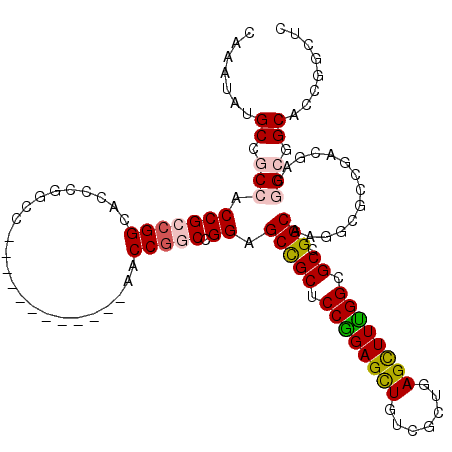
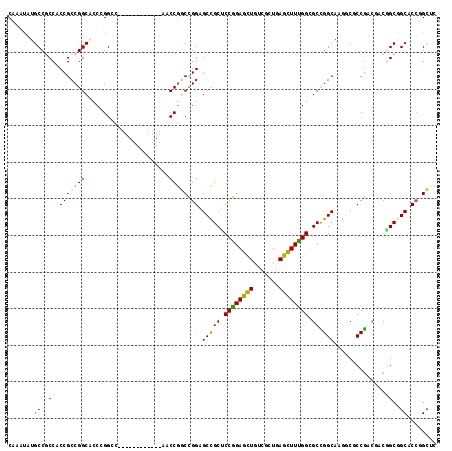
| Location | 6,021,553 – 6,021,658 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 75.46 |
| Mean single sequence MFE | -52.93 |
| Consensus MFE | -29.65 |
| Energy contribution | -29.93 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.880736 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
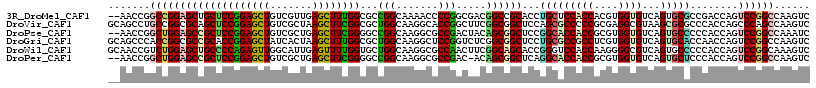
>3R_DroMel_CAF1 6021553 105 + 27905053 CGAAUAUGCCGCCACCGCCGGCUCCCGGCC------------AACCGGCCGGAGCUGCUCCGGAGCUGUCGUUGAGCUUUGGCGCCGGCAAAACCCCGGCGACGGCCGCACCUGCUC ......(((.(((..((((((...((((((------------....)))))).(((((.((((((((.......)))))))).).))))......))))))..))).)))....... ( -52.20) >DroPse_CAF1 31528 105 + 1 CCAAUAUGCCACCUCCGCCGGCACCGGGGC------------AACCGGCUGGAGCCGCUCCGGAGCUGUCGCUGAGCUUCGGGGCCGGCAAGGCGCCGACUACAGCGGCUCCGGCAC ......((((......((((....).((..------------..))))).(((((((((((((((((.......))))))))((.((((.....)))).))..))))))))))))). ( -54.70) >DroGri_CAF1 39605 117 + 1 CAAAUAUGCCGCCACCGCCGGCACCCGGUCAGGUGCCGCCGCAGCCCACCGGCGCCGCACCGGAGCUAUCACUAAGCUUUGGCGCUGGCAAGGCUCCGGUCUCGGCGGCUCCUGCGC .......((((((......((((((......))))))((((.((((..(((((((((.....(((((.......))))))))))))))...)))).))))...))))))........ ( -59.00) >DroSim_CAF1 33444 105 + 1 CGAACAUGCCGCCACCGCCGGCUCCCGGCC------------AACCGGCCGGAGCUGCUCCGGAGCUGUCGUUGAGCUUUGGCGCCGGCAAAACCCCGGCGACGGCUGCACCUGCUC ......(((.(((..((((((...((((((------------....)))))).(((((.((((((((.......)))))))).).))))......))))))..))).)))....... ( -52.20) >DroWil_CAF1 35870 117 + 1 CAAAUAUGCCGCCACCGCCGGCACCAGGCCAAUUGCCACCGCAACCGUCUGGAGCUGCCCCAGAGUUGGCAUUGAGUUUUGGUGCUGGCAAGGCGCCAACUUCGGCAGCACCGGGUC .......((((((...(((((((((((((((((.((((.((....))(((((.......)))))..)))))))).).))))))))))))..)))(((......))).......))). ( -47.70) >DroPer_CAF1 31524 104 + 1 CCAAUAUGCCACCUCCGCCGGCACCGGGGC------------AACCGGCUGGAGCCGCUCCGGAGCUGUCGCUGAGCUUCGGGGCCGGCAAGGCGCCGAC-ACAGCGGCUCAGGCAC ......((((.........(((.(((((((------------....(((....)))))))))).)))(((((((.(..((((.(((.....))).)))))-.)))))))...)))). ( -51.80) >consensus CAAAUAUGCCGCCACCGCCGGCACCCGGCC____________AACCGGCCGGAGCCGCUCCGGAGCUGUCGCUGAGCUUUGGCGCCGGCAAGGCGCCGACGACGGCGGCACCGGCUC .......((.(((.(((((((.......................))))).)).(((((.((((((((.......)))))))).)).)))..............))).))........ (-29.65 = -29.93 + 0.28)

| Location | 6,021,553 – 6,021,658 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 75.46 |
| Mean single sequence MFE | -57.25 |
| Consensus MFE | -33.91 |
| Energy contribution | -34.42 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.694087 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6021553 105 - 27905053 GAGCAGGUGCGGCCGUCGCCGGGGUUUUGCCGGCGCCAAAGCUCAACGACAGCUCCGGAGCAGCUCCGGCCGGUU------------GGCCGGGAGCCGGCGGUGGCGGCAUAUUCG (((...((((.(((..((((((((((((((....).)))))))).......(((((((((...))))((((....------------)))).))))))))))..))).)))).))). ( -55.70) >DroPse_CAF1 31528 105 - 1 GUGCCGGAGCCGCUGUAGUCGGCGCCUUGCCGGCCCCGAAGCUCAGCGACAGCUCCGGAGCGGCUCCAGCCGGUU------------GCCCCGGUGCCGGCGGAGGUGGCAUAUUGG ((((((((((((((...((((((.....)))))).(((.((((.......)))).)))))))))))).((((((.------------((....))))))))......)))))..... ( -57.80) >DroGri_CAF1 39605 117 - 1 GCGCAGGAGCCGCCGAGACCGGAGCCUUGCCAGCGCCAAAGCUUAGUGAUAGCUCCGGUGCGGCGCCGGUGGGCUGCGGCGGCACCUGACCGGGUGCCGGCGGUGGCGGCAUAUUUG ..((....(((((((...(((.(((((((((.(((((..((((.......))))..))))))))).....))))).)))((((((((....)))))))).))))))).))....... ( -64.60) >DroSim_CAF1 33444 105 - 1 GAGCAGGUGCAGCCGUCGCCGGGGUUUUGCCGGCGCCAAAGCUCAACGACAGCUCCGGAGCAGCUCCGGCCGGUU------------GGCCGGGAGCCGGCGGUGGCGGCAUGUUCG (((((..(((.(((..((((((((((((((....).)))))))).......(((((((((...))))((((....------------)))).))))))))))..))).)))))))). ( -56.30) >DroWil_CAF1 35870 117 - 1 GACCCGGUGCUGCCGAAGUUGGCGCCUUGCCAGCACCAAAACUCAAUGCCAACUCUGGGGCAGCUCCAGACGGUUGCGGUGGCAAUUGGCCUGGUGCCGGCGGUGGCGGCAUAUUUG ......(((((((((..(((((((((..(((((((((....((((..(....)..))))((((((......))))))))))....)))))..)))))))))..)))))))))..... ( -56.50) >DroPer_CAF1 31524 104 - 1 GUGCCUGAGCCGCUGU-GUCGGCGCCUUGCCGGCCCCGAAGCUCAGCGACAGCUCCGGAGCGGCUCCAGCCGGUU------------GCCCCGGUGCCGGCGGAGGUGGCAUAUUGG (((((.((((((((..-((((((.....)))))).(((.((((.......)))).)))))))))))..((((((.------------((....))))))))......)))))..... ( -52.60) >consensus GAGCAGGAGCCGCCGUAGCCGGCGCCUUGCCGGCGCCAAAGCUCAACGACAGCUCCGGAGCAGCUCCAGCCGGUU____________GGCCGGGUGCCGGCGGUGGCGGCAUAUUCG ........((((((((.(((((.(((..((((((.....((((.......)))).(((((...)))))))))))..................))).))))).))))))))....... (-33.91 = -34.42 + 0.50)



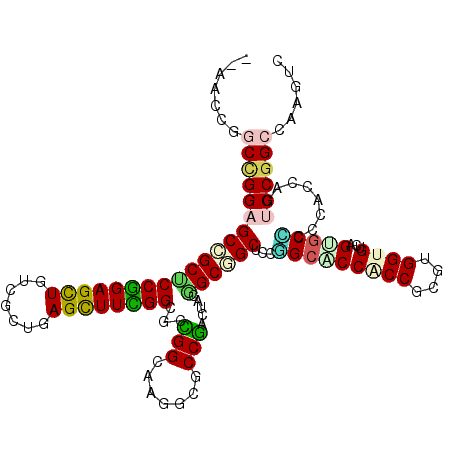
| Location | 6,021,583 – 6,021,698 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.67 |
| Mean single sequence MFE | -56.73 |
| Consensus MFE | -35.34 |
| Energy contribution | -35.32 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.830788 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 6021583 115 + 27905053 --AACCGGCCGGAGCUGCUCCGGAGCUGUCGUUGAGCUUUGGCGCCGGCAAAACCCCGGCGACGGCCGCACCUGCUCCACCACGUGGUGUCAGUGCGCCGACCAGUCCGGCCAAGUC --....(((((((.(((..((((((((.......))))))))((((((.......)))))).((((.((((..((.((((...)))).))..))))))))..))))))))))..... ( -58.80) >DroVir_CAF1 35012 117 + 1 GCAGCCUGCCGGCGCAGCUCCGGAGCUGUCGCUAAGCUUCGGCGCUGGCAAGGCACCGGCUUCGGCGGCUCCAGCGCCCCCGCGAGGCGUAAGCGCGCCCACCAGCCCAGCCAAGUC ((.((((((((((((.....(((((((.......))))))))))))))).))))...((((..(((((((...(((((.......))))).))).))))....))))..))...... ( -57.10) >DroPse_CAF1 31558 115 + 1 --AACCGGCUGGAGCCGCUCCGGAGCUGUCGCUGAGCUUCGGGGCCGGCAAGGCGCCGACUACAGCGGCUCCGGCACCACCGCGUGGUGUCAGUGCCCCCACCAGUCCGGCCAAAUC --..((((((((.((((((((((((((.......)))))))))).))))..(((((((.(....)))))...((((((((...))))))))...)))....)))).))))....... ( -62.40) >DroGri_CAF1 39645 117 + 1 GCAGCCCACCGGCGCCGCACCGGAGCUAUCACUAAGCUUUGGCGCUGGCAAGGCUCCGGUCUCGGCGGCUCCUGCGCCGCCUCGUGGUGUCAGUGCACCAACCAGUCCGGCCAAGUC ...(((....)))(((((.((((((((.......)))))))).)).)))..((((((((.((.((((((......))))))...((((((....))))))...)).))))...)))) ( -53.80) >DroWil_CAF1 35910 117 + 1 GCAACCGUCUGGAGCUGCCCCAGAGUUGGCAUUGAGUUUUGGUGCUGGCAAGGCGCCAACUUCGGCAGCACCGGGUCCACCAAGGGGCGUCAGUGCCCCCACCAGUCCGGCAAAGUC ....(((.((((.((((((...((((((((((..(...)..))(((.....))))))))))).))))))...((.....))..((((((....))))))..))))..)))....... ( -48.10) >DroPer_CAF1 31554 114 + 1 --AACCGGCUGGAGCCGCUCCGGAGCUGUCGCUGAGCUUCGGGGCCGGCAAGGCGCCGAC-ACAGCGGCUCAGGCACCACCGCGUGGUGUCAGUGCUCCCACCAGUCCGGCCAAGUC --..((((((((.((((((((((((((.......)))))))))).))))..(((((((..-....))))...((((((((...))))))))...)))....)))).))))....... ( -60.20) >consensus __AACCGGCCGGAGCCGCUCCGGAGCUGUCGCUGAGCUUCGGCGCCGGCAAGGCGCCGACUACGGCGGCUCCGGCACCACCGCGUGGUGUCAGUGCCCCCACCAGUCCGGCCAAGUC .......((((((((((((((((((((.......))))))))...(((.......))).....))))))...(((((((((....))))...)))))........))))))...... (-35.34 = -35.32 + -0.02)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:27:41 2006