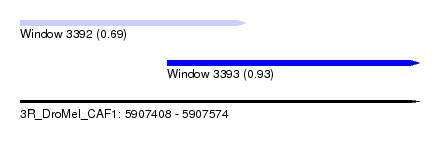
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 5,907,408 – 5,907,574 |
| Length | 166 |
| Max. P | 0.931226 |

| Location | 5,907,408 – 5,907,502 |
|---|---|
| Length | 94 |
| Sequences | 4 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 89.52 |
| Mean single sequence MFE | -29.30 |
| Consensus MFE | -21.62 |
| Energy contribution | -21.38 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.32 |
| SVM RNA-class probability | 0.687200 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
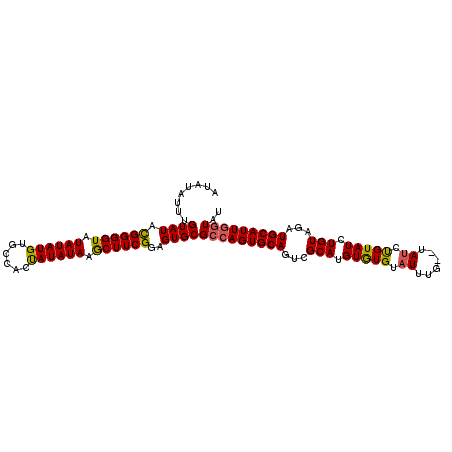
>3R_DroMel_CAF1 5907408 94 + 27905053 UGGCGGUAACUGUGUUUGUGGAAGUGCAAGCCAUGGCUGUGUGUGUAUCGGGCGAGUGGUUAUAUAUUUGUAUAUGGGGUAUAU------AUGUACCACC .((.((((.((((((((((......))))).)))))..(((((((..(((.(((((((......)))))))...)))..)))))------)).)))).)) ( -24.10) >DroSec_CAF1 21176 94 + 1 UGGCGGUGACUGUGCUUGUGGAAGUGCAAGCCAUGGCUGUGUGUGUAUCGGGCGAGUGGUUAUAUAGUUGUAUACGGGGUAUAU------AUGUGACUCU .(((.(((.(((..(((....)))..).)).))).)))(((((((..(((.((((.((......)).))))...)))..)))))------))........ ( -24.90) >DroSim_CAF1 9221 94 + 1 UGGCGGUGACUGUGCUUGUGGAAGUGCAAGCCAUGGCUGUGUGUGUAUCGGGCGAGUGGUUAUAUAUUUGUAUACGGGGUAUAU------AUGUGCCACU (((((....)((..(((....)))..)).))))((((((((((((..(((.(((((((......)))))))...)))..)))))------))).)))).. ( -32.80) >DroYak_CAF1 11248 100 + 1 UGGUGGUGACUGUGCCUGUGGAAGUGCAAGCCAUGGCUGUGUGAGUAUCGCGCGAGCAGUUAUAUAUUUGUAUAUGGGGUAUAUACCAGUAUGUGCCACU .(((((((((((((((.((((.........)))))))((((((.....)))))).))))))(((((((.(((((((...))))))).))))))))))))) ( -35.40) >consensus UGGCGGUGACUGUGCUUGUGGAAGUGCAAGCCAUGGCUGUGUGUGUAUCGGGCGAGUGGUUAUAUAUUUGUAUACGGGGUAUAU______AUGUGCCACU .....((((((.((((((.....(..((.((....))))..)......))))))...))))))............((((((((((....)))))))).)) (-21.62 = -21.38 + -0.25)



| Location | 5,907,469 – 5,907,574 |
|---|---|
| Length | 105 |
| Sequences | 4 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 90.00 |
| Mean single sequence MFE | -31.77 |
| Consensus MFE | -24.55 |
| Energy contribution | -24.30 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.21 |
| SVM RNA-class probability | 0.931226 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5907469 105 + 27905053 AUAUAUUUGUAUAUGGGGUAUAUAUGUACCACCAUAUAAGCUUCGGAGUGCGCCAGUGCAGUCGCAUGUGUGUAUUUG--UAUCCGUAUCUGUAGCUGCAUUGGUAU .........((((((((((((....))))).))))))).((........))(((((((((((..((.(((((.((...--.)).))))).))..))))))))))).. ( -37.20) >DroSec_CAF1 21237 107 + 1 AUAUAGUUGUAUACGGGGUAUAUAUGUGACUCUAUAUAAGCUUCGGAGUGCGCCAGUGCAGUCGCAUGUGUGUAUUUGUGUAUCUGUAUCUGUAGAUGCAUUGGUAU .............((((((.((((((......)))))).))))))((((((((..((((....))))..))))))))(((((((((......)))))))))...... ( -30.30) >DroSim_CAF1 9282 107 + 1 AUAUAUUUGUAUACGGGGUAUAUAUGUGCCACUAUAUAAACUUCGGAGUGCGCCAGUGCAGUCGCAUGUAUGUAUUUGUGUAUCUGUAUCUGUAGAUGCAUUGGUAU ((((((.(((...((((((.((((((......)))))).))))))((.((((....)))).))))).))))))((..(((((((((......)))))))))..)).. ( -31.50) >DroEre_CAF1 11006 105 + 1 AUGUAUUUGUAUAUGGGGUAUAUAUGUGCCACUAUAUAAGCUUCGGUGUGCGCCAGUGCAAUCGCAUGUGUGUAUUUG--UAGCUGUAUCUGUGUCUGCAUUCAUAU (((((....((((((((((((....))))).)))))))((((.(((.((((((..((((....))))..)))))))))--.))))...........)))))...... ( -28.10) >consensus AUAUAUUUGUAUACGGGGUAUAUAUGUGCCACUAUAUAAGCUUCGGAGUGCGCCAGUGCAGUCGCAUGUGUGUAUUUG__UAUCUGUAUCUGUAGAUGCAUUGGUAU ........((((.((((((.((((((......)))))).))))))..))))(((((((((...(((.(((((.((......)).))))).)))...))))))))).. (-24.55 = -24.30 + -0.25)

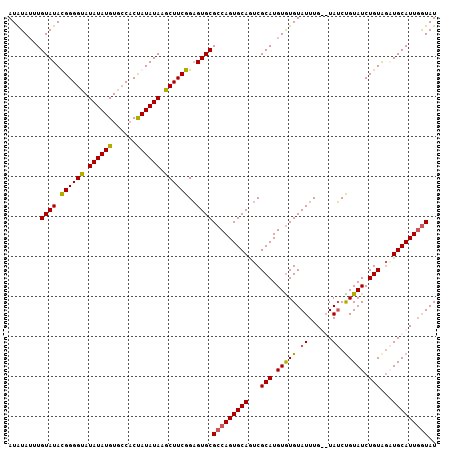
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:26:45 2006