
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 5,643,464 – 5,643,664 |
| Length | 200 |
| Max. P | 0.980431 |

| Location | 5,643,464 – 5,643,584 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.50 |
| Mean single sequence MFE | -35.28 |
| Consensus MFE | -31.80 |
| Energy contribution | -31.88 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.86 |
| SVM RNA-class probability | 0.980431 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5643464 120 - 27905053 GGUGUACAUAGGCUUCUUGUACAUGUAUUAUUAUGGAUACGGCGGCCAUGGAUUCUUCUUGAUCACCUUCAACGUGGUGGUGCACACCAUGAUGUACACCUACUACUACCAGUCGUCCCU (((((((((.((((.(((((.((((......)))).))).)).))))...........((((......))))(((((((.....)))))))))))))))).................... ( -35.10) >DroSec_CAF1 17238 120 - 1 GGUGUACGUAAGCUUCUUGUACCUGUAUUAUUAUGGAUACGGCGGCCAUGGGCUCUUCUUGGUCACCAUCAACGUGGUGGUGCACACCAUGAUGUACACCUACUACUACCAAUCCUCCCU (((((((((.(((((..((..((((((((......))))))).)..)).))))).....(((((((((((.....)))))))...))))..))))))))).................... ( -38.50) >DroSim_CAF1 18350 120 - 1 GGUGUACGCAGGCUUCUUGUACCUGUAUUAUUAUGGAUACGGCGGCCAUGGGCUCUUCUUGGUCACCUUCAACGUGGUGGUGCACACCAUGAUGUACACCUACUACUACCAGUCCUCCCU ((((((((((((.........))))).......((((...((.(((((.(((....)))))))).)))))).(((((((.....)))))))..))))))).................... ( -37.20) >DroEre_CAF1 17674 120 - 1 GGUGUACGUGGGCUUUUUGUACAUGUAUUAUUAUGGAUACGGCGGCCAUGGGUUCUUCUUGGUCACUUUCAACGUGGUUGUGCACACCAUGAUGUACACCUACUACUAUCAGUCAUCCCU ((((((.(((((.....((((((((((((......)))))((.(((((.(((....)))))))).)).....((((((.(....)))))))))))))))))))))).))).......... ( -32.50) >DroYak_CAF1 18235 120 - 1 GGUGUACAUAGGCUUUUUGUACAUGUACUACUAUGGAUACGGCGGCCAUGGCUUCUUCUUGGUCACUUUCAACGUGGUUGUGCACACCAUGAUGUACACCUACUACUAUCAGUCUUCCCU (((((((((..((.....))...(((((.((((((.....((.(((((.((.....)).))))).)).....)))))).))))).......))))))))).................... ( -33.10) >consensus GGUGUACGUAGGCUUCUUGUACAUGUAUUAUUAUGGAUACGGCGGCCAUGGGUUCUUCUUGGUCACCUUCAACGUGGUGGUGCACACCAUGAUGUACACCUACUACUACCAGUCCUCCCU (((((((((..((.....))...((((((((((((.....((.(((((.(((....)))))))).)).....)))))))))))).......))))))))).................... (-31.80 = -31.88 + 0.08)

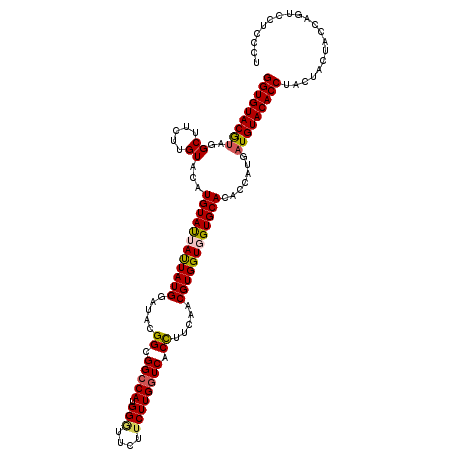

| Location | 5,643,504 – 5,643,624 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.67 |
| Mean single sequence MFE | -35.86 |
| Consensus MFE | -31.82 |
| Energy contribution | -31.78 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.25 |
| SVM RNA-class probability | 0.936192 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5643504 120 - 27905053 GCCAGAUAUCGUUCCUGCACGUUUUCCACCACGUAUACAUGGUGUACAUAGGCUUCUUGUACAUGUAUUAUUAUGGAUACGGCGGCCAUGGAUUCUUCUUGAUCACCUUCAACGUGGUGG ((..((......))..)).......(((((((((...(((((((((((.((....))))))).((((((......))))))...))))))((((......)))).......))))))))) ( -35.20) >DroSec_CAF1 17278 120 - 1 GCCAGAUAUCCUUCCUGCACGUCUUCCACCACGUAUACAUGGUGUACGUAAGCUUCUUGUACCUGUAUUAUUAUGGAUACGGCGGCCAUGGGCUCUUCUUGGUCACCAUCAACGUGGUGG ((..((......))..)).......(((((((((....((((((..((..(((((..((..((((((((......))))))).)..)).))))).....))..))))))..))))))))) ( -37.30) >DroSim_CAF1 18390 120 - 1 GCCAGAUAUCCUUCCUGCACGUCUUCCACCACGUAUACAUGGUGUACGCAGGCUUCUUGUACCUGUAUUAUUAUGGAUACGGCGGCCAUGGGCUCUUCUUGGUCACCUUCAACGUGGUGG ((..((......))..)).......(((((((((((.(((((((...(((((.........)))))..))))))).))))((.(((((.(((....)))))))).))......))))))) ( -38.60) >DroEre_CAF1 17714 120 - 1 GACAGAUAUCCUUUCUGCACGUUUUCCACCACGUAUACAUGGUGUACGUGGGCUUUUUGUACAUGUAUUAUUAUGGAUACGGCGGCCAUGGGUUCUUCUUGGUCACUUUCAACGUGGUUG ..((((.......))))((((((........(((((.(((((((((((((..(.....)..)))))).))))))).)))))..(((((.(((....))))))))......)))))).... ( -35.70) >DroYak_CAF1 18275 120 - 1 GUCAGAUAUCCUUCCUGCACGUUUUCCACCAUGUGUACAUGGUGUACAUAGGCUUUUUGUACAUGUACUACUAUGGAUACGGCGGCCAUGGCUUCUUCUUGGUCACUUUCAACGUGGUUG (((((.........))).)).......(((((((...((((((((((((..((.....))..)))))).))))))((...((.(((((.((.....)).))))).)).)).))))))).. ( -32.50) >consensus GCCAGAUAUCCUUCCUGCACGUUUUCCACCACGUAUACAUGGUGUACGUAGGCUUCUUGUACAUGUAUUAUUAUGGAUACGGCGGCCAUGGGUUCUUCUUGGUCACCUUCAACGUGGUGG (.(((.........)))).......(((((((((((.((((((((((((..((.....))..)))))).)))))).))))((.(((((.(((....)))))))).))......))))))) (-31.82 = -31.78 + -0.04)



| Location | 5,643,544 – 5,643,664 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.75 |
| Mean single sequence MFE | -30.48 |
| Consensus MFE | -26.76 |
| Energy contribution | -26.32 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.82 |
| SVM RNA-class probability | 0.978702 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5643544 120 + 27905053 GUAUCCAUAAUAAUACAUGUACAAGAAGCCUAUGUACACCAUGUAUACGUGGUGGAAAACGUGCAGGAACGAUAUCUGGCGAUACUUCUUUCUGAAUAUAAAGAACACAGUCUCCAGCAG ...(((............(((((((....)).)))))((((((....)))))))))...(((......)))....((((.(((..((((((........))))))....))).))))... ( -26.40) >DroSec_CAF1 17318 120 + 1 GUAUCCAUAAUAAUACAGGUACAAGAAGCUUACGUACACCAUGUAUACGUGGUGGAAGACGUGCAGGAAGGAUAUCUGGCGAUACUUCUUUCUGAGUAUAAAGAACACCGUCUCCAGCAG (((((............))))).....(((......(((((((....)))))))(.(((((..(((((((((((((....)))).))))))))).((.......))..))))).)))).. ( -32.40) >DroSim_CAF1 18430 120 + 1 GUAUCCAUAAUAAUACAGGUACAAGAAGCCUGCGUACACCAUGUAUACGUGGUGGAAGACGUGCAGGAAGGAUAUCUGGCGAUACUUCUUUCUGAGUAUAAAGAACACAGUCUCCAGCAG (((((............))))).......((((...(((((((....)))))))(.((((((((((((((((((((....)))).)))))))))...........))).)))).).)))) ( -34.10) >DroEre_CAF1 17754 120 + 1 GUAUCCAUAAUAAUACAUGUACAAAAAGCCCACGUACACCAUGUAUACGUGGUGGAAAACGUGCAGAAAGGAUAUCUGUCGAUACUUCUUUCUCAGUAUAAAGAAUACAGUCUCCAGCAG ((((........)))).(((((........(((((.(((((((....)))))))....))))).((((((((((((....)))).))))))))..))))).................... ( -29.20) >DroYak_CAF1 18315 120 + 1 GUAUCCAUAGUAGUACAUGUACAAAAAGCCUAUGUACACCAUGUACACAUGGUGGAAAACGUGCAGGAAGGAUAUCUGACGAUACUUCUUUCUCAGUAUAAAGAAUACAGUCUCCAGCAG ...(((......(((((((..(.....)..)))))))((((((....))))))))).....(((((((((((((((....)))).))))))))..((((.....))))........))). ( -30.30) >consensus GUAUCCAUAAUAAUACAUGUACAAGAAGCCUACGUACACCAUGUAUACGUGGUGGAAAACGUGCAGGAAGGAUAUCUGGCGAUACUUCUUUCUGAGUAUAAAGAACACAGUCUCCAGCAG ((((........))))...........((.(((((.(((((((....)))))))....)))))(((((((((((((....)))).)))))))))......................)).. (-26.76 = -26.32 + -0.44)

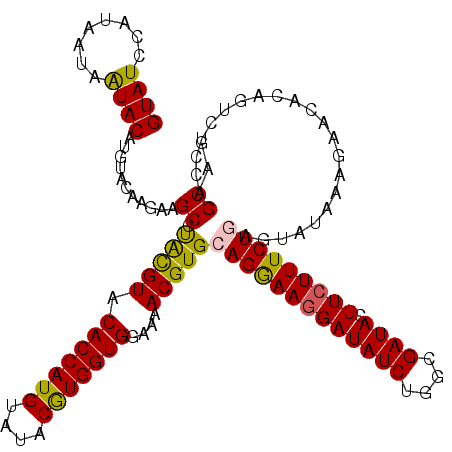

| Location | 5,643,544 – 5,643,664 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.75 |
| Mean single sequence MFE | -30.68 |
| Consensus MFE | -25.84 |
| Energy contribution | -25.24 |
| Covariance contribution | -0.60 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.810035 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5643544 120 - 27905053 CUGCUGGAGACUGUGUUCUUUAUAUUCAGAAAGAAGUAUCGCCAGAUAUCGUUCCUGCACGUUUUCCACCACGUAUACAUGGUGUACAUAGGCUUCUUGUACAUGUAUUAUUAUGGAUAC .((.((((((.((((((((((........))))).(((((....))))).......))))).)))))).)).((((.((((((((((((..((.....))..))))).))))))).)))) ( -29.40) >DroSec_CAF1 17318 120 - 1 CUGCUGGAGACGGUGUUCUUUAUACUCAGAAAGAAGUAUCGCCAGAUAUCCUUCCUGCACGUCUUCCACCACGUAUACAUGGUGUACGUAAGCUUCUUGUACCUGUAUUAUUAUGGAUAC ..((((((((((((((.....)))).(((.(((..(((((....))))).))).)))..)))))))....((((((((...)))))))).))).....(((.(((((....))))).))) ( -29.70) >DroSim_CAF1 18430 120 - 1 CUGCUGGAGACUGUGUUCUUUAUACUCAGAAAGAAGUAUCGCCAGAUAUCCUUCCUGCACGUCUUCCACCACGUAUACAUGGUGUACGCAGGCUUCUUGUACCUGUAUUAUUAUGGAUAC .((.(((((((.(((((((((........))))).(((((....))))).......))))))).)))).)).((((.(((((((...(((((.........)))))..))))))).)))) ( -29.50) >DroEre_CAF1 17754 120 - 1 CUGCUGGAGACUGUAUUCUUUAUACUGAGAAAGAAGUAUCGACAGAUAUCCUUUCUGCACGUUUUCCACCACGUAUACAUGGUGUACGUGGGCUUUUUGUACAUGUAUUAUUAUGGAUAC ..((((((((..((((.....))))((((((((..(((((....))))).)))))).))...))))))((((((((((...))))))))))))........((((......))))..... ( -32.40) >DroYak_CAF1 18315 120 - 1 CUGCUGGAGACUGUAUUCUUUAUACUGAGAAAGAAGUAUCGUCAGAUAUCCUUCCUGCACGUUUUCCACCAUGUGUACAUGGUGUACAUAGGCUUUUUGUACAUGUACUACUAUGGAUAC .((.((((((((((((((((......))))).((((((((....))))..)))).)))).).)))))).)).((((.((((((((((((..((.....))..)))))).)))))).)))) ( -32.40) >consensus CUGCUGGAGACUGUGUUCUUUAUACUCAGAAAGAAGUAUCGCCAGAUAUCCUUCCUGCACGUUUUCCACCACGUAUACAUGGUGUACGUAGGCUUCUUGUACAUGUAUUAUUAUGGAUAC .((.(((((((((((((((((........))))))(((((....)))))......)))).))).)))).)).((((.((((((((((((..((.....))..)))))).)))))).)))) (-25.84 = -25.24 + -0.60)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:24:40 2006