
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 5,484,845 – 5,485,005 |
| Length | 160 |
| Max. P | 0.999987 |

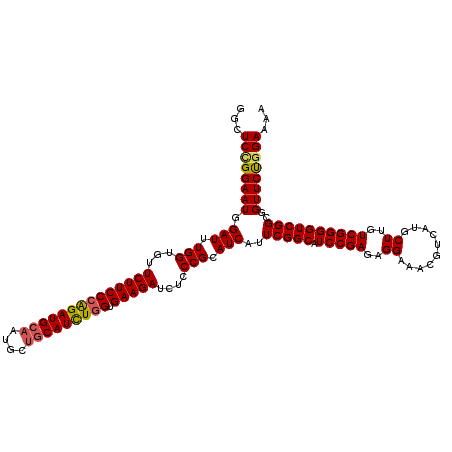
| Location | 5,484,845 – 5,484,965 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.78 |
| Mean single sequence MFE | -48.52 |
| Consensus MFE | -44.06 |
| Energy contribution | -43.95 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.91 |
| SVM decision value | 3.02 |
| SVM RNA-class probability | 0.998143 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5484845 120 - 27905053 GGGUCGGGAAUGGAUUUGGUGUUCUUCCCGGAUGCAAUGCUGCAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGGGGAUCCGUUAUGCUUGUCGGGGUCGGCGGUUCUGGAAAA ...((.(((((.(.........((((((((((((((....))))))))).)))))..(((((((.(((.(((.((((....)))))))..)))..)).)))))....).))))).))... ( -47.40) >DroVir_CAF1 23319 120 - 1 GGCUCCGGAAUGGAUUUGGUGUUCUUCCCAGAUGCAAUGCUGCAUUUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGAGGCAACGUCAUGCUGGUCGGGGUCGGAGGUUCUGGAAAA ...((((((((.(((.(((.(.((((((((((((((....))))))))).)))))...)))).))).((((((.(((((..((((......))))..))))))))))).))))))))... ( -53.40) >DroGri_CAF1 16904 120 - 1 GGAUCCGGAAUGGAUUUGGUUUUCUUCCCAGAUGCCAUGCUACAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGAGGCAACGUCAUGCUGGUCGGGGUCGGCGGUUCCGGAAAA ...((((((((.(((.(((...((((((((((((........))))))).)))))....))).)))..(((((.(((((..((((......))))..))))))))))..))))))))... ( -51.60) >DroWil_CAF1 16929 120 - 1 GGCUCUGGAAUGGAUUUGGUUUUCUUCCCAGAUGCAAUGUUGCAUUUAGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGAGGAUCGGUCAUGCUUGUCGGGGUCGGCGGUUCUGGAAAA (((((((((((((....((...((((((.(((((((....))))))).).)))))...))....))))))(((((((((.....))))..)))))...)))))))............... ( -38.70) >DroMoj_CAF1 23221 120 - 1 GGCUCCGGAAUGGAUUUGGUUUUCUUCCCAGAUGCAAUGUUGCAUUUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGAGGCAACGUUAUGCUAGUCGGGGUCGGAGGUUCUGGAAAA ...((((((((.(((.(((...((((((((((((((....))))))))).)))))....))).))).((((((.(((((..((((......))))..))))))))))).))))))))... ( -51.90) >DroPer_CAF1 12992 120 - 1 GGAUCUGGAAUGGAUUUGGUGUUCUUCCCGGAUGCAAUGCUGCAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGAGGAUCGGUCAUGCUUGUCGGGGUCGGCGGUUCUGGAAAG ...((..((((.(...((((((((((((((((((((....))))))))).)))))......)))))).(((((.(((((.(((..........))).))))))))))).))))..))... ( -48.10) >consensus GGCUCCGGAAUGGAUUUGGUGUUCUUCCCAGAUGCAAUGCUGCAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAGAGGAAACGUCAUGCUUGUCGGGGUCGGCGGUUCUGGAAAA ...((((((((.(((.(((...((((((((((((((....))))))))).)))))....))).)))..(((((.(((((..((..........))..))))))))))..))))))))... (-44.06 = -43.95 + -0.11)


| Location | 5,484,885 – 5,485,005 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.67 |
| Mean single sequence MFE | -41.50 |
| Consensus MFE | -34.86 |
| Energy contribution | -35.75 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.19 |
| Structure conservation index | 0.84 |
| SVM decision value | 2.18 |
| SVM RNA-class probability | 0.989698 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5484885 120 + 27905053 CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACCAGAUGCAGCAUUGCAUCCGGGAAGAACACCAAAUCCAUUCCCGACCCUCGCACCAUUUCAUUAAACUGGGCGAGGAACAUGACGAGA ((((.(((..(((((((...((.(.(((((.((.(((((......))))).))))))).).))..)).))))).....((((((.(((.((.....)).)))))))))..)))..)))). ( -43.50) >DroVir_CAF1 23359 120 + 1 CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACCAAAUGCAGCAUUGCAUCUGGGAAGAACACCAAAUCCAUUCCGGAGCCUCGCACCAUCUCAUUGAAUUGGGCGAGAAACAUAACGAGA ((((....(((((((((...((.(.(((((.(((.((((......)))).)))))))).).))..)).)))).)))...(((((.(((..((...))..))))))))........)))). ( -41.40) >DroGri_CAF1 16944 120 + 1 CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACCAGAUGUAGCAUGGCAUCUGGGAAGAAAACCAAAUCCAUUCCGGAUCCUCGCACCAUCUCAUUGAAUUGGGCGAGGAACAUAACGAGA ((((....(((((((((...((...(((((.((((((((......)))))))))))))...))..)).)))).))).(((((((.(((..((...))..))))))))))......)))). ( -49.20) >DroWil_CAF1 16969 120 + 1 CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACUAAAUGCAACAUUGCAUCUGGGAAGAAAACCAAAUCCAUUCCAGAGCCCCUCACCAUUUCAUUAAAUUGGGCAAGGAACAUGACGAGA ((((.((((((((((((...((...(((((.(((.(((((....))))).))))))))...))..)).)))))....((((...................))))..))..)))..)))). ( -32.11) >DroMoj_CAF1 23261 120 + 1 CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACCAAAUGCAACAUUGCAUCUGGGAAGAAAACCAAAUCCAUUCCGGAGCCUCGCACCAUCUCAUUGAAUUGGGCGAGAAACAUAACGAGA ((((....(((((((((...((...(((((.(((.(((((....))))).))))))))...))..)).)))).)))...(((((.(((..((...))..))))))))........)))). ( -40.60) >DroAna_CAF1 16916 120 + 1 CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACCAGAUGCAGCAUUGCAUCCGGGAAGAAAACCAAAUCCAUUCCCGACCCUCGCACCAUCUCGUUAAACUGGGCGAGGAACAUGACGAGA ((((.(((..(((((((...((...(((((.((.(((((......))))).)))))))...))..)).))))).....((((((.(((...........)))))))))..)))..)))). ( -42.20) >consensus CUCGGAUGCCGAAUGAUGCGGGAGAUCUUCACCAAAUGCAGCAUUGCAUCUGGGAAGAAAACCAAAUCCAUUCCGGAGCCUCGCACCAUCUCAUUAAAUUGGGCGAGGAACAUAACGAGA ((((.(((..(((((((...((...(((((.(((.((((......)))).))))))))...))..)).))))).....((((((.(((...........)))))))))..)))..)))). (-34.86 = -35.75 + 0.89)



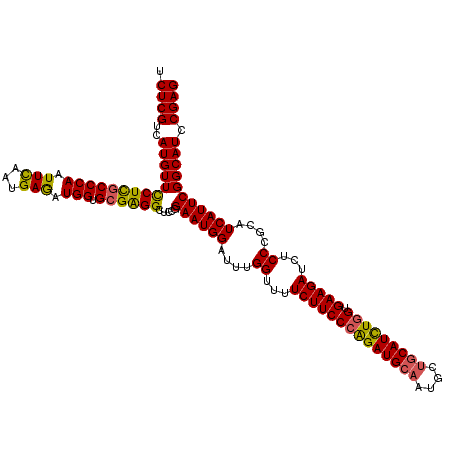
| Location | 5,484,885 – 5,485,005 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.67 |
| Mean single sequence MFE | -45.32 |
| Consensus MFE | -44.76 |
| Energy contribution | -43.98 |
| Covariance contribution | -0.78 |
| Combinations/Pair | 1.16 |
| Mean z-score | -3.09 |
| Structure conservation index | 0.99 |
| SVM decision value | 5.45 |
| SVM RNA-class probability | 0.999987 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5484885 120 - 27905053 UCUCGUCAUGUUCCUCGCCCAGUUUAAUGAAAUGGUGCGAGGGUCGGGAAUGGAUUUGGUGUUCUUCCCGGAUGCAAUGCUGCAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG .((((..((((((((((((((...........))).)))))).....((((((....((.(.((((((((((((((....))))))))).))))).).))....))))))))))).)))) ( -49.60) >DroVir_CAF1 23359 120 - 1 UCUCGUUAUGUUUCUCGCCCAAUUCAAUGAGAUGGUGCGAGGCUCCGGAAUGGAUUUGGUGUUCUUCCCAGAUGCAAUGCUGCAUUUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG .((((..((((((((((((((.(((...))).))).)))))).....((((((....((.(.((((((((((((((....))))))))).))))).).))....))))))))))).)))) ( -45.50) >DroGri_CAF1 16944 120 - 1 UCUCGUUAUGUUCCUCGCCCAAUUCAAUGAGAUGGUGCGAGGAUCCGGAAUGGAUUUGGUUUUCUUCCCAGAUGCCAUGCUACAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG .((((..(((.((((((((((.(((...))).))).))))))).(((((...(((.(((...((((((((((((........))))))).)))))....))).))).)))))))).)))) ( -46.60) >DroWil_CAF1 16969 120 - 1 UCUCGUCAUGUUCCUUGCCCAAUUUAAUGAAAUGGUGAGGGGCUCUGGAAUGGAUUUGGUUUUCUUCCCAGAUGCAAUGUUGCAUUUAGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG ....(((..((((((..((..((((....))))))..))))))....((((((....((...((((((.(((((((....))))))).).)))))...))....)))))))))....... ( -36.50) >DroMoj_CAF1 23261 120 - 1 UCUCGUUAUGUUUCUCGCCCAAUUCAAUGAGAUGGUGCGAGGCUCCGGAAUGGAUUUGGUUUUCUUCCCAGAUGCAAUGUUGCAUUUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG .((((..((((((((((((((.(((...))).))).)))))).....((((((....((...((((((((((((((....))))))))).)))))...))....))))))))))).)))) ( -44.70) >DroAna_CAF1 16916 120 - 1 UCUCGUCAUGUUCCUCGCCCAGUUUAACGAGAUGGUGCGAGGGUCGGGAAUGGAUUUGGUUUUCUUCCCGGAUGCAAUGCUGCAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG .((((..((((((((((((((...........))).)))))).....((((((....((...((((((((((((((....))))))))).)))))...))....))))))))))).)))) ( -49.00) >consensus UCUCGUCAUGUUCCUCGCCCAAUUCAAUGAGAUGGUGCGAGGCUCCGGAAUGGAUUUGGUUUUCUUCCCAGAUGCAAUGCUGCAUCUGGUGAAGAUCUCCCGCAUCAUUCGGCAUCCGAG .((((..((((((((((((((.(((...))).))).)))))).....((((((....((...((((((((((((((....))))))))).)))))...))....))))))))))).)))) (-44.76 = -43.98 + -0.78)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:23:29 2006