
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 5,271,860 – 5,272,004 |
| Length | 144 |
| Max. P | 0.983748 |

| Location | 5,271,860 – 5,271,974 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.82 |
| Mean single sequence MFE | -58.28 |
| Consensus MFE | -39.14 |
| Energy contribution | -38.32 |
| Covariance contribution | -0.83 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.977406 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5271860 114 + 27905053 GACAGCCGCUGCCGCAUCUGCGGCCACCGCCAGCAAUGUGUUCGGUGCCGUUGGCCAGGUAAAUCUGGAGCAAUAUGCCGCAGCGGUGGCCUCUGCAGCUGCCGCCAGCCGGCC ....((((((((.(((.(((((((((((((((....))....(((((..((((.(((((....)))))..)))).)))))..)))))))))...)))).))).).))).)))). ( -54.20) >DroVir_CAF1 45097 111 + 1 AACUGCGGCCGCGGCUACGGCGGCCACAGCGAGCAAUGUUUUUGGCGCUGUCGGUCAGGUUAACUUUGAG---UAUGCUGCCGCGGCGGCUGCUGCUGCGGCGGCCAGUCGGCC ......((((((.((....))((((((((((..(((.....))).)))))).))))..............---...(((((((((((((...)))))))))))))..).))))) ( -55.40) >DroPse_CAF1 48681 111 + 1 GACGGCGGCCGCCA---AUGCGGCCAACGCCAGCAAUGUGUUUGGCGCUGUCGGCCAGGUGAAUCUGGAGCAGUAUGCCGCUGCAGCAGCGGCUGCCGCAGCCGCCAGCCGGCC ((((((((((((..---..))))))...((((((.....).)))))))))))((((.(((....(((.(((.(....).))).)))..(((((((...)))))))..))))))) ( -59.90) >DroWil_CAF1 56150 114 + 1 GACAGCUGCUGCUGCAACAGCGGCCACCGCUGGCAAUGUCUUCGGUGCUGUUGGCCAGGUCAAUUUGGAGCAAUACGCUGCCGCGGCAGCUGCUGCAGCAGCAGCCGGUCGGCC (((.(((((((((((..(((((((.((((..(((...)))..))))))))))).(((((....)))))((((....(((((....))))))))))))))))))))..))).... ( -60.60) >DroMoj_CAF1 46659 111 + 1 GACAGCCGCCGCAGCGUCAGCGGCCACGGCCAGCAAUGUCUUCGGCGCUGUUGGGCAGGUGAACUUUGAG---UACGCUGCCGCUGCGGCAGCUGCUGCGGCGGCCAAUCGGCC ....((((((((((((.(((((..(..(.((((((.((.(....))).)))))).)(((....)))...)---..))))).))))))))).((....)))))((((....)))) ( -59.70) >DroPer_CAF1 49221 111 + 1 GACGGCGGCCGCCA---AUGCGGCCAACGCCAGCAAUGUGUUUGGCGCUGUCGGCCAGGUGAAUCUGGAGCAGUAUGCCGCUGCAGCAGCGGCUGCCGCAGCCGCCAGCCGGCC ((((((((((((..---..))))))...((((((.....).)))))))))))((((.(((....(((.(((.(....).))).)))..(((((((...)))))))..))))))) ( -59.90) >consensus GACAGCGGCCGCCGC__CUGCGGCCACCGCCAGCAAUGUGUUCGGCGCUGUCGGCCAGGUGAAUCUGGAGCA_UAUGCCGCCGCGGCAGCGGCUGCAGCAGCCGCCAGCCGGCC ((((((.(((((.......)))))....(((((........)))))))))))(((((((....)))..........((.(((((.((((...)))).))))).)).....)))) (-39.14 = -38.32 + -0.83)

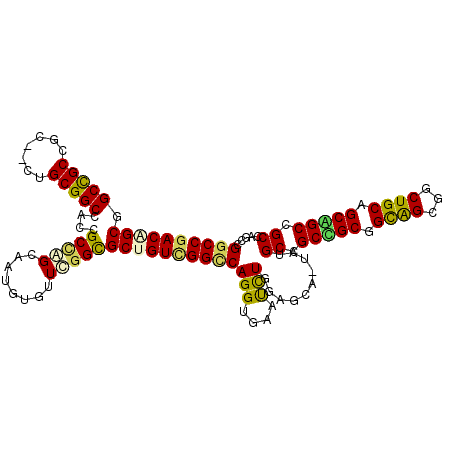
| Location | 5,271,860 – 5,271,974 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.82 |
| Mean single sequence MFE | -57.35 |
| Consensus MFE | -37.90 |
| Energy contribution | -37.60 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.48 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.95 |
| SVM RNA-class probability | 0.983748 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5271860 114 - 27905053 GGCCGGCUGGCGGCAGCUGCAGAGGCCACCGCUGCGGCAUAUUGCUCCAGAUUUACCUGGCCAACGGCACCGAACACAUUGCUGGCGGUGGCCGCAGAUGCGGCAGCGGCUGUC (((((.(((.(.(((.((((...(((((((((..(((((...((.(((((......)))(((...)))...)).))...)))))))))))))))))).))).)))))))))... ( -56.40) >DroVir_CAF1 45097 111 - 1 GGCCGACUGGCCGCCGCAGCAGCAGCCGCCGCGGCAGCAUA---CUCAAAGUUAACCUGACCGACAGCGCCAAAAACAUUGCUCGCUGUGGCCGCCGUAGCCGCGGCCGCAGUU (((((..((((.(((((((((((((.(((.(((((((...(---(.....))....))).))).).))).........))))).)))))))).))))......)))))...... ( -46.50) >DroPse_CAF1 48681 111 - 1 GGCCGGCUGGCGGCUGCGGCAGCCGCUGCUGCAGCGGCAUACUGCUCCAGAUUCACCUGGCCGACAGCGCCAAACACAUUGCUGGCGUUGGCCGCAU---UGGCGGCCGCCGUC ((((((.((((.((((((((((...)))))))))).))...(((...)))...)).))))))((((((((((..........)))))))((((((..---..))))))...))) ( -60.40) >DroWil_CAF1 56150 114 - 1 GGCCGACCGGCUGCUGCUGCAGCAGCUGCCGCGGCAGCGUAUUGCUCCAAAUUGACCUGGCCAACAGCACCGAAGACAUUGCCAGCGGUGGCCGCUGUUGCAGCAGCAGCUGUC ....(((.((((((((((((((((((.(((((((.(((.....))))).......((((((......(......).....))))).)))))).))))))))))))))))))))) ( -62.10) >DroMoj_CAF1 46659 111 - 1 GGCCGAUUGGCCGCCGCAGCAGCUGCCGCAGCGGCAGCGUA---CUCAAAGUUCACCUGCCCAACAGCGCCGAAGACAUUGCUGGCCGUGGCCGCUGACGCUGCGGCGGCUGUC ((((....))))......(((((((((((((((((((.(((---(.....))..)))))))...((((((((....((....))....))).)))))..)))))))))))))). ( -58.30) >DroPer_CAF1 49221 111 - 1 GGCCGGCUGGCGGCUGCGGCAGCCGCUGCUGCAGCGGCAUACUGCUCCAGAUUCACCUGGCCGACAGCGCCAAACACAUUGCUGGCGUUGGCCGCAU---UGGCGGCCGCCGUC ((((((.((((.((((((((((...)))))))))).))...(((...)))...)).))))))((((((((((..........)))))))((((((..---..))))))...))) ( -60.40) >consensus GGCCGACUGGCGGCUGCAGCAGCAGCCGCCGCGGCAGCAUA_UGCUCCAAAUUCACCUGGCCAACAGCGCCAAACACAUUGCUGGCGGUGGCCGCAG__GCGGCGGCCGCUGUC ....(((.(((((((((.((.((.((((((((((((((.....)))..........(((.....))).............))).)))))))).))....)).)))))))))))) (-37.90 = -37.60 + -0.30)


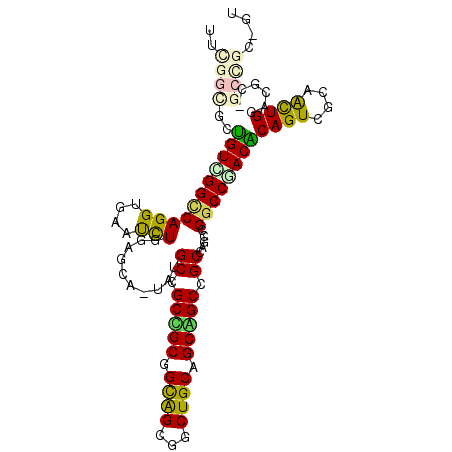
| Location | 5,271,900 – 5,272,004 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 76.23 |
| Mean single sequence MFE | -45.47 |
| Consensus MFE | -30.84 |
| Energy contribution | -30.40 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.04 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.546651 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5271900 104 + 27905053 UUCGGUGCCGUUGGCCAGGUAAAUCUGGAGCAAUAUGCCGCAGCGGUGGCCUCUGCAGCUGCCGCCAGCCGGCCGACGCAGUCGCAACUGGACGCUGCCGCAGU ..(((((..((((.(((((....)))))..)))).)))))..((((..((.(((((.(((((.(((....)))....))))).))....))).))..))))... ( -42.30) >DroVir_CAF1 45137 92 + 1 UUUGGCGCUGUCGGUCAGGUUAACUUUGAG---UAUGCUGCCGCGGCGGCUGCUGCUGCGGCGGCCAGUCGGCCAACGCAGUCUCAGCUGGAG---------GU ((..((.(((.....)))........((((---..(((.((((((((((...))))))))))((((....))))...)))..))))))..)).---------.. ( -42.00) >DroPse_CAF1 48718 104 + 1 UUUGGCGCUGUCGGCCAGGUGAAUCUGGAGCAGUAUGCCGCUGCAGCAGCGGCUGCCGCAGCCGCCAGCCGGCCGACACAGUCGCAGCUGGACGCCGCCGCGGU ...(((((((((((((.(((....(((.(((.(....).))).)))..(((((((...)))))))..)))))))))))((((....))))...).))))..... ( -49.30) >DroWil_CAF1 56190 104 + 1 UUCGGUGCUGUUGGCCAGGUCAAUUUGGAGCAAUACGCUGCCGCGGCAGCUGCUGCAGCAGCAGCCGGUCGGCCAACACAGUCACAAUUGGAAGCGGCUGCUGU ...((((((((((.(((((....)))))..))))).)).)))(((((....))))).((((((((((.(...((((...........)))).).)))))))))) ( -43.10) >DroMoj_CAF1 46699 92 + 1 UUCGGCGCUGUUGGGCAGGUGAACUUUGAG---UACGCUGCCGCUGCGGCAGCUGCUGCGGCGGCCAAUCGGCCCACGCAGUCGCAACUGGAG---------GU ....((((((((((((.((((.((.....)---).))))(((((((((((....)))))))))))......))))).)))).)))........---------.. ( -46.80) >DroPer_CAF1 49258 104 + 1 UUUGGCGCUGUCGGCCAGGUGAAUCUGGAGCAGUAUGCCGCUGCAGCAGCGGCUGCCGCAGCCGCCAGCCGGCCGACACAGUCGCAGCUGGACGCCGCCGCGGU ...(((((((((((((.(((....(((.(((.(....).))).)))..(((((((...)))))))..)))))))))))((((....))))...).))))..... ( -49.30) >consensus UUCGGCGCUGUCGGCCAGGUGAAUCUGGAGCA_UAUGCCGCCGCGGCAGCGGCUGCAGCAGCCGCCAGCCGGCCGACACAGUCGCAACUGGACGC_GCCGC_GU ..((((..(((((((((((....)))..........((.(((((.((((...)))).))))).)).....))))))))((((....))))......)))).... (-30.84 = -30.40 + -0.44)


| Location | 5,271,900 – 5,272,004 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 76.23 |
| Mean single sequence MFE | -44.52 |
| Consensus MFE | -24.70 |
| Energy contribution | -25.07 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.695318 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5271900 104 - 27905053 ACUGCGGCAGCGUCCAGUUGCGACUGCGUCGGCCGGCUGGCGGCAGCUGCAGAGGCCACCGCUGCGGCAUAUUGCUCCAGAUUUACCUGGCCAACGGCACCGAA ..(((.(((((.....))))).....(((.((((((((((.(((((((((((.((...)).)))))))....))))))))......)))))).))))))..... ( -44.60) >DroVir_CAF1 45137 92 - 1 AC---------CUCCAGCUGAGACUGCGUUGGCCGACUGGCCGCCGCAGCAGCAGCCGCCGCGGCAGCAUA---CUCAAAGUUAACCUGACCGACAGCGCCAAA ..---------.....((((...(((((.(((((....))))).)))))...))))(((.(((((((...(---(.....))....))).))).).)))..... ( -34.30) >DroPse_CAF1 48718 104 - 1 ACCGCGGCGGCGUCCAGCUGCGACUGUGUCGGCCGGCUGGCGGCUGCGGCAGCCGCUGCUGCAGCGGCAUACUGCUCCAGAUUCACCUGGCCGACAGCGCCAAA ..(((.(((((.....))))).....((((((((((.((((.((((((((((...)))))))))).))...(((...)))...)).)))))))))))))..... ( -56.30) >DroWil_CAF1 56190 104 - 1 ACAGCAGCCGCUUCCAAUUGUGACUGUGUUGGCCGACCGGCUGCUGCUGCAGCAGCUGCCGCGGCAGCGUAUUGCUCCAAAUUGACCUGGCCAACAGCACCGAA ...((((.(((........))).))))((((((((..((((.(((((....))))).)))).((.(((.....))))).........))))))))......... ( -42.60) >DroMoj_CAF1 46699 92 - 1 AC---------CUCCAGUUGCGACUGCGUGGGCCGAUUGGCCGCCGCAGCAGCUGCCGCAGCGGCAGCGUA---CUCAAAGUUCACCUGCCCAACAGCGCCGAA ..---------...(((((((...((((..((((....))))..))))))))))).(((...(((((.(((---(.....))..))))))).....)))..... ( -33.00) >DroPer_CAF1 49258 104 - 1 ACCGCGGCGGCGUCCAGCUGCGACUGUGUCGGCCGGCUGGCGGCUGCGGCAGCCGCUGCUGCAGCGGCAUACUGCUCCAGAUUCACCUGGCCGACAGCGCCAAA ..(((.(((((.....))))).....((((((((((.((((.((((((((((...)))))))))).))...(((...)))...)).)))))))))))))..... ( -56.30) >consensus AC_GCGGC_GCGUCCAGCUGCGACUGCGUCGGCCGACUGGCGGCUGCAGCAGCAGCCGCCGCGGCAGCAUA_UGCUCCAAAUUCACCUGGCCAACAGCGCCAAA ........................(((((((((((....(((((.((.((....)).)).)))))(((.....)))...........)))))))).)))..... (-24.70 = -25.07 + 0.36)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:20:47 2006