
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 5,073,413 – 5,073,554 |
| Length | 141 |
| Max. P | 0.987190 |

| Location | 5,073,413 – 5,073,527 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 75.65 |
| Mean single sequence MFE | -45.65 |
| Consensus MFE | -28.95 |
| Energy contribution | -29.57 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.987011 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
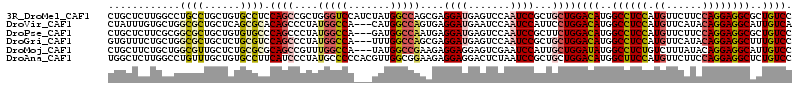
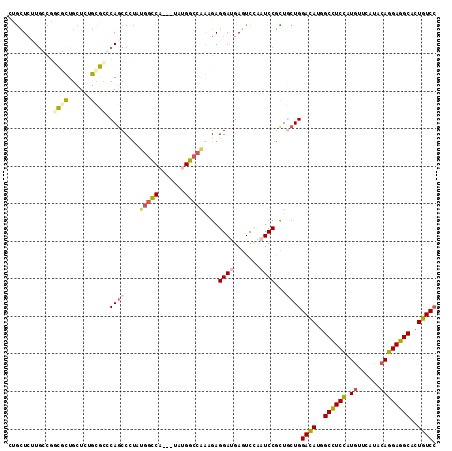
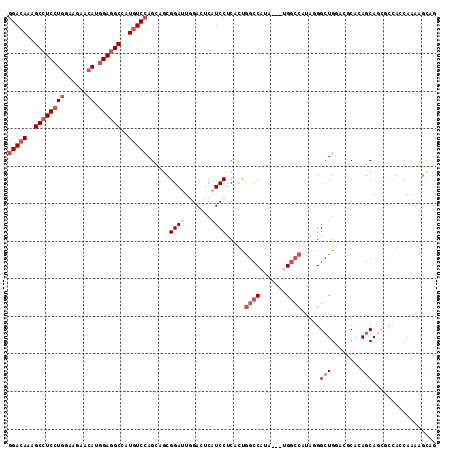
>3R_DroMel_CAF1 5073413 114 + 27905053 CUGCUCUUGGCCUGCCUGCUGUGCCUCCAGCCGCUGGGUCCAUCUAUGGCCAGCGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCCAUGUUCUUCCAGGAGGCGCUGUCC ..((....((....)).)).(((((((((((((((((..(((....))))))))).((((.......)))))))...((((((((...)))))))).....))))))))..... ( -47.50) >DroVir_CAF1 19 111 + 1 CUAUUUGUGCUGGCGCUGCUCAGCGCACAGCCCUAUGGCCA---CAUGGCCAGUGAGGAUGAAUCCAAUCCAUUCCUGGACAUGGCCUCCAUGUUCAUACAGGAGGCAUUGUCA ..(((..((((((((((....))))).))))(((.(((((.---...)))))...))))..)))..............((((..((((((.(((....)))))))))..)))). ( -47.20) >DroPse_CAF1 22 111 + 1 CUGCUCUUCGCGGCGCUGCUGUGUGCCCAGCCCUAUGGCCA---GAUGGCCAAUGAGGAUGAGUCCAAUCCGCUUCUGGACAUGGCCUCCAUGUUCUUCCAGGAGGCGCUGUCC ..((.....))(((((......)))))(((((((.(((((.---...)))))...)))....(((((.........)))))...((((((.((......))))))))))))... ( -44.10) >DroGri_CAF1 911 111 + 1 GUGUUUCUGCUGGCGCUGCUCUGCGUCCAGCCCUAUGGCCA---UUUGGCCAGCGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCCAUGUUCAUACAGGAGGCUUUGUCC ........((((((((......))).))))).....((((.---...))))((((.((((.......))))..))))(((((.(((((((.(((....)))))))))).))))) ( -52.60) >DroMoj_CAF1 890 111 + 1 CUGCUUCUGCUGGCGUUGCUCUGCGCGCAGCCGUUUGGCCA---UAUGGCCGAAGAGGAGGAGUCGAAUCCAUUGCUGGAUAUGGCCUCUGUCUUUAUACAGGAGGCAUUGUCC .(((((((((((((((......)))).))))..(((((((.---...)))))))((((((((((((.(((((....))))).))).)))).))))).....)))))))...... ( -44.10) >DroAna_CAF1 373 114 + 1 UGGCUCUUGGCCUGUUUGCUGUGCCUUCAUCCCUAUGCCCCCACGUUGGCGGAAGAGGAGGACUCUAAUCCGCUGCUGGACAUGGCUUCCAUGUUCUUCCAGGAGGCUCUGUCC .(((.....))).....((.(((....(((....)))....)))...((((((((((.....))))..)))))))).(((((.(((((((.((......))))))))).))))) ( -38.40) >consensus CUGCUCUUGCCGGCGCUGCUCUGCGCCCAGCCCUAUGGCCA___UAUGGCCAAAGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCCAUGUUCAUACAGGAGGCACUGUCC ............((((......)))).((((....(((((.......)))))....((((.......))))...))))((((..((((((.((......))))))))..)))). (-28.95 = -29.57 + 0.62)

| Location | 5,073,413 – 5,073,527 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 75.65 |
| Mean single sequence MFE | -45.17 |
| Consensus MFE | -23.82 |
| Energy contribution | -25.65 |
| Covariance contribution | 1.83 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.53 |
| SVM decision value | 2.07 |
| SVM RNA-class probability | 0.987190 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
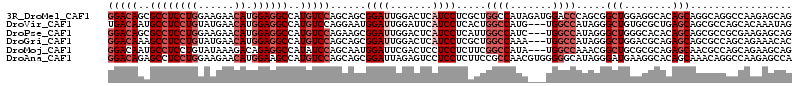
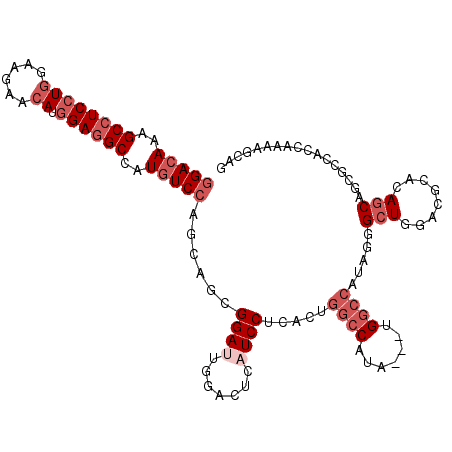
>3R_DroMel_CAF1 5073413 114 - 27905053 GGACAGCGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCGCUGGCCAUAGAUGGACCCAGCGGCUGGAGGCACAGCAGGCAGGCCAAGAGCAG (((((..((((((((......)).))))))..)))))....((...((((.((...((((((((((((....)))..))))))((((......)))))))..))))))..)).. ( -50.10) >DroVir_CAF1 19 111 - 1 UGACAAUGCCUCCUGUAUGAACAUGGAGGCCAUGUCCAGGAAUGGAUUGGAUUCAUCCUCACUGGCCAUG---UGGCCAUAGGGCUGUGCGCUGAGCAGCGCCAGCACAAAUAG .((((..(((((((((....))).))))))..)))).((((.(((((...)))))))))...(((((...---.)))))....((((.(((((....)))))))))........ ( -45.80) >DroPse_CAF1 22 111 - 1 GGACAGCGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGAAGCGGAUUGGACUCAUCCUCAUUGGCCAUC---UGGCCAUAGGGCUGGGCACACAGCAGCGCCGCGAAGAGCAG (((((..((((((((......)).))))))..)))))....((((...........(((...(((((...---.))))).)))((((......))))....))))......... ( -47.30) >DroGri_CAF1 911 111 - 1 GGACAAAGCCUCCUGUAUGAACAUGGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCGCUGGCCAAA---UGGCCAUAGGGCUGGACGCAGAGCAGCGCCAGCAGAAACAC (((((..(((((((((....))).))))))..)))))...(((((...(((....))))))))((((...---.)))).....(((((.(((......))))))))........ ( -50.20) >DroMoj_CAF1 890 111 - 1 GGACAAUGCCUCCUGUAUAAAGACAGAGGCCAUAUCCAGCAAUGGAUUCGACUCCUCCUCUUCGGCCAUA---UGGCCAAACGGCUGCGCGCAGAGCAACGCCAGCAGAAGCAG (((....(((((.(((......))))))))(..(((((....)))))..)..)))..((((((((((...---.)))).....((((.(((........))))))).)))).)) ( -40.00) >DroAna_CAF1 373 114 - 1 GGACAGAGCCUCCUGGAAGAACAUGGAAGCCAUGUCCAGCAGCGGAUUAGAGUCCUCCUCUUCCGCCAACGUGGGGGCAUAGGGAUGAAGGCACAGCAAACAGGCCAAGAGCCA (((((..((.(((((......)).))).))..))))).((.((((((....))))(((((((((((....)))))))....)))).....))...)).....(((.....))). ( -37.60) >consensus GGACAAAGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCACUGGCCAUA___UGGCCAUAGGGCUGGACGCACAGCAGCGCCACCAAAAGCAG (((((..((((((((......)).))))))..)))))......((((.......)))).....((((.......)))).....(((........)))................. (-23.82 = -25.65 + 1.83)
| Location | 5,073,453 – 5,073,554 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 80.22 |
| Mean single sequence MFE | -40.75 |
| Consensus MFE | -24.82 |
| Energy contribution | -25.35 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.874001 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
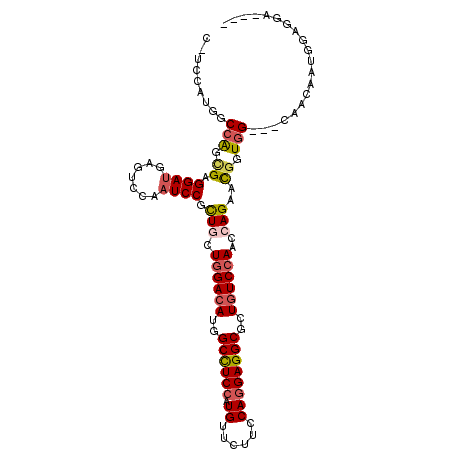
>3R_DroMel_CAF1 5073453 101 + 27905053 CAUCUAUGGCCAGCGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCCAUGUUCUUCCAGGAGGCGCUGUCCAACCAGAACGGUGG---CAACAAUGGAGGA---- ..(((((.((((.((.((((.......)))).(((.((((((..((((((.((......))))))))..))))))..)))..)).)))---)....)))))...---- ( -41.80) >DroVir_CAF1 59 102 + 1 A---CAUGGCCAGUGAGGAUGAAUCCAAUCCAUUCCUGGACAUGGCCUCCAUGUUCAUACAGGAGGCAUUGUCAAAUCAGAACGGUGG---CGGUGGCGGUGCUGCUG .---(((.((((.((.((((.......))))....(((((((..((((((.(((....)))))))))..))))....)))..)).)))---).)))(((....))).. ( -38.80) >DroPse_CAF1 62 98 + 1 A---GAUGGCCAAUGAGGAUGAGUCCAAUCCGCUUCUGGACAUGGCCUCCAUGUUCUUCCAGGAGGCGCUGUCCAACCAGAAUGGUGG---CGGCGGAGGAGCA---- .---.....((.....(((....)))..((((((.(((((((..((((((.((......))))))))..))))).(((.....)))))---.))))))))....---- ( -37.80) >DroSim_CAF1 150 101 + 1 CUUCCAUGGCCAGCGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCCAUGUUCUUCCAGGAGGCGCUGUCCAACCAGAACGGUGG---CAACAAUGGAGGA---- (((((((.((((.((.((((.......)))).(((.((((((..((((((.((......))))))))..))))))..)))..)).)))---)....))))))).---- ( -47.30) >DroYak_CAF1 153 101 + 1 CUUCUCUCGCCAGCGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCGAUGUUCUUCCAGGAGGCACUCUCCAACCAGAACGGUGG---CAACAAUGGUGGC---- ......((((((.(((((....(((((.........)))))....))))).((((((....((((.....))))....)))))).((.---...)).)))))).---- ( -32.80) >DroAna_CAF1 413 104 + 1 CCACGUUGGCGGAAGAGGAGGACUCUAAUCCGCUGCUGGACAUGGCUUCCAUGUUCUUCCAGGAGGCUCUGUCCAAUCAGAAUGGUGGCGGCAACAACGGUGGC---- ((((((((..(((((((.....))))..)))(((((((((((.(((((((.((......))))))))).))))).(((.....)))))))))..)))).)))).---- ( -46.00) >consensus C_UCCAUGGCCAGCGAGGAUGAGUCCAAUCCGCUGCUGGACAUGGCCUCCAUGUUCUUCCAGGAGGCGCUGUCCAACCAGAACGGUGG___CAACAAUGGAGGA____ .........(((.((.((((.......)))).(((.((((((..((((((.((......))))))))..))))))..)))..)).))).................... (-24.82 = -25.35 + 0.53)


| Location | 5,073,453 – 5,073,554 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 80.22 |
| Mean single sequence MFE | -37.58 |
| Consensus MFE | -25.51 |
| Energy contribution | -25.98 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.74 |
| SVM RNA-class probability | 0.838680 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 5073453 101 - 27905053 ----UCCUCCAUUGUUG---CCACCGUUCUGGUUGGACAGCGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCGCUGGCCAUAGAUG ----.......((((.(---(((((((....((((((((..((((((((......)).))))))..)))))))).))))...(((....)))....)))).))))... ( -40.20) >DroVir_CAF1 59 102 - 1 CAGCAGCACCGCCACCG---CCACCGUUCUGAUUUGACAAUGCCUCCUGUAUGAACAUGGAGGCCAUGUCCAGGAAUGGAUUGGAUUCAUCCUCACUGGCCAUG---U .....(((..((((...---...(((((((.....((((..(((((((((....))).))))))..))))..)))))))...(((....)))....))))..))---) ( -34.10) >DroPse_CAF1 62 98 - 1 ----UGCUCCUCCGCCG---CCACCAUUCUGGUUGGACAGCGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGAAGCGGAUUGGACUCAUCCUCAUUGGCCAUC---U ----.(((..(((((.(---(((......))))((((((..((((((((......)).))))))..))))))...)))))..(((....))).....)))....---. ( -36.10) >DroSim_CAF1 150 101 - 1 ----UCCUCCAUUGUUG---CCACCGUUCUGGUUGGACAGCGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCGCUGGCCAUGGAAG ----...(((((....(---(((((((....((((((((..((((((((......)).))))))..)))))))).))))...(((....)))....)))).))))).. ( -43.00) >DroYak_CAF1 153 101 - 1 ----GCCACCAUUGUUG---CCACCGUUCUGGUUGGAGAGUGCCUCCUGGAAGAACAUCGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCGCUGGCGAGAGAAG ----.......((.(((---(((..(((((....((((.....))))....)))))..(((((....(((((((....).))))))....))))).)))))).))... ( -34.90) >DroAna_CAF1 413 104 - 1 ----GCCACCGUUGUUGCCGCCACCAUUCUGAUUGGACAGAGCCUCCUGGAAGAACAUGGAAGCCAUGUCCAGCAGCGGAUUAGAGUCCUCCUCUUCCGCCAACGUGG ----.((((.((((...............((.(((((((..((.(((((......)).))).))..)))))))))(((((..((((.....))))))))))))))))) ( -37.20) >consensus ____UCCACCAUUGUUG___CCACCGUUCUGGUUGGACAGCGCCUCCUGGAAGAACAUGGAGGCCAUGUCCAGCAGCGGAUUGGACUCAUCCUCGCUGGCCAUAGA_G .......................(((.((((.(((((((..((((((((......)).))))))..)))))))...)))).)))........................ (-25.51 = -25.98 + 0.47)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:18:37 2006