
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,904,807 – 4,904,913 |
| Length | 106 |
| Max. P | 0.789352 |

| Location | 4,904,807 – 4,904,913 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 76.35 |
| Mean single sequence MFE | -22.18 |
| Consensus MFE | -8.26 |
| Energy contribution | -9.08 |
| Covariance contribution | 0.82 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.629809 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
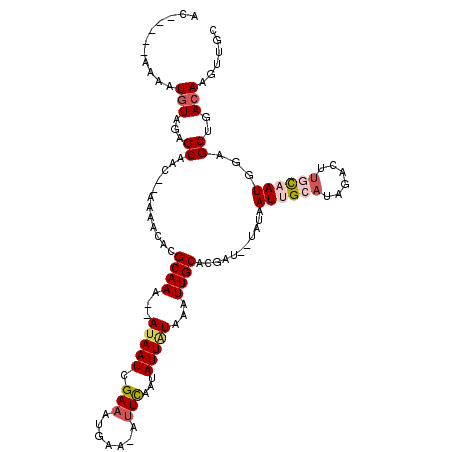
>3R_DroMel_CAF1 4904807 106 + 27905053 AA-----AGAAUAUAGAGCAAC--AAAGCACGCAAA--AUAAUCGAAAUGAAAAUUCAGCAUUAUAAAUUGCACGAU--UAUAAUUGCAUAGACUUGCAAUGGAGCUGACAAGUUGC ..-----..........(((((--....((.((...--(((((((...(((....)))(((........))).))))--))).((((((......))))))...))))....))))) ( -23.90) >DroVir_CAF1 16379 104 + 1 AU-----UAAGUGUAGCGCACACGAAGACACGCAAA--AUAAUAGAAAUGAA-AUUUGAAAUUUUAAAUUGCAUUAUAUUGUUAUGGCAUGAA----UAAUA-UGCUGACAAGUUGU ..-----...((((..((....))...))))(((((--((((((..((((.(-((((((....))))))).)))).)))))))..((((((..----...))-))))......)))) ( -19.40) >DroGri_CAF1 16393 105 + 1 UG-----CAAUUGUAGCGCAAUUGCAGACACGCAACAUAUAAUAGAAAUGGA-AUUUGAGAUUAUAAAUUGCAUUAUUUUGUUAUGGC-UGAA----UAAUA-UGCUGACAUGUUGG ((-----((((((.....))))))))......((((((((((((((((((.(-(((((......)))))).)))..)))))))))(((-....----.....-.)))...)))))). ( -25.20) >DroSec_CAF1 12645 106 + 1 AC-----AGAAUGUAGAGCAAC--AAAGCACGCAAA--AUAAUCGAAAUGAAAAUUCAAUAUUAUAAAUUGCACGAU--UAUAAUUGCAUAGACUUGCAAUGGAGCUGACAAGUUGC ..-----..........(((((--....((.((...--(((((((.(((....)))((((.......))))..))))--))).((((((......))))))...))))....))))) ( -19.60) >DroSim_CAF1 12630 106 + 1 AC-----AGAAUGUAGAGCAAC--AAAGCACGCAAA--AUAAUCGAAAUGAAAACUCAAUAUUAUAAAUUGCACGAU--UAUAAUUGCAUAGACUUGCAAUGGAGCUGACAAGUUGC ..-----..........(((((--....((.((...--(((((((.(((..................)))...))))--))).((((((......))))))...))))....))))) ( -18.17) >DroYak_CAF1 12487 110 + 1 AUACAUGAAAAUGUAGAGCAAC--AAAACACGCAAA--AUAAUCGAAAUGAA-AUUCGAUAUUGUAAAUUGCACGAU--UAUAAUUGCACAGACGUGCAAUGUGGCUGACAAGUUGC .(((((....)))))..(((((--.......(((((--(((.(((((.....-.))))))))).....)))).((.(--((((.((((((....))))))))))).))....))))) ( -26.80) >consensus AC_____AAAAUGUAGAGCAAC__AAAACACGCAAA__AUAAUCGAAAUGAA_AUUCAAUAUUAUAAAUUGCACGAU__UAUAAUUGCAUAGACUUGCAAUGGAGCUGACAAGUUGC ...........(((...((............((((...(((((.(((.......)))...)))))...))))...........((((((......))))))...))..)))...... ( -8.26 = -9.08 + 0.82)

| Location | 4,904,807 – 4,904,913 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 76.35 |
| Mean single sequence MFE | -21.42 |
| Consensus MFE | -7.87 |
| Energy contribution | -9.97 |
| Covariance contribution | 2.10 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.789352 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
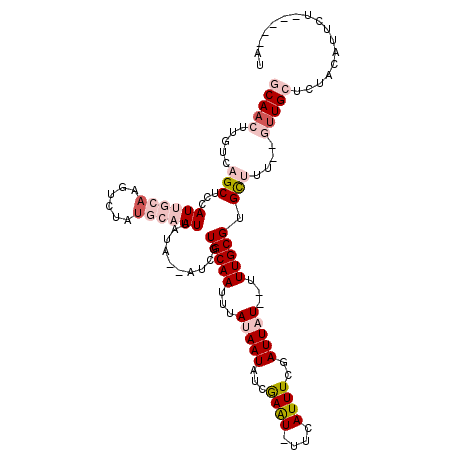
>3R_DroMel_CAF1 4904807 106 - 27905053 GCAACUUGUCAGCUCCAUUGCAAGUCUAUGCAAUUAUA--AUCGUGCAAUUUAUAAUGCUGAAUUUUCAUUUCGAUUAU--UUUGCGUGCUUU--GUUGCUCUAUAUUCU-----UU (((((......((......))((((..((((((..(((--((((.(((........)))(((....)))...)))))))--.)))))))))).--)))))..........-----.. ( -22.50) >DroVir_CAF1 16379 104 - 1 ACAACUUGUCAGCA-UAUUA----UUCAUGCCAUAACAAUAUAAUGCAAUUUAAAAUUUCAAAU-UUCAUUUCUAUUAU--UUUGCGUGUCUUCGUGUGCGCUACACUUA-----AU ......(((..(((-(((..----..(((((...((.((((.((((.(((((........))))-).))))..)))).)--)..))))).....))))))...)))....-----.. ( -15.80) >DroGri_CAF1 16393 105 - 1 CCAACAUGUCAGCA-UAUUA----UUCA-GCCAUAACAAAAUAAUGCAAUUUAUAAUCUCAAAU-UCCAUUUCUAUUAUAUGUUGCGUGUCUGCAAUUGCGCUACAAUUG-----CA ...(((((.(((((-(((..----....-.............((((.(((((........))))-).))))......)))))))))))))..(((((((.....))))))-----). ( -20.11) >DroSec_CAF1 12645 106 - 1 GCAACUUGUCAGCUCCAUUGCAAGUCUAUGCAAUUAUA--AUCGUGCAAUUUAUAAUAUUGAAUUUUCAUUUCGAUUAU--UUUGCGUGCUUU--GUUGCUCUACAUUCU-----GU (((((......((......))((((..((((((..(((--((((...((((((......)))))).......)))))))--.)))))))))).--)))))..........-----.. ( -22.00) >DroSim_CAF1 12630 106 - 1 GCAACUUGUCAGCUCCAUUGCAAGUCUAUGCAAUUAUA--AUCGUGCAAUUUAUAAUAUUGAGUUUUCAUUUCGAUUAU--UUUGCGUGCUUU--GUUGCUCUACAUUCU-----GU (((((......((......))((((..((((((..(((--((((...((((((......)))))).......)))))))--.)))))))))).--)))))..........-----.. ( -22.00) >DroYak_CAF1 12487 110 - 1 GCAACUUGUCAGCCACAUUGCACGUCUGUGCAAUUAUA--AUCGUGCAAUUUACAAUAUCGAAU-UUCAUUUCGAUUAU--UUUGCGUGUUUU--GUUGCUCUACAUUUUCAUGUAU (((((....((((...((((((((..((((....))))--..)))))))).......((((((.-.....))))))...--...)).))....--)))))..(((((....))))). ( -26.10) >consensus GCAACUUGUCAGCUCCAUUGCAAGUCUAUGCAAUUAUA__AUCGUGCAAUUUAUAAUAUCGAAU_UUCAUUUCGAUUAU__UUUGCGUGCUUU__GUUGCUCUACAUUCU_____AU (((((.....(((...((((((......))))))..........(((((...(((((...((((....))))..)))))...))))).)))....)))))................. ( -7.87 = -9.97 + 2.10)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:17:27 2006