
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,898,521 – 4,898,666 |
| Length | 145 |
| Max. P | 0.991922 |

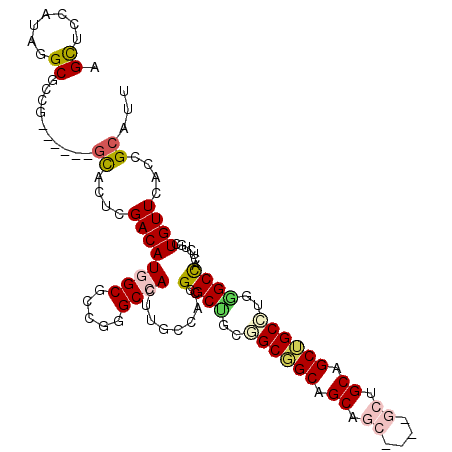
| Location | 4,898,521 – 4,898,635 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 72.52 |
| Mean single sequence MFE | -50.18 |
| Consensus MFE | -21.79 |
| Energy contribution | -22.48 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.43 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.828770 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
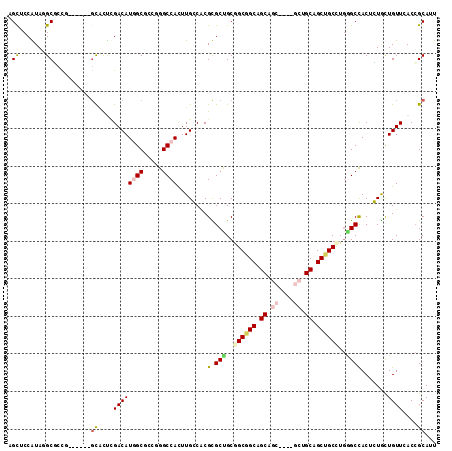
>3R_DroMel_CAF1 4898521 114 + 27905053 GGCUCCGUAGGCUCCACUGGCCGCUCCCGACAUCGCUCCCGGCCACUUGCCACGCGCUGCGGCGGCUGCUGCGGCAGCUGCUGCUGCCUGGGCCACUGUGCUGUUCACCGCAUU ((((((((.(((.....((((((....((....))....))))))...)))))).((.(((((((((((....))))))))))).))..)))))...((((.(....).)))). ( -54.00) >DroVir_CAF1 8379 105 + 1 AGCUCCAUACGCGCUC------GCAUUGGACAUGGCGCCGGGCCACUUGGCGCGUGCUGCGGCCGCAGCAGC---UGCUGCAGCGGCCUGGGCCACGCUACUGUUGACCGCAUU ..........(((.((------(((.(((.(.((((.(((((((.((((((((.((((((....))))))))---.)))).)).))))))))))).)))).)).))).)))... ( -51.30) >DroGri_CAF1 8968 102 + 1 CGCUCCAUAAGCGCUG------GCAUUCGACAUGGCCCCGGGCCAUUUGGCGCGUGCCGCAGCAGCUGCCAC------AGCAGCCGCCUGGGCCACACUAUUGUUGACCGCAUU ((((.....))))...------((..((((((((((((((.(((....))).)).......((.(((((...------.))))).))..))))))......))))))..))... ( -39.80) >DroWil_CAF1 7754 108 + 1 AGUUCCAUAGGCCCC---GGCGGCACUUGACAUGGCACCUGGCCAUUUGCCACGUGCCGCUGCGGCAGCAGCAGCGGCUGCAGCUGC---AGCUACUGUGCUGUUGACGGCAUU ..........(((.(---((((((((.......(((((.((((.....)))).)))))((((((((.(((((....))))).)))))---)))....)))))))))..)))... ( -59.10) >DroYak_CAF1 6597 114 + 1 UGCGCCGUAGGCUCCGCUGGCCGCUCCCGACAUCGCUCCCGGCCACUUGCCACGCGCUGCGGCGGCUGCUGCGGCAGCUGCUGCUGCCUGGGCCACUGUGCUGUUCACCGCAUU ((((..((..((..(((((((((....((....))....))))))...(((.((.((.(((((((((((....))))))))))).)).)))))....)))..))..)))))).. ( -52.60) >DroMoj_CAF1 7818 102 + 1 CGCUCCGUAUGCGCUG------CUAUUCGACAUGGCGCCGGGCCAUUUGGCUCGCGCUGCAGCCGCAGCAGC------UGCAGCGGCUUGAGCCACGCUGCUGUUCACCGCAUU ........((((((((------((.........(((((.(((((....))))))))))((....))))))))------.((((((((....)))..)))))........)))). ( -44.30) >consensus AGCUCCAUAGGCGCCG______GCACUCGACAUGGCGCCGGGCCACUUGCCACGCGCUGCGGCGGCAGCAGC____GCUGCAGCUGCCUGGGCCACUCUGCUGUUCACCGCAUU .((.......))..........((....((((((((.....))))........(.(((..((((((.((.((....)).)).))))))..)))).......))))....))... (-21.79 = -22.48 + 0.70)

| Location | 4,898,521 – 4,898,635 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 72.52 |
| Mean single sequence MFE | -50.60 |
| Consensus MFE | -22.33 |
| Energy contribution | -22.92 |
| Covariance contribution | 0.58 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.44 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.514602 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
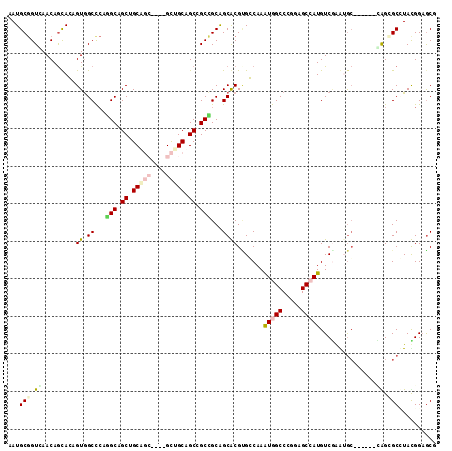
>3R_DroMel_CAF1 4898521 114 - 27905053 AAUGCGGUGAACAGCACAGUGGCCCAGGCAGCAGCAGCUGCCGCAGCAGCCGCCGCAGCGCGUGGCAAGUGGCCGGGAGCGAUGUCGGGAGCGGCCAGUGGAGCCUACGGAGCC ..(((.(....).)))((.(((((..((((((....))))))...((..(((.((..((.(.((((.....)))).).))..)).)))..))))))).))..(((....).)). ( -49.40) >DroVir_CAF1 8379 105 - 1 AAUGCGGUCAACAGUAGCGUGGCCCAGGCCGCUGCAGCA---GCUGCUGCGGCCGCAGCACGCGCCAAGUGGCCCGGCGCCAUGUCCAAUGC------GAGCGCGUAUGGAGCU .(((((.((..((...(((((((((.((((((((((((.---...)))))(((.((.....))))).))))))).)).)))))))....)).------)).)))))........ ( -53.20) >DroGri_CAF1 8968 102 - 1 AAUGCGGUCAACAAUAGUGUGGCCCAGGCGGCUGCU------GUGGCAGCUGCUGCGGCACGCGCCAAAUGGCCCGGGGCCAUGUCGAAUGC------CAGCGCUUAUGGAGCG ..((.(((((((....)).)))))))(((((((((.------...)))))))))((((((..((....((((((...))))))..))..)))------).))((((...)))). ( -45.40) >DroWil_CAF1 7754 108 - 1 AAUGCCGUCAACAGCACAGUAGCU---GCAGCUGCAGCCGCUGCUGCUGCCGCAGCGGCACGUGGCAAAUGGCCAGGUGCCAUGUCAAGUGCCGCC---GGGGCCUAUGGAACU ...(((.....((((((((..(((---((....)))))..))).)))))(((..(((((((.(((((..((((.....))))))))).))))))))---))))).......... ( -55.40) >DroYak_CAF1 6597 114 - 1 AAUGCGGUGAACAGCACAGUGGCCCAGGCAGCAGCAGCUGCCGCAGCAGCCGCCGCAGCGCGUGGCAAGUGGCCGGGAGCGAUGUCGGGAGCGGCCAGCGGAGCCUACGGCGCA ..(((((((.....))).(((((...((((((....))))))......)))))))))(((((((((..(((((((....((....))....)))))).)...))).)).)))). ( -51.40) >DroMoj_CAF1 7818 102 - 1 AAUGCGGUGAACAGCAGCGUGGCUCAAGCCGCUGCA------GCUGCUGCGGCUGCAGCGCGAGCCAAAUGGCCCGGCGCCAUGUCGAAUAG------CAGCGCAUACGGAGCG .(((((.((.....(.(((((((....(((((((((------((((...))))))))))..(.(((....))).))))))))))).).....------)).)))))........ ( -48.80) >consensus AAUGCGGUCAACAGCACAGUGGCCCAGGCAGCUGCAGC____GCUGCAGCCGCCGCAGCACGUGCCAAAUGGCCCGGAGCCAUGUCGAAUGC______CAGCGCCUACGGAGCG ...(((.((.........((.((...(((.((.(((((....))))).)).)))...)))).......(((((.....)))))...............)).))).......... (-22.33 = -22.92 + 0.58)


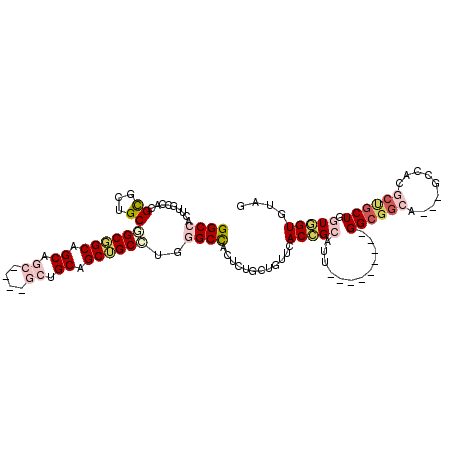
| Location | 4,898,561 – 4,898,666 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 73.58 |
| Mean single sequence MFE | -52.00 |
| Consensus MFE | -23.45 |
| Energy contribution | -25.00 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.45 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948257 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
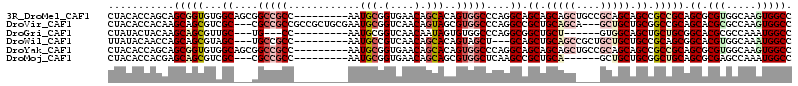
>3R_DroMel_CAF1 4898561 105 + 27905053 GGCCACUUGCCACGCGCUGCGGCGGCUGCUGCGGCAGCUGCUGCUGCCUGGGCCACUGUGCUGUUCACCGCAUU---------GGCGGCCGCUGCCACACCGCUGCUGGUGUAG ((((.....(((.(.((.(((((((((((....))))))))))).))))))((((.((((........)))).)---------)))))))......((((((....)))))).. ( -54.00) >DroVir_CAF1 8413 108 + 1 GGCCACUUGGCGCGUGCUGCGGCCGCAGCAGC---UGCUGCAGCGGCCUGGGCCACGCUACUGUUGACCGCAUUCGCAGCGGCGGCGGCG---GCGACGCUGCUUGUGGUGUAG .(((((..(((((((((((((((.((....))---.))))))))(((....))).(((..((((((.((((.......))))))))))..---))))))).))).))))).... ( -62.10) >DroGri_CAF1 9002 93 + 1 GGCCAUUUGGCGCGUGCCGCAGCAGCUGCCAC------AGCAGCCGCCUGGGCCACACUAUUGUUGACCGCAUU---------GG---CA---GCAACGCUGCUUGUAGUAUAG ((((....((((..(((....)))(((((...------.)))))))))..))))............((..((..---------((---((---((...))))))))..)).... ( -34.90) >DroWil_CAF1 7791 99 + 1 GGCCAUUUGCCACGUGCCGCUGCGGCAGCAGCAGCGGCUGCAGCUGC---AGCUACUGUGCUGUUGACGGCAUU---------GGCGGCA---GCUACGCUGCUGGUUGUAUAA ((((....((((.((((((...((((((((.(((..(((((....))---)))..))))))))))).)))))))---------)))((((---((...))))))))))...... ( -55.30) >DroYak_CAF1 6637 105 + 1 GGCCACUUGCCACGCGCUGCGGCGGCUGCUGCGGCAGCUGCUGCUGCCUGGGCCACUGUGCUGUUCACCGCAUU---------GGCGGCCGCUGCCACACCGCUGCUGGUGUAG ((((.....(((.(.((.(((((((((((....))))))))))).))))))((((.((((........)))).)---------)))))))......((((((....)))))).. ( -54.00) >DroMoj_CAF1 7852 96 + 1 GGCCAUUUGGCUCGCGCUGCAGCCGCAGCAGC------UGCAGCGGCUUGAGCCACGCUGCUGUUCACCGCAUU---------GGCGGCG---GCGACGCUGCUCGUGGUGUAG (((....((((((((((((((((.......))------)))))))...)))))))....)))...((((((...---------(((((((---....))))))).))))))... ( -51.70) >consensus GGCCACUUGCCACGCGCUGCGGCGGCAGCAGC____GCUGCAGCUGCCUGGGCCACUCUGCUGUUCACCGCAUU_________GGCGGCA___GCCACGCUGCUGGUGGUGUAG ((((.........((...))((((((.((.((....)).)).))))))..))))............(((((............((((((.........)))))).))))).... (-23.45 = -25.00 + 1.56)

| Location | 4,898,561 – 4,898,666 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 73.58 |
| Mean single sequence MFE | -49.87 |
| Consensus MFE | -20.82 |
| Energy contribution | -22.32 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.42 |
| SVM decision value | 2.30 |
| SVM RNA-class probability | 0.991922 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4898561 105 - 27905053 CUACACCAGCAGCGGUGUGGCAGCGGCCGCC---------AAUGCGGUGAACAGCACAGUGGCCCAGGCAGCAGCAGCUGCCGCAGCAGCCGCCGCAGCGCGUGGCAAGUGGCC ...(((..((.(((.((((((.((((((((.---------..(((.(....).)))..))))))..((((((....))))))......)).)))))).)))...))..)))... ( -54.60) >DroVir_CAF1 8413 108 - 1 CUACACCACAAGCAGCGUCGC---CGCCGCCGCCGCUGCGAAUGCGGUCAACAGUAGCGUGGCCCAGGCCGCUGCAGCA---GCUGCUGCGGCCGCAGCACGCGCCAAGUGGCC .....((((..((.(((((((---.(((((.((.(((((...(((........)))(((((((....))))).)).)))---)).)).))))).)).).))))))...)))).. ( -52.30) >DroGri_CAF1 9002 93 - 1 CUAUACUACAAGCAGCGUUGC---UG---CC---------AAUGCGGUCAACAAUAGUGUGGCCCAGGCGGCUGCU------GUGGCAGCUGCUGCGGCACGCGCCAAAUGGCC ..............((((.((---((---(.---------..((.(((((((....)).)))))))(((((((((.------...))))))))))))))))))(((....))). ( -42.20) >DroWil_CAF1 7791 99 - 1 UUAUACAACCAGCAGCGUAGC---UGCCGCC---------AAUGCCGUCAACAGCACAGUAGCU---GCAGCUGCAGCCGCUGCUGCUGCCGCAGCGGCACGUGGCAAAUGGCC ........((((((((...))---))).(((---------(.((((((...((((((((..(((---((....)))))..))).))))).....))))))..))))...))).. ( -47.40) >DroYak_CAF1 6637 105 - 1 CUACACCAGCAGCGGUGUGGCAGCGGCCGCC---------AAUGCGGUGAACAGCACAGUGGCCCAGGCAGCAGCAGCUGCCGCAGCAGCCGCCGCAGCGCGUGGCAAGUGGCC ...(((..((.(((.((((((.((((((((.---------..(((.(....).)))..))))))..((((((....))))))......)).)))))).)))...))..)))... ( -54.60) >DroMoj_CAF1 7852 96 - 1 CUACACCACGAGCAGCGUCGC---CGCCGCC---------AAUGCGGUGAACAGCAGCGUGGCUCAAGCCGCUGCA------GCUGCUGCGGCUGCAGCGCGAGCCAAAUGGCC .....(((...((.((.((((---(((....---------...)))))))...)).)).((((((..((.((((((------((((...))))))))))))))))))..))).. ( -48.10) >consensus CUACACCACCAGCAGCGUCGC___CGCCGCC_________AAUGCGGUCAACAGCACAGUGGCCCAGGCAGCUGCAGC____GCUGCAGCCGCCGCAGCACGUGCCAAAUGGCC ...........(((((...((....(((((............(((.(....).)))..)))))....)).((.(((((....))))).)).))))).((.(((.....))))). (-20.82 = -22.32 + 1.50)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:17:25 2006