
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,895,553 – 4,895,645 |
| Length | 92 |
| Max. P | 0.997308 |

| Location | 4,895,553 – 4,895,645 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 77.28 |
| Mean single sequence MFE | -28.82 |
| Consensus MFE | -13.34 |
| Energy contribution | -12.98 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.71 |
| Structure conservation index | 0.46 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963967 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
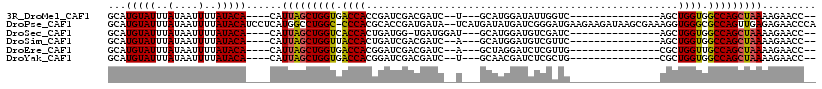
>3R_DroMel_CAF1 4895553 92 + 27905053 --GGUUCUUUUAGCUGGCCACCAGCU---------------GACCAAUAUCCAUGC---A--GAUCGUCGAUCGGUGGUCACCAGCUAAUG----UGUAUAAAAUUAUAAAUACAUGC --.......(((((((((((((.(((---------------(.........)).))---.--((((...)))))))))...)))))))).(----(((((..........)))))).. ( -26.00) >DroPse_CAF1 30450 115 + 1 UGGGUUCUCUCAACUGGCGCCCACCUUUCGCUUAUCUUCUUCAUCCCGAUCAUAUCAUGA--UAUCAUCGGUGCGUGGG-GCCAGGCCAUGAGGAUGUAUAAAAUUAUAAAUACAUGC ((.((((((....(((((.(((((.....((..............(((((.((((....)--))).))))).)))))))-))))).....)))))).))................... ( -31.16) >DroSec_CAF1 3585 93 + 1 --GGUUCUUUUAGCUGGCCACCAGCU---------------GAUCGACAUCCAUGC---AUCCAUCA-CCAUCAGUGGUGACCAGCUAAUG----UGUAUAAAAUUAUAAAUACAUGC --.......((((((((.(((((.((---------------(((.(..........---........-).)))))))))).)))))))).(----(((((..........)))))).. ( -26.27) >DroSim_CAF1 3518 92 + 1 --GGUUCUUUUAGCUGGCCACCAGCU---------------GAACGACAUCCAUGC---U--GAUCGUCGAUCAGUGGUAACCAGCUAAUG----UGUAUAAAAUUAUAAAUACAUGC --.......((((((((..((((.((---------------((.(((((((.....---.--))).)))).))))))))..)))))))).(----(((((..........)))))).. ( -31.80) >DroEre_CAF1 3129 92 + 1 --GGUUCUUUUAGCUGGCAACCAGCG---------------CAACGAGAUCCUAGC---U--GAUCGUCGAUCCGUGGUCACCAGCUAAUG----UGUAUAAAAUUAUAAAUACAUGC --.......((((((((..((((.((---------------...(((((((.....---.--)))).)))...))))))..)))))))).(----(((((..........)))))).. ( -27.40) >DroYak_CAF1 3626 92 + 1 --GGUUCUUUUAGCUGGCCACCAGCG---------------CAGCGAGAUCGUUGC---A--GAUCGUCGAUCCGUGGUCACCAGCUAAUG----UGUAUAAAAUUAUAAAUACAUGC --.......((((((((..((((..(---------------(((((....))))))---.--((((...))))..))))..)))))))).(----(((((..........)))))).. ( -30.30) >consensus __GGUUCUUUUAGCUGGCCACCAGCU_______________CAACGAGAUCCAUGC___A__GAUCGUCGAUCCGUGGUCACCAGCUAAUG____UGUAUAAAAUUAUAAAUACAUGC .........((((((((..((((..................(.....)..............((((...))))..))))..))))))))......(((((..........)))))... (-13.34 = -12.98 + -0.36)


| Location | 4,895,553 – 4,895,645 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 77.28 |
| Mean single sequence MFE | -34.57 |
| Consensus MFE | -17.41 |
| Energy contribution | -17.19 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.16 |
| Mean z-score | -4.00 |
| Structure conservation index | 0.50 |
| SVM decision value | 2.83 |
| SVM RNA-class probability | 0.997308 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4895553 92 - 27905053 GCAUGUAUUUAUAAUUUUAUACA----CAUUAGCUGGUGACCACCGAUCGACGAUC--U---GCAUGGAUAUUGGUC---------------AGCUGGUGGCCAGCUAAAAGAACC-- ...(((((..(....)..)))))----..(((((((((.(((((.((((((..(((--(---....)))).))))))---------------.).)))).))))))))).......-- ( -31.30) >DroPse_CAF1 30450 115 - 1 GCAUGUAUUUAUAAUUUUAUACAUCCUCAUGGCCUGGC-CCCACGCACCGAUGAUA--UCAUGAUAUGAUCGGGAUGAAGAAGAUAAGCGAAAGGUGGGCGCCAGUUGAGAGAACCCA ...(((((..(....)..)))))((((((....(((((-(((((...(((((.(((--(....)))).)))))...............(....)))))).))))).)))).))..... ( -37.30) >DroSec_CAF1 3585 93 - 1 GCAUGUAUUUAUAAUUUUAUACA----CAUUAGCUGGUCACCACUGAUGG-UGAUGGAU---GCAUGGAUGUCGAUC---------------AGCUGGUGGCCAGCUAAAAGAACC-- ...(((((..(....)..)))))----..(((((((((((((((((((.(-(.(((...---.))).)....).)))---------------)).)))))))))))))).......-- ( -33.30) >DroSim_CAF1 3518 92 - 1 GCAUGUAUUUAUAAUUUUAUACA----CAUUAGCUGGUUACCACUGAUCGACGAUC--A---GCAUGGAUGUCGUUC---------------AGCUGGUGGCCAGCUAAAAGAACC-- ...(((((..(....)..)))))----..((((((((((((((((((.((((.(((--.---.....))))))).))---------------)).)))))))))))))).......-- ( -37.60) >DroEre_CAF1 3129 92 - 1 GCAUGUAUUUAUAAUUUUAUACA----CAUUAGCUGGUGACCACGGAUCGACGAUC--A---GCUAGGAUCUCGUUG---------------CGCUGGUUGCCAGCUAAAAGAACC-- ...(((((..(....)..)))))----..((((((((..((((((.(.(((.((((--.---.....))))))).).---------------)).))))..)))))))).......-- ( -34.50) >DroYak_CAF1 3626 92 - 1 GCAUGUAUUUAUAAUUUUAUACA----CAUUAGCUGGUGACCACGGAUCGACGAUC--U---GCAACGAUCUCGCUG---------------CGCUGGUGGCCAGCUAAAAGAACC-- ...(((((..(....)..)))))----..(((((((((.((((((.(.(((.((((--.---.....))))))).).---------------)).)))).))))))))).......-- ( -33.40) >consensus GCAUGUAUUUAUAAUUUUAUACA____CAUUAGCUGGUGACCACGGAUCGACGAUC__U___GCAAGGAUCUCGAUC_______________AGCUGGUGGCCAGCUAAAAGAACC__ ...(((((..(....)..)))))......(((((((((.((((....................................................)))).)))))))))......... (-17.41 = -17.19 + -0.22)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:17:17 2006