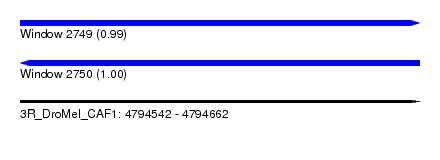
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,794,542 – 4,794,662 |
| Length | 120 |
| Max. P | 0.997691 |

| Location | 4,794,542 – 4,794,662 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.89 |
| Mean single sequence MFE | -34.92 |
| Consensus MFE | -29.95 |
| Energy contribution | -31.90 |
| Covariance contribution | 1.95 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.21 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.45 |
| SVM RNA-class probability | 0.994136 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
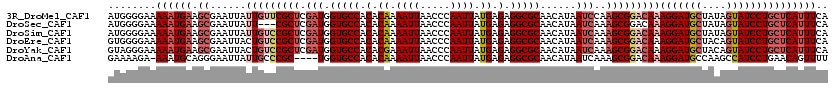
>3R_DroMel_CAF1 4794542 120 + 27905053 AUGGGGAAAAAUGAAGCGAAUUAUUGUUCGCUCGAUGGUGCCACACAAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCCAAGCGGACAAAGGAUGCUAUAGUAUCCUGCUCAUUUCA ........((((((.((......(((((((((.(((.(((((.(.((.((((.....)))).)).).)))))......)))..)))))))))(((((((....))))))))))))))).. ( -36.70) >DroSec_CAF1 29929 117 + 1 AUGGGGAAAAAUGAAGCGAAUUAUU---CGCUCGAUGGUGCCACACAAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCAAAGCGGACAAAGGAUGCUAUAGUAUCCUGCUCAUUUCA ........((((((.((......((---((((.(((.(((((.(.((.((((.....)))).)).).)))))......)))..))))))...(((((((....))))))))))))))).. ( -31.10) >DroSim_CAF1 25962 120 + 1 AUGGGGAAAAAUGAAGCGAAUUAUUGUCCGCUCGAUGGUGCCACACAAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCAAAGCGGACAAAGGAUGCUAUAGUAUCCUGCUCAUUUCA ........((((((.((......(((((((((.(((.(((((.(.((.((((.....)))).)).).)))))......)))..)))))))))(((((((....))))))))))))))).. ( -39.50) >DroEre_CAF1 30504 120 + 1 GUGGGGAAAAAUGAAGCGAAUUACUGUCCGCUCGAUGGUGCCACACAAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCAAAGCGGACAAAGGAUGCUACAGUAUCCUGCUCAUUUCA ........((((((.((.......((((((((.(((.(((((.(.((.((((.....)))).)).).)))))......)))..)))))))).(((((((....))))))))))))))).. ( -38.40) >DroYak_CAF1 30895 120 + 1 GUAGGGAAAAAUGAAGCGAAUUACUGUCCGCUCGAUGGUGCCACACGAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCAAAGCGGACAAAGGAUGCUACAGUAUCCUGCUCAUUUCA ........((((((.((.......((((((((.(((.(((((.(.((.((((.....)))).)).).)))))......)))..)))))))).(((((((....))))))))))))))).. ( -37.70) >DroAna_CAF1 29711 115 + 1 GAAAAGA-AAAUGCAGGGAAUUAUUGCCCGC----UGGUGCCACACAAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCAAAGCGGACAAAGGAUGCCAAGCCAUCCUGAACAGUUUU ......(-((((.(((((.....(((.((((----(.(((((.(.((.((((.....)))).)).).)))))...........))))).))).((........)).)))))....))))) ( -26.10) >consensus AUGGGGAAAAAUGAAGCGAAUUAUUGUCCGCUCGAUGGUGCCACACAAAAUUAACCCAAUUAUGAGAGGCGCAACAUAAUCAAAGCGGACAAAGGAUGCUAUAGUAUCCUGCUCAUUUCA ........((((((.((......(((((((((.(((.(((((.(.((.((((.....)))).)).).)))))......)))..)))))))))(((((((....))))))))))))))).. (-29.95 = -31.90 + 1.95)


| Location | 4,794,542 – 4,794,662 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.89 |
| Mean single sequence MFE | -34.67 |
| Consensus MFE | -31.54 |
| Energy contribution | -32.93 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.60 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.91 |
| SVM RNA-class probability | 0.997691 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4794542 120 - 27905053 UGAAAUGAGCAGGAUACUAUAGCAUCCUUUGUCCGCUUGGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUUGUGUGGCACCAUCGAGCGAACAAUAAUUCGCUUCAUUUUUCCCCAU .((((((((((((((.(....).)))))((((.(((((((....(((..(((..((((....))))........)))..)))...))))))).))))......)).)))))))....... ( -32.60) >DroSec_CAF1 29929 117 - 1 UGAAAUGAGCAGGAUACUAUAGCAUCCUUUGUCCGCUUUGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUUGUGUGGCACCAUCGAGCG---AAUAAUUCGCUUCAUUUUUCCCCAU .((((((((((((((.(....).)))))((((.(((((.(((..(((..(((..((((....))))........)))..)))..))))))))---.))))...)).)))))))....... ( -30.50) >DroSim_CAF1 25962 120 - 1 UGAAAUGAGCAGGAUACUAUAGCAUCCUUUGUCCGCUUUGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUUGUGUGGCACCAUCGAGCGGACAAUAAUUCGCUUCAUUUUUCCCCAU .((((((((((((((.(....).)))))((((((((((.(((..(((..(((..((((....))))........)))..)))..)))))))))))))......)).)))))))....... ( -39.20) >DroEre_CAF1 30504 120 - 1 UGAAAUGAGCAGGAUACUGUAGCAUCCUUUGUCCGCUUUGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUUGUGUGGCACCAUCGAGCGGACAGUAAUUCGCUUCAUUUUUCCCCAC .((((((((((((((.(....).)))))((((((((((.(((..(((..(((..((((....))))........)))..)))..)))))))))))))......)).)))))))....... ( -38.90) >DroYak_CAF1 30895 120 - 1 UGAAAUGAGCAGGAUACUGUAGCAUCCUUUGUCCGCUUUGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUCGUGUGGCACCAUCGAGCGGACAGUAAUUCGCUUCAUUUUUCCCUAC .((((((((((((((.(....).)))))((((((((((.(((..(((..(((..((((....))))........)))..)))..)))))))))))))......)).)))))))....... ( -38.90) >DroAna_CAF1 29711 115 - 1 AAAACUGUUCAGGAUGGCUUGGCAUCCUUUGUCCGCUUUGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUUGUGUGGCACCA----GCGGGCAAUAAUUCCCUGCAUUU-UCUUUUC ..........((((((......))))))(((((((((.......(((..(((..((((....))))........)))..)))..)----))))))))...............-....... ( -27.90) >consensus UGAAAUGAGCAGGAUACUAUAGCAUCCUUUGUCCGCUUUGAUUAUGUUGCGCCUCUCAUAAUUGGGUUAAUUUUGUGUGGCACCAUCGAGCGGACAAUAAUUCGCUUCAUUUUUCCCCAC .((((((((((((((........)))))((((((((((.(((..(((..(((..((((....))))........)))..)))..)))))))))))))......)).)))))))....... (-31.54 = -32.93 + 1.39)

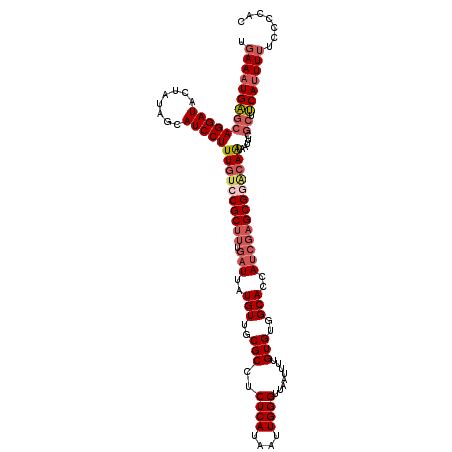

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:16:20 2006