
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,778,318 – 4,778,477 |
| Length | 159 |
| Max. P | 0.780319 |

| Location | 4,778,318 – 4,778,438 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.28 |
| Mean single sequence MFE | -46.03 |
| Consensus MFE | -40.56 |
| Energy contribution | -41.25 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.780319 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
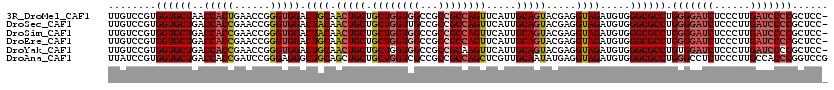
>3R_DroMel_CAF1 4778318 120 + 27905053 AUUACUCAAGUAGCGCGGAUUGAUCAUCAGACCAUUUGGAUUGUCCGUGGUGCUAACCACUGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGU ....(((.((((.(((((((.((((..((((...))))))))))))))).))))..(((((......)))))(((((((.((..((((((((...)))))))).)))))))))..))).. ( -47.90) >DroSec_CAF1 13807 120 + 1 AUUACUCAAGUAGCGCGGAUUGAUCAUCAGACCAUUCGGAUUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUACAACUGCUGCUGGUUGCCGCCGCCAGUUCAUUGCAGUACGAGGU ....(((.((((.(((((((.((((....((....)).))))))))))).))))..(((((......)))))......(((((.((((((.......)))))).....)))))..))).. ( -42.80) >DroSim_CAF1 9045 120 + 1 AUUACUCAAGUAGCGCGGAUUGAUCAUCAGACCAUUUGGAUUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUACAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGU ....(((.((((.(((((((.((((..((((...))))))))))))))).))))..(((((......)))))......(((((.((((((((...)))))))).....)))))..))).. ( -46.70) >DroEre_CAF1 13968 120 + 1 AUUGCUCAAGUAGCGCGGAUUGAUCAUCAGACCAUUCGGAUUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGU ....(((.((((.(((((((.((((....((....)).))))))))))).))))..(((((......)))))(((((((.((..((((((((...)))))))).)))))))))..))).. ( -51.00) >DroYak_CAF1 14110 120 + 1 AUUACUCAAGUAGCGCGGAUUGAUCAUCAGACCAUUUGGAUUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCAAGUUCAUUGCAGUACGAGGU ....(((.((((.(((((((.((((..((((...))))))))))))))).))))..(((((......)))))(((((((..(((((.(((....))))).)))...)))))))..))).. ( -44.90) >DroAna_CAF1 13550 120 + 1 AUUGCUGAGAUAGCGGGGAUUGAUCAUCAGACCGUUGGGAUUAUCCGUGGUGCUGACCACCGAUCCGGGAGGGCUGCAGCUGCUGCUGGUCGCCGCCGCCAGCUCGUUGCAAUAUGAGGU (((((((((...((((((...(((((.(((.(.((((((.....))(((((....)))))((..((....))..)))))).)))).))))).)).))))...))))..)))))....... ( -42.90) >consensus AUUACUCAAGUAGCGCGGAUUGAUCAUCAGACCAUUUGGAUUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGU ....(((.((((.(((((((.((((.............))))))))))).))))..(((((......)))))(((((((.((..((((((((...)))))))).)))))))))..))).. (-40.56 = -41.25 + 0.70)

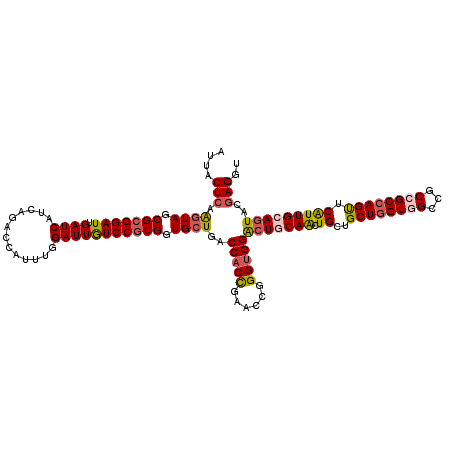

| Location | 4,778,318 – 4,778,438 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.28 |
| Mean single sequence MFE | -40.35 |
| Consensus MFE | -36.07 |
| Energy contribution | -36.35 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.26 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.650922 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4778318 120 - 27905053 ACCUCGUACUGCAAUGAACUGGCGGCGGCCACCAGCAGCAGUUGCAGUCCACCCGGUUCAGUGGUUAGCACCACGGACAAUCCAAAUGGUCUGAUGAUCAAUCCGCGCUACUUGAGUAAU ..((((((.(((..((((((((..(.(((...((((....))))..))))..))))))))(((((....)))))((.....))...(((((....)))))....))).)))..))).... ( -36.10) >DroSec_CAF1 13807 120 - 1 ACCUCGUACUGCAAUGAACUGGCGGCGGCAACCAGCAGCAGUUGUAGUCCACCCGGUUCGGUGGUCAGCACCACGGACAAUCCGAAUGGUCUGAUGAUCAAUCCGCGCUACUUGAGUAAU ..((((((.(((...(((((((.(((.(((((........))))).)))...)))))))(((.(((((.(((((((.....)))..))))))))).))).....))).)))..))).... ( -42.30) >DroSim_CAF1 9045 120 - 1 ACCUCGUACUGCAAUGAACUGGCGGCGGCCACCAGCAGCAGUUGUAGUCCACCCGGUUCGGUGGUCAGCACCACGGACAAUCCAAAUGGUCUGAUGAUCAAUCCGCGCUACUUGAGUAAU ..((((((.(((...(((((((..(.(((...((((....))))..))))..)))))))(((.(((((.((((.((.....))...))))))))).))).....))).)))..))).... ( -39.40) >DroEre_CAF1 13968 120 - 1 ACCUCGUACUGCAAUGAACUGGCGGCGGCCACCAGCAGCAGUUGCAGUCCACCCGGUUCGGUGGUCAGCACCACGGACAAUCCGAAUGGUCUGAUGAUCAAUCCGCGCUACUUGAGCAAU ..((((((.(((...(((((((..(.(((...((((....))))..))))..)))))))(((.(((((.(((((((.....)))..))))))))).))).....))).)))..))).... ( -41.70) >DroYak_CAF1 14110 120 - 1 ACCUCGUACUGCAAUGAACUUGCGGCGGCCACCAGCAGCAGUUGCAGUCCACCCGGUUCGGUGGUCAGCACCACGGACAAUCCAAAUGGUCUGAUGAUCAAUCCGCGCUACUUGAGUAAU ..((((.((((((((....((((((......)).))))..)))))))).)...(((...(((.(((((.((((.((.....))...))))))))).)))...)))........))).... ( -37.00) >DroAna_CAF1 13550 120 - 1 ACCUCAUAUUGCAACGAGCUGGCGGCGGCGACCAGCAGCAGCUGCAGCCCUCCCGGAUCGGUGGUCAGCACCACGGAUAAUCCCAACGGUCUGAUGAUCAAUCCCCGCUAUCUCAGCAAU .......(((((...(((.((((((.(((...((((....))))..))).....((((.(((.(((((.(((..((......))...)))))))).))).)))))))))).))).))))) ( -45.60) >consensus ACCUCGUACUGCAAUGAACUGGCGGCGGCCACCAGCAGCAGUUGCAGUCCACCCGGUUCGGUGGUCAGCACCACGGACAAUCCAAAUGGUCUGAUGAUCAAUCCGCGCUACUUGAGUAAU ..((((....((...(((((((..(.(((...((((....))))..))))..)))))))(((.(((((.((((.((.....))...))))))))).))).......))....)))).... (-36.07 = -36.35 + 0.28)

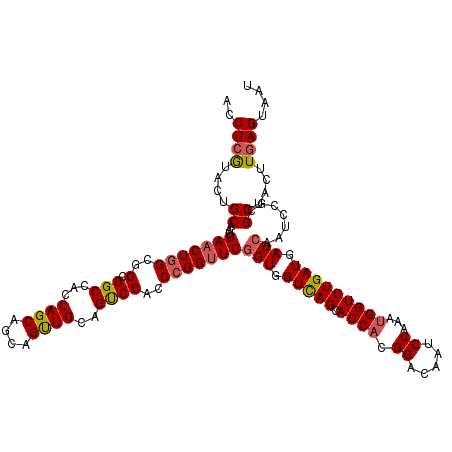
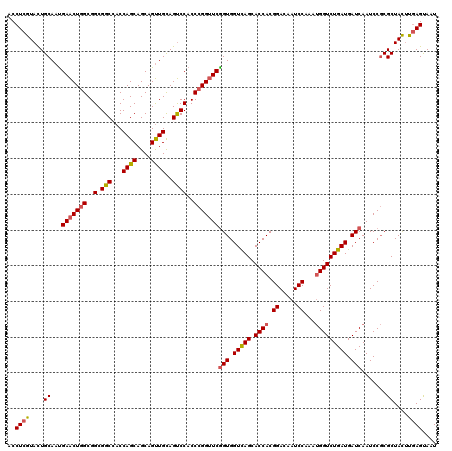
| Location | 4,778,358 – 4,778,477 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.46 |
| Mean single sequence MFE | -51.10 |
| Consensus MFE | -46.74 |
| Energy contribution | -47.60 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.09 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.628268 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4778358 119 + 27905053 UUGUCCGUGGUGCUAACCACUGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGUAGAUGUGGGCGCCUGGGGAUCUCCCUUGAUCCCCGCUCC- ..(((((((((....)))))...(((...((.(((((((.((..((((((((...)))))))).))))))))).)).)))......))))((..(((((((......)))))))))...- ( -54.40) >DroSec_CAF1 13847 119 + 1 UUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUACAACUGCUGCUGGUUGCCGCCGCCAGUUCAUUGCAGUACGAGGUAGAUGUGGGCGCCUGGGGAUCUCCCUUGAUCCCCGCUCC- ........((((((.((((((......))))).((((.(((((.((((((.......)))))).....))))).....))))...).)))))).(((((((......))))))).....- ( -49.50) >DroSim_CAF1 9085 119 + 1 UUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUACAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGUAGAUGUGGGCGCCUGGGGAUCUCCCUUGAUCCCCGCUCC- ........((((((.((((((......))))).((((.(((((.((((((((...)))))))).....))))).....))))...).)))))).(((((((......))))))).....- ( -54.30) >DroEre_CAF1 14008 119 + 1 UUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGUAGAUGUGGGCGCCUGGGGAUCUCCCUUGAUCCCCGCUCC- ........((((((.((((((......)))))(((((((.((..((((((((...)))))))).)))))))))............).)))))).(((((((......))))))).....- ( -54.90) >DroYak_CAF1 14150 119 + 1 UUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCAAGUUCAUUGCAGUACGAGGUAGAUGUGGGCGCCUGUGGAUCUCCCUUGAUCCCCGCUCC- ...((.(((((....))))).))...(((((((((((((..(((((.(((....))))).)))...))))))).(((((.(((((..(....)..)..)))).)))))....)))))).- ( -43.60) >DroAna_CAF1 13590 120 + 1 UUAUCCGUGGUGCUGACCACCGAUCCGGGAGGGCUGCAGCUGCUGCUGGUCGCCGCCGCCAGCUCGUUGCAAUAUGAGGUAGAUGUGGGCGCCUGGGCCUCUCCCUUGCCACCCGGUCCG ..(((.(((((....))))).)))(((((..(((((((((.((((.((((....)))).))))..))))))....((((.(((....(((......)))))).))))))).))))).... ( -49.90) >consensus UUGUCCGUGGUGCUGACCACCGAACCGGGUGGACUGCAACUGCUGCUGGUGGCCGCCGCCAGUUCAUUGCAGUACGAGGUAGAUGUGGGCGCCUGGGGAUCUCCCUUGAUCCCCGCUCC_ ........((((((..(((((......))))).((((.(((((.((((((((...)))))))).....))))).....)))).....)))))).(((((((......)))))))...... (-46.74 = -47.60 + 0.86)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:15:45 2006