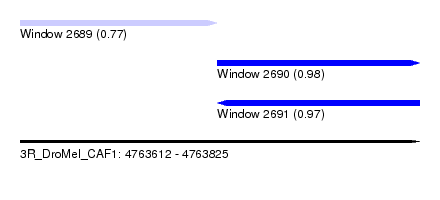
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,763,612 – 4,763,825 |
| Length | 213 |
| Max. P | 0.982637 |

| Location | 4,763,612 – 4,763,717 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 82.48 |
| Mean single sequence MFE | -37.98 |
| Consensus MFE | -23.22 |
| Energy contribution | -24.42 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.774696 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4763612 105 + 27905053 AAAAUUCGGCAUGCUCCUGUAUCACACAACCUGCCACUUGCC-CUUAAAAAGAAGGGCGCAGUGGGCGUGGUACGU--AUACAUAUGAGCCAUGCUGCAUUUUGGCCG ......((((((((....))))....(((..(((((((((((-(((......))))))).))))((((((((.(((--(....)))).)))))))))))..))))))) ( -41.10) >DroSec_CAF1 2806 103 + 1 AAAAUUCGGCAUGCUCCUGUAUCA--CAACCUGCCACUUCCC-CUUAAACAGAAGGGCGUAGUGGGCGUGGUACGU--AUACAUAUGAGCCAUGCUGCAUUUUGGCCG ......((((((((....))))..--(((..(((((((.(((-(((......))))).).))))((((((((.(((--(....)))).)))))))))))..))))))) ( -37.20) >DroSim_CAF1 2808 103 + 1 AAAAUUCGGCAUGCUCCUGUAUCA--CAACCUGCCACUUCCC-CUUAAGAAGAAGGGCGGAGUGGGCGUGGUACGU--AUACAUAUGAGCCAUGCUGCAUUUUGGCCG ......((((((((....))))..--(((..(((((((((((-(((......))))).))))))((((((((.(((--(....)))).)))))))))))..))))))) ( -41.10) >DroEre_CAF1 2754 103 + 1 AAAAUACGUCAUGCUCCUGUAUCG--CAACCUGCCACUUCCC-CUUAAUCAUAUGGGCGGAGUGGGCGUGGUCCGU--AUACAUAUGAGCCAUGCUGCAUUUUGGCCG .......(((((((....)))..(--((.....(((((((((-(..........))).)))))))((((((..(((--(....))))..)))))))))....)))).. ( -35.70) >DroYak_CAF1 2798 104 + 1 AAAAUGCGUCAUGCUCCUGUAUCG--CAACCUGCCACUUCCAACUUUAGUUUGAGGGCGGAGUGGGCGCGGUCCGU--AUACAUAUGAGCCAUGCUGGAUUUUGGCCG (((((.((.(((((((.(((((((--..(((((((..((((..((((.....))))..))))..)))).))).)).--)))))...))).)))).)).)))))..... ( -31.30) >DroAna_CAF1 2658 101 + 1 AAAGUGCUGCAUGCUCAUGUAUAG--GAAUCGGAUGCUUCCC-CCCAAG----AGUGGGUGGCUGGCGGGUUCCAUACAUACAUUUGAGUCAUGCCGCAUUUUGGUCG (((((((.((((((((((((((.(--(((((...((((..((-((((..----..)))).))..)))))))))))))))......)))).))))).)))))))..... ( -41.50) >consensus AAAAUUCGGCAUGCUCCUGUAUCA__CAACCUGCCACUUCCC_CUUAAGAAGAAGGGCGGAGUGGGCGUGGUACGU__AUACAUAUGAGCCAUGCUGCAUUUUGGCCG (((((.((((((((((.(((((......((((((((((.(((.(((......))))).).))).)))).)))......)))))...))).))))))).)))))..... (-23.22 = -24.42 + 1.20)

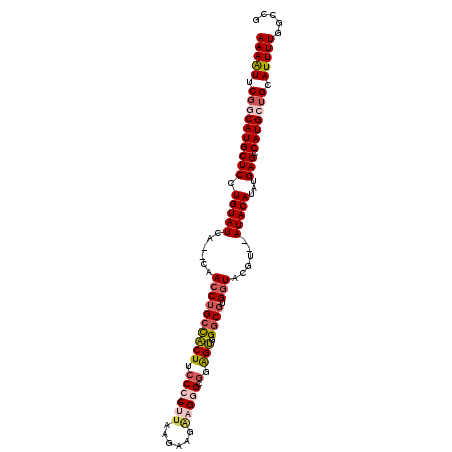

| Location | 4,763,717 – 4,763,825 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 79.11 |
| Mean single sequence MFE | -31.08 |
| Consensus MFE | -30.26 |
| Energy contribution | -29.43 |
| Covariance contribution | -0.83 |
| Combinations/Pair | 1.30 |
| Mean z-score | -3.25 |
| Structure conservation index | 0.97 |
| SVM decision value | 1.92 |
| SVM RNA-class probability | 0.982637 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4763717 108 + 27905053 UA--AUGAAAAUGCAC-UGCUUUACUUGGAAAAUUCGCUUGCACAAAAUCAACGCCGCGGCUGAUUUAUUAU-UGAUGUGCUCAAGCAA-AUUCAAGUGAAGCAUUUGCGCGA ..--........(((.-(((((((((((((...((.(((((((((((((((..((....)))))))).....-...)))))..)))).)-))))))))))))))..))).... ( -32.00) >DroVir_CAF1 3385 107 + 1 UC--AUGAAAAUGCAUUUGCUUCACUUAUAG-CCUGGCUUGCACAAAAUCAAUACCGCGGUUGAUUUUAU---AAAUGUGUUUAGGUCAAUUUUAAGUGAAACAAUUGCCUUA ..--........(((.(((.((((((((...-..(((((((((((((((((((......))))))))...---...)))))..))))))....)))))))).))).))).... ( -28.70) >DroPse_CAF1 3214 111 + 1 UAGACUGAAAAUGCACUUGCUUCACUUUAAGCCUUCGCUUGCACAGAAUCAACGCCGCGGCUGAUUUAUUAU-UAAUGUGUUUAAGCAA-AUUCAAGUGAAGCAUUUGCGCAA ............(((..((((((((((.((......(((((((((((((((..((....)))))))).....-...)))))..))))..-.)).))))))))))..))).... ( -30.70) >DroGri_CAF1 4044 110 + 1 CC--AUGAAAAUGCAUUUGUUUCACUUAAUG-CUUGGCUUGCACAGAAUCAAUACCGCGGUUGAUUUCCUUUAAAUUGUGCUUAAGCUUUAUUUAAGUGAAACAUUUGCACAA ..--.......((((..((((((((((((..-...((((((((((((((((((......)))))))).........)))))..)))))....))))))))))))..))))... ( -36.90) >DroAna_CAF1 2759 108 + 1 CG--AUGAAAAUGCAUUUGCUUCCCUUGAAGCACUCGCUUGCACAAAAUCAACGCCGCGGCUGAUUUAUUAU-UGAUGUGC-CAAGCAA-UUUCAAGUGAAACAUUUGUGAGA .(--(((......))))((((((....))))))((((((((((((((((((..((....)))))))).....-...)))).-)))))..-...((((((...)))))).))). ( -27.50) >DroPer_CAF1 2884 111 + 1 UAGACUGAAAAUGCACUUGCUUCACUUUAAGCCUUCGCUUGCACAGAAUCAACGCCGCGGCUGAUUUAUUAU-UAAUGUGUUUAAGCAA-AUUCAAGUGAAGCAUUUGCGCAA ............(((..((((((((((.((......(((((((((((((((..((....)))))))).....-...)))))..))))..-.)).))))))))))..))).... ( -30.70) >consensus UA__AUGAAAAUGCACUUGCUUCACUUAAAGCCUUCGCUUGCACAAAAUCAACGCCGCGGCUGAUUUAUUAU_UAAUGUGCUUAAGCAA_AUUCAAGUGAAACAUUUGCGCAA ............(((..(((((((((((((......(((((((((((((((..((....)))))))).........)))))..))))....)))))))))))))..))).... (-30.26 = -29.43 + -0.83)
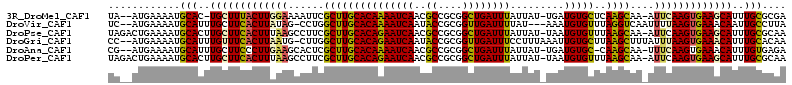
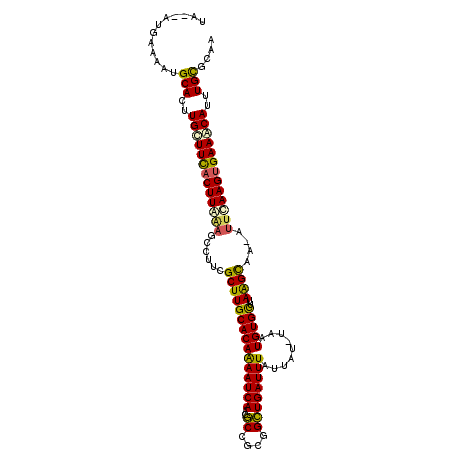
| Location | 4,763,717 – 4,763,825 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 79.11 |
| Mean single sequence MFE | -31.18 |
| Consensus MFE | -24.49 |
| Energy contribution | -25.77 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.71 |
| SVM RNA-class probability | 0.973572 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4763717 108 - 27905053 UCGCGCAAAUGCUUCACUUGAAU-UUGCUUGAGCACAUCA-AUAAUAAAUCAGCCGCGGCGUUGAUUUUGUGCAAGCGAAUUUUCCAAGUAAAGCA-GUGCAUUUUCAU--UA ..((((...(((((.((((((((-((((((..(((((...-.....((((((((......))))))))))))))))))))))...))))).)))))-))))........--.. ( -34.80) >DroVir_CAF1 3385 107 - 1 UAAGGCAAUUGUUUCACUUAAAAUUGACCUAAACACAUUU---AUAAAAUCAACCGCGGUAUUGAUUUUGUGCAAGCCAGG-CUAUAAGUGAAGCAAAUGCAUUUUCAU--GA ....(((.((((((((((((...................(---(((((((((((....))..))))))))))..(((...)-)).)))))))))))).)))........--.. ( -27.40) >DroPse_CAF1 3214 111 - 1 UUGCGCAAAUGCUUCACUUGAAU-UUGCUUAAACACAUUA-AUAAUAAAUCAGCCGCGGCGUUGAUUCUGUGCAAGCGAAGGCUUAAAGUGAAGCAAGUGCAUUUUCAGUCUA .(((((...((((((((((...(-((((((...((((...-......(((((((......))))))).)))).)))))))......)))))))))).)))))........... ( -32.60) >DroGri_CAF1 4044 110 - 1 UUGUGCAAAUGUUUCACUUAAAUAAAGCUUAAGCACAAUUUAAAGGAAAUCAACCGCGGUAUUGAUUCUGUGCAAGCCAAG-CAUUAAGUGAAACAAAUGCAUUUUCAU--GG ..(((((..((((((((((((.....((((..(((((........(((.(((((....))..)))))))))))))))....-..))))))))))))..)))))......--.. ( -28.70) >DroAna_CAF1 2759 108 - 1 UCUCACAAAUGUUUCACUUGAAA-UUGCUUG-GCACAUCA-AUAAUAAAUCAGCCGCGGCGUUGAUUUUGUGCAAGCGAGUGCUUCAAGGGAAGCAAAUGCAUUUUCAU--CG .........((((((.((((((.-(((((((-.((((...-.....((((((((......)))))))))))))))))))....)))))).)))))).............--.. ( -31.00) >DroPer_CAF1 2884 111 - 1 UUGCGCAAAUGCUUCACUUGAAU-UUGCUUAAACACAUUA-AUAAUAAAUCAGCCGCGGCGUUGAUUCUGUGCAAGCGAAGGCUUAAAGUGAAGCAAGUGCAUUUUCAGUCUA .(((((...((((((((((...(-((((((...((((...-......(((((((......))))))).)))).)))))))......)))))))))).)))))........... ( -32.60) >consensus UUGCGCAAAUGCUUCACUUGAAU_UUGCUUAAACACAUUA_AUAAUAAAUCAGCCGCGGCGUUGAUUCUGUGCAAGCGAAGGCUUAAAGUGAAGCAAAUGCAUUUUCAU__UA ....(((..(((((((((((((..((((((...((((..........(((((((......))))))).)))).))))))....)))))))))))))..)))............ (-24.49 = -25.77 + 1.28)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:15:23 2006