
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,715,995 – 4,716,116 |
| Length | 121 |
| Max. P | 0.858708 |

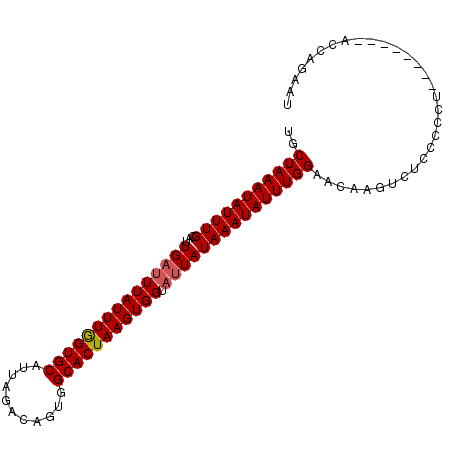
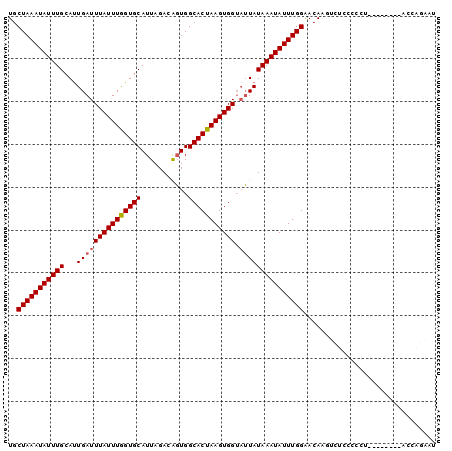
| Location | 4,715,995 – 4,716,087 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 84.08 |
| Mean single sequence MFE | -20.22 |
| Consensus MFE | -18.47 |
| Energy contribution | -18.67 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.858708 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4715995 92 - 27905053 UGCUAAAUAUUUGCAUUGAUUUAUUUAGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGAACAAGUCUCCCACU--------ACCAGAAU ..(((((((((((...((((((((((((((((((....))).))))))))))).)))))))))))))))...............--------........ ( -19.70) >DroSec_CAF1 61195 92 - 1 UGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGAACAAGUCUACCCCC--------AGCAGAAU ((((.......(((((..(.....)..))))).(((((..((...(((((((..........)))))))..)).))))).....--------)))).... ( -19.60) >DroSim_CAF1 64670 78 - 1 UGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGAACAA----------------------GAAU ..(((((((((((...((((((((((((((((((....))).))))))))))).))))))))))))))).....----------------------.... ( -19.40) >DroEre_CAF1 64926 92 - 1 UGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGAACAAGUCUCACCCU--------ACCGGAAU ..(((((((((((...((((((((((((((((((....))).))))))))))).)))))))))))))))............((.--------...))... ( -19.90) >DroWil_CAF1 65770 100 - 1 UGCUAAAUAUUUGCACUGAUUUAUUUGGUGCAUUAGAAGUUGGCACUAAGUGGAUAUAUAAAUAUUUGGGACAUGGCUAUAUAUUAUUUUUUACAUACAU ..(((((((((((.....(((((((((((((...........)))))))))))))...)))))))))))............................... ( -21.50) >DroYak_CAF1 64549 92 - 1 UGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGAACAGGUCUCUCCCU--------ACCAGAAU ..(((((((((((...((((((((((((((((((....))).))))))))))).)))))))))))))))....(((........--------)))..... ( -21.20) >consensus UGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGAACAAGUCUCCCCCU________ACCAGAAU ..(((((((((((...(((((((((((((((...........))))))))))).)))))))))))))))............................... (-18.47 = -18.67 + 0.20)

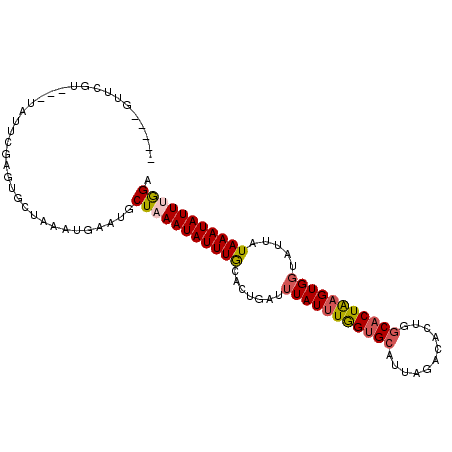
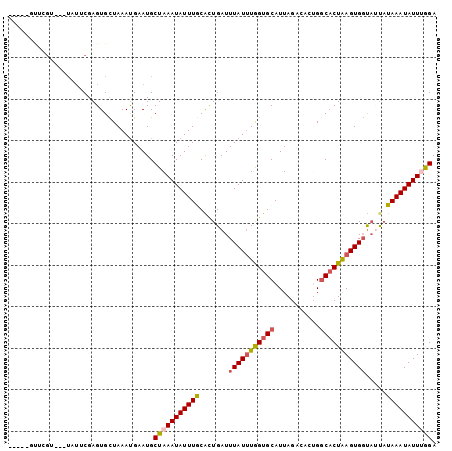
| Location | 4,716,017 – 4,716,116 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 80.63 |
| Mean single sequence MFE | -22.98 |
| Consensus MFE | -15.19 |
| Energy contribution | -15.47 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831125 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4716017 99 - 27905053 U----GUAUGUUUGUAGCCGAGUGCUAAAUGAAUGCUAAAUAUUUGCAUUGAUUUAUUUAGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGA .----..((((((((((((((((((((......((((((.....((((((((.....)))))))))))).)).)))))))...))))..)))))))))..... ( -25.40) >DroVir_CAF1 84158 99 - 1 AUAACGCUCGU---UUUUCGAGUGUUAAAUGAAUGCUAAAUAUUUACGCUGAUUUAUUUGGUGCAUUAGAACCGGGCACUAAGUGCUA-UAUAAAUAUUAAGA .(((((((((.---....)))))))))........((.(((((((((((..(.....)..)))...........(((((...))))).-..)))))))).)). ( -25.10) >DroEre_CAF1 64948 98 - 1 -----GUUGGUUUGUAUGCGAGUGCUAAAUGAAUGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGA -----(((.((((((((....)))).)))).))).(((((((((((...((((((((((((((((((....))).))))))))))).))))))))))))))). ( -23.90) >DroYak_CAF1 64571 99 - 1 C----GUAUGUUUGUAUGCGAGUGCUAAAUGAAUGCUAAAUAUUUGCAUUGAUUUAUUUGGUGCAUUAGACAGUGGCACUAAGUGGUAUUAUAAAUAUUUGGA (----(.((((((((((((.(((((((......((((((.....(((((..(.....)..))))))))).)).))))))).....))).))))))))).)).. ( -24.60) >DroMoj_CAF1 80331 94 - 1 -----ACUCGU---UUUUCGAGUGCUAAAUGAAUGCUAAAUAUUUACGCUGAUUUAUUUGGUGCAUUAGAACCGGGCACUGAGUGGCA-UAUAAAUAUUGAGA -----(((((.---....)))))............((.((((((((.(((.....(((..((((...........))))..)))))).-..)))))))).)). ( -19.50) >DroAna_CAF1 61876 88 - 1 ---------------GUUCGAGUGCUAAACGAAUGCUAAAUAUUUGCACUGAUUUAUAUGGUGCAUUAGACCCUGACUCUAAGUGGUUUCUUAAAUAUUUGGA ---------------(((((.........))))).(((((((((((((((.........)))))...(((((((.......)).)))))...)))))))))). ( -19.40) >consensus _____GUUCGU___UAUUCGAGUGCUAAAUGAAUGCUAAAUAUUUGCACUGAUUUAUUUGGUGCAUUAGACACUGGCACUAAGUGGUAUUAUAAAUAUUUGGA ...................................(((((((((((.......(((((((((((...........))))))))))).....))))))))))). (-15.19 = -15.47 + 0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:15:08 2006