| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 762,978 – 763,095 |
| Length | 117 |
| Max. P | 0.918585 |

| Location | 762,978 – 763,095 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 76.63 |
| Mean single sequence MFE | -45.28 |
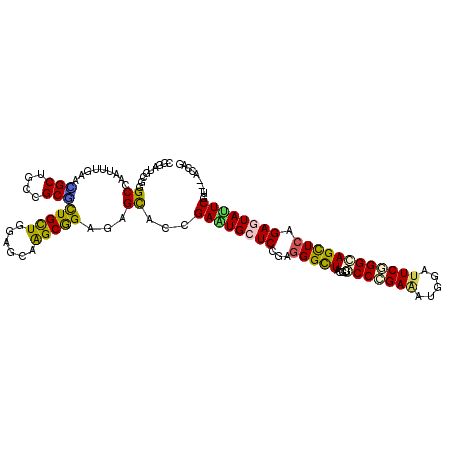
| Consensus MFE | -27.61 |
| Energy contribution | -26.70 |
| Covariance contribution | -0.91 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.918585 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
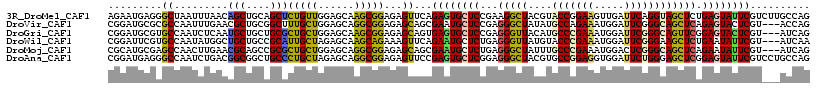
>3R_DroMel_CAF1 762978 117 + 27905053 AGAAUGAGGGCUAAUUUAACAGCUGCAGCUCUGUUGGAGCAAGCGGAGAGUUCAGAGUGCUCCGAAGGCUACGUACCGGAAGUUGAUUCAGGUAGCUCUGAGUAUUCGUCUUGCCAG ........(((..........(((.((((...)))).)))(((((((...(((((((((((.....)))....((((.(((.....))).))))))))))))..)))).)))))).. ( -35.70) >DroVir_CAF1 24214 114 + 1 CGGAUGCGCGCCAAUUUGAACGCUGCGGCUUUGCUGGAGCAGGCGGAGAGCAGCGAAUGCUCCGAGGGCUAUAUGCCAGAAAUGGAUUCGGGCAGCUCAGAGUACUCGU---ACCAG .((.((((.(..........((((((..(((((((......))))))).))))))..(((((.(((.(((.....(((....))).....)))..))).)))))).)))---))).. ( -45.70) >DroGri_CAF1 22439 114 + 1 CGGAUGCGUGCCAAUCUCAAUGCUGCUGCGCUGCUGGAGCAAGCGGAGAGCAGUGAGUGCUCCGAGGGUUACAUGCCCGAAAUGGAUUCGGGCAGUUCGGAGUACUCGU---AUCAG ..(((((..............((((((.(.(((((......))))).)))))))(((((((((((((....).((((((((.....)))))))).))))))))))))))---))).. ( -55.00) >DroWil_CAF1 21907 114 + 1 CGGAUUCGUGCCAAUAUGGCUGCUGCCGCAUUGCUAGAGCAAGCAGAAAGUUCAGAAUGCUCUGAGGGUUAUGUACCCGAAAUGGAUUCGGGAAGCUCUGAAUAUUCGU---AUCAA ..(((.((.((.(((.((((....)))).)))))((((((..(((.((..((((((....))))))..)).))).((((((.....))))))..))))))......)).---))).. ( -35.60) >DroMoj_CAF1 22100 114 + 1 CGCAUGCGAGCCAACUUGAACGCAGCCGCGCUGCUGGAGCAGGCGGAGAGCAGCGAAUGCUCUGAGGGCUAUUUGCCCGAAAUGGACUCGGGCAGCUCAGAAUAUUCGU---AUCAG .((.((((..(......)..)))).((((.((((....))))))))...)).(((((((.((((((......((((((((.......)))))))))))))).)))))))---..... ( -47.50) >DroAna_CAF1 20159 117 + 1 CGGAUGAGGGCCAAUCUGACGGCGGCUGCCCUGCUAGAGCAGGCGGAGAGUUCCGAGUGCUCGGAGGGCUACGUGCCGGAGGUGGAUUCUGGGAGCUCGGAGUAUUCGUCCUGCCAG .(((((((..(((..((..((((((..(((((.((.(((((..(((......)))..))))))))))))..).)))))..)))))(((((((....))))))).)))))))...... ( -52.20) >consensus CGGAUGCGGGCCAAUUUGAACGCUGCCGCGCUGCUGGAGCAAGCGGAGAGCACCGAAUGCUCCGAGGGCUACGUGCCCGAAAUGGAUUCGGGCAGCUCAGAGUAUUCGU___ACCAG .........((.........(((....)))(((((......)))))...))...((((((((...(((((....(((((((.....)))))))))))).)))))))).......... (-27.61 = -26.70 + -0.91)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:34:24 2006