
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 4,214,201 – 4,214,353 |
| Length | 152 |
| Max. P | 0.975600 |

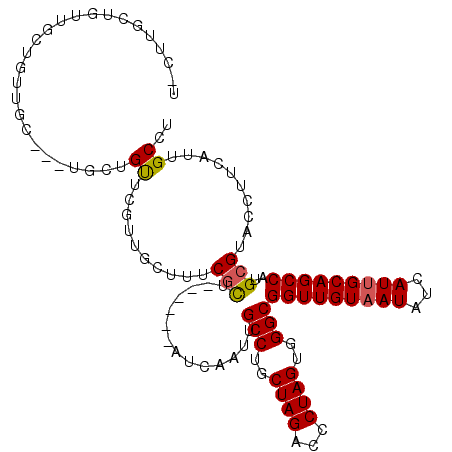
| Location | 4,214,201 – 4,214,316 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 79.03 |
| Mean single sequence MFE | -35.93 |
| Consensus MFE | -23.74 |
| Energy contribution | -23.60 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.84 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975600 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4214201 115 + 27905053 U-CUUGUUGUUGCUGUUGCGUUAGCUGUUCGUUGGAUUCGCUAGUGGUAUCAAUUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCU .-...((((.(((....))).)))).((.((((((....((.((((((((((((((((..((((...))))..))))))))..))))))))))..))).))).))........... ( -36.90) >DroVir_CAF1 43658 107 + 1 AGCUUGCUGUUGUUGAUAC---UGAAGCUCGUUGCUUUCGUU------AUCAACUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCU .((..(((((...((((..---.(((((.....))))).(((------(.((((((((..((((...))))..))))))))))))))))..)))))...))............... ( -38.90) >DroGri_CAF1 32646 107 + 1 UUCUGGCUGUGGUUGAUAG---UGAAGCGCGUUGCUUUCGUU------AUCAACUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCU ...((((((..((.(((..---.(((((.....))))).(((------(.((((((((..((((...))))..)))))))))))))))))..)))))).(((.........))).. ( -41.50) >DroEre_CAF1 26099 115 + 1 U-GUUGCUGUUGCUGUUGCGUUAGCUGUUCGUUGGAUUCGCUAGUGGUAUCAAUUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCU .-...((((.(((....))).)))).((.((((((....((.((((((((((((((((..((((...))))..))))))))..))))))))))..))).))).))........... ( -39.30) >DroWil_CAF1 30716 88 + 1 -------------------------UGUUCCGUACUUUCGUUAC---UAUCAAUUGCCUGCUAGACUCUAGUGGGCGGUUGUAAUAUCAUAGCAGCCAUGCGUACCUUCAUUGCCU -------------------------......((((.........---...((((((((..((((...))))..))))))))..........(((....)))))))........... ( -24.50) >DroAna_CAF1 27957 109 + 1 U-GUUCGUGUUUGUGUUGCGUUUGCUGUUCGUUGGUAUCACU------AUCAAUUGCCUGCUAGACUCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCU .-....(.((..(((.(((((..(((((....((((((....------..((((((((..((((...))))..))))))))..))))))..)))))...)))))....))).))). ( -34.50) >consensus U_CUUGCUGUUGCUGUUGC___UGCUGUUCGUUGCUUUCGCU______AUCAAUUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCU ..........................((..........(((..............(((..((((...))))..)))(((((((((...)))))))))..)))..........)).. (-23.74 = -23.60 + -0.14)


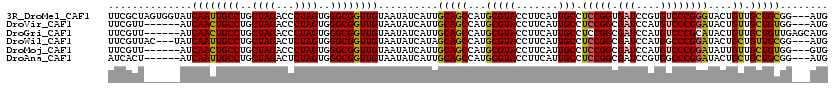
| Location | 4,214,236 – 4,214,353 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.53 |
| Mean single sequence MFE | -41.08 |
| Consensus MFE | -34.00 |
| Energy contribution | -33.50 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.865784 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 4214236 117 + 27905053 UUCGCUAGUGGUAUCAAUUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGUGAUCCGUGUCCCGGGUACUGUUGCUGCGG---AUG (((((.((((((((((((((((..((((...))))..))))))))..))))))))(((((..((((((.......)))..((((.(((....))))))))))..))))).))))---).. ( -44.60) >DroVir_CAF1 43691 111 + 1 UUCGUU------AUCAACUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGUCCCGGAUACUGUUGCUGUGG---AUG ...(((------(.((((((((..((((...))))..)))))))))))).((((.(((((...(((.........))).(((((.(((....))))))))....))))).))))---... ( -41.00) >DroGri_CAF1 32679 114 + 1 UUCGUU------AUCAACUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGUCCCGCAUACUGUUGCUGUUGAGCAUG ...(((------(.((((((((..((((...))))..)))))))))))).(((..(((((.(((((.((....((((((...))))))....))..)))))...)))))...)))..... ( -40.80) >DroWil_CAF1 30727 114 + 1 UUCGUUAC---UAUCAAUUGCCUGCUAGACUCUAGUGGGCGGUUGUAAUAUCAUAGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGGCCCGGAUACUGCUGUUGCGG---AUG (((((.((---...((((((((..((((...))))..)))))))).........(((((..((.((..((...((((((...))))))....))..)).)).))))))).))))---).. ( -40.30) >DroMoj_CAF1 32429 111 + 1 UUCGUU------AUCAACUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGUCCCGGAUAUUGUUGCUGUGG---GUG ...(((------(.((((((((..((((...))))..)))))))))))).((((.(((((...(((.........))).(((((.(((....))))))))....))))).))))---... ( -41.00) >DroAna_CAF1 27992 111 + 1 AUCACU------AUCAAUUGCCUGCUAGACUCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCGUGGCCCGGAUACUGCUGCUGCGG---AUG (((...------..((((((((..((((...))))..)))))))).........((((((...(((((......(((((...)))))((((.....))))))).)).)))))))---)). ( -38.80) >consensus UUCGUU______AUCAACUGCCUGCUAGACCCUAGUGGGCGGUUGUAAUAUCAUUGCAGCCAUGCGUACCUUCAUUGCCUCCGGCGAUCCAUGUCCCGGAUACUGUUGCUGCGG___AUG ..............((((((((..((((...))))..))))))))..........(((((...(((((.......))).(((((.(((....))))))))....)).)))))........ (-34.00 = -33.50 + -0.50)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:16 2006