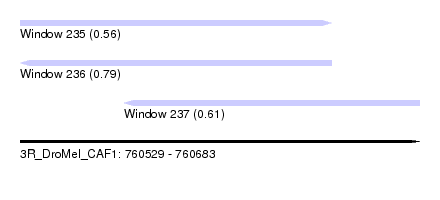
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 760,529 – 760,683 |
| Length | 154 |
| Max. P | 0.792984 |

| Location | 760,529 – 760,649 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.67 |
| Mean single sequence MFE | -41.60 |
| Consensus MFE | -23.88 |
| Energy contribution | -23.72 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.561208 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
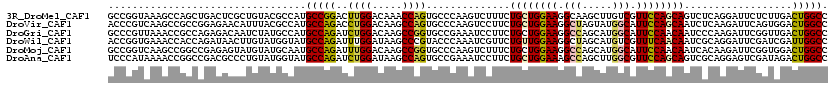
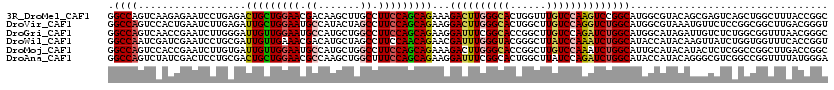
>3R_DroMel_CAF1 760529 120 + 27905053 GCCGGUAAAGCCAGCUGACUCGCUGUACGCCAUGCCGGACUUGGACAAACCAGUGCCCAAGUCUUUCUGCUGGAAGGCAAGCUUGUCGUUCCAGCAGUCUCAGGAUUCUCUUGACUGGCC (((((((..((((((......))))...))..))))((((((((.((......)).))))))))..(((((((((((((....)))).)))))))))..(((((.....)))))..))). ( -48.10) >DroVir_CAF1 21744 120 + 1 ACCCGUCAAGCCGCCGGAGAACAUUUACGCCAUGCCAGACCUGGACAAGCCAGUGCCCAAGUCCUUCUGCUGGAAGGCUAGUAUGGCAUUCCAGCAAUCUCAAGAUUCAGUGGACUGGCC ............(((((...........(((((((..(..((((.....))))..)...((.(((((.....))))))).)))))))..((((..((((....))))...))))))))). ( -36.30) >DroGri_CAF1 19942 120 + 1 GCCCGUUAAACCGCCAGAGACAAUCUAUGCCAUGCCAGAUCUGGACAAGCCGGUGCCGAAAUCCUUCUGCUGGAAGGCCAGCAUGGCAUUCCAACAAUCCCAAGAUUCGGUUGACUGGCC (((.(((((.(((..(((.....)))(((((((((.....((((.....)))).(((....(((.......))).)))..)))))))))..................)))))))).))). ( -37.40) >DroWil_CAF1 19449 120 + 1 ACCGGUGAAACCACCAGAUAACUUGUAUGGUAUGCCAGAUUUGGAUAAGCCCGUACCCAAAUCGUUCUGUUGGAAGGCUAGCAUGUCGUUUCAACAAUCGCAGGAUUCGAUCGAUUGGCC .((((((....)))).(((..(((((.(((....)))(((((((.((......)).)))))))....(((((((((((......))).))))))))...))))).....)))....)).. ( -32.30) >DroMoj_CAF1 19621 120 + 1 GCCGGUCAAGCCGGCCGAGAGUAUGUAUGCAAUGCCAGAUUUGGACAAGCCGGUGCCCAAGUCUUUCUGCUGGAAGGCCAGCAUGGCAUUCCAACAAUCACAAGAUUCGGUGGACUGGCC (((((((..(((((.(....)..(((.((.((((((((((((((.((......)).)))))))....((((((....))))))))))))).)))))..........))))).))))))). ( -47.40) >DroAna_CAF1 17698 120 + 1 UCCCAUAAAACCGGCCGACGCCCUGUAUGGUAUGCCAGAUCUGGAUAAGCCAGUGCCGAAAUCCUUCUGCUGGAAAGCCAGCUUGGCGUUCCAGCAGUCGCAGGAGUCGAUAGACUGGCC ..(((((.....(((....)))...)))))...(((((..((((.....))))...(((..((((.(((((((((.(((.....))).)))))))))....)))).))).....))))). ( -48.10) >consensus GCCCGUAAAACCGCCAGAGACCAUGUAUGCCAUGCCAGAUCUGGACAAGCCAGUGCCCAAAUCCUUCUGCUGGAAGGCCAGCAUGGCAUUCCAACAAUCGCAAGAUUCGGUGGACUGGCC .................................(((((..((((.....))))..............((((((((.(((.....))).))))))))..................))))). (-23.88 = -23.72 + -0.16)
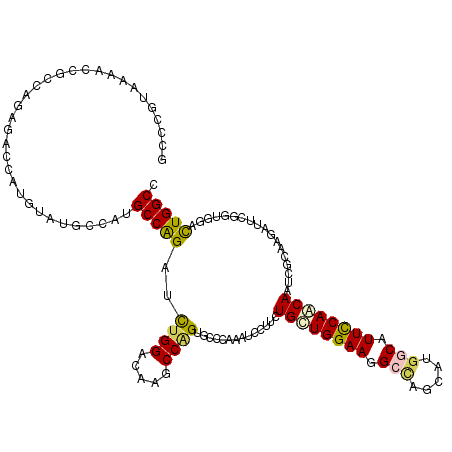
| Location | 760,529 – 760,649 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.67 |
| Mean single sequence MFE | -44.05 |
| Consensus MFE | -29.33 |
| Energy contribution | -29.25 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.792984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
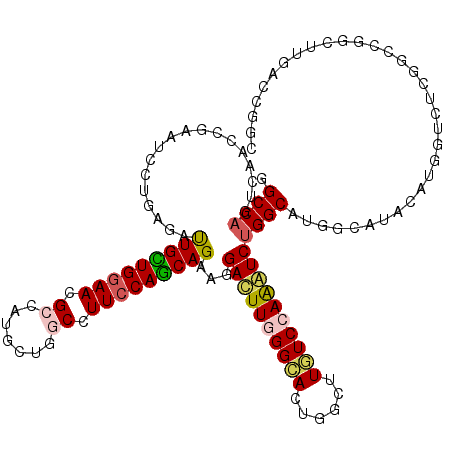
>3R_DroMel_CAF1 760529 120 - 27905053 GGCCAGUCAAGAGAAUCCUGAGACUGCUGGAACGACAAGCUUGCCUUCCAGCAGAAAGACUUGGGCACUGGUUUGUCCAAGUCCGGCAUGGCGUACAGCGAGUCAGCUGGCUUUACCGGC .(((((((...............(((((((((.(.((....))).)))))))))...((((((((((......)))))))))).))).))))(((.(((.((....)).))).))).... ( -48.50) >DroVir_CAF1 21744 120 - 1 GGCCAGUCCACUGAAUCUUGAGAUUGCUGGAAUGCCAUACUAGCCUUCCAGCAGAAGGACUUGGGCACUGGCUUGUCCAGGUCUGGCAUGGCGUAAAUGUUCUCCGGCGGCUUGACGGGU .(((.(((.....((((....))))(((((((((((((.(((((((((.....)))))(((((((((......))))))))))))).)))))))........)))))).....))).))) ( -45.10) >DroGri_CAF1 19942 120 - 1 GGCCAGUCAACCGAAUCUUGGGAUUGUUGGAAUGCCAUGCUGGCCUUCCAGCAGAAGGAUUUCGGCACCGGCUUGUCCAGAUCUGGCAUGGCAUAGAUUGUCUCUGGCGGUUUAACGGGC .(((.((((.(((.((((.(((...(((((..((((.((((((....))))))(((....))))))))))))...))))))).)))..)))).((((((((.....))))))))...))) ( -43.90) >DroWil_CAF1 19449 120 - 1 GGCCAAUCGAUCGAAUCCUGCGAUUGUUGAAACGACAUGCUAGCCUUCCAACAGAACGAUUUGGGUACGGGCUUAUCCAAAUCUGGCAUACCAUACAAGUUAUCUGGUGGUUUCACCGGU .(((...((((((.......)))))).((((((.........((((((.....))).((((((((((......)))))))))).))).(((((...........)))))))))))..))) ( -31.70) >DroMoj_CAF1 19621 120 - 1 GGCCAGUCCACCGAAUCUUGUGAUUGUUGGAAUGCCAUGCUGGCCUUCCAGCAGAAAGACUUGGGCACCGGCUUGUCCAAAUCUGGCAUUGCAUACAUACUCUCGGCCGGCUUGACCGGC (((.((((..(((((((....)))(((((.((((((.((((((....))))))...(((.(((((((......))))))).))))))))).)).))).....))))..)))).).))... ( -43.70) >DroAna_CAF1 17698 120 - 1 GGCCAGUCUAUCGACUCCUGCGACUGCUGGAACGCCAAGCUGGCUUUCCAGCAGAAGGAUUUCGGCACUGGCUUAUCCAGAUCUGGCAUACCAUACAGGGCGUCGGCCGGUUUUAUGGGA (((((((...((((.((((....(((((((((.(((.....))).))))))))).))))..)))).)))))))..((((...(((((...((.....))......))))).....)))). ( -51.40) >consensus GGCCAGUCAACCGAAUCCUGAGAUUGCUGGAACGCCAUGCUGGCCUUCCAGCAGAAGGACUUGGGCACUGGCUUGUCCAAAUCUGGCAUGGCAUACAUGGUCUCGGCCGGCUUGACCGGC .((((..................(((((((((.((.......)).)))))))))...((((((((((......))))))))))))))................................. (-29.33 = -29.25 + -0.08)
| Location | 760,569 – 760,683 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 81.05 |
| Mean single sequence MFE | -43.60 |
| Consensus MFE | -30.94 |
| Energy contribution | -30.75 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.42 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.608414 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
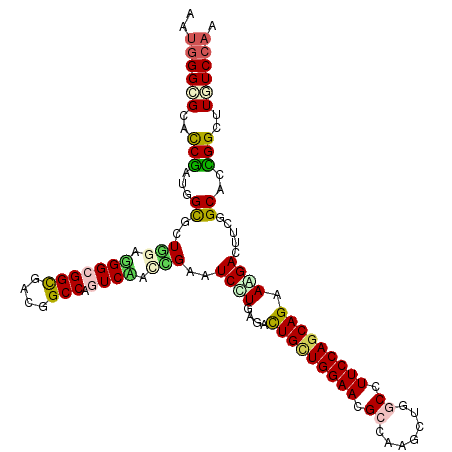
>3R_DroMel_CAF1 760569 114 - 27905053 UAUGGGAGCCCCGAUAGCACUAGAUGGCGGCGACGGCCAGUCAAGAGAAUCCUGAGACUGCUGGAACGACAAGCUUGCCUUCCAGCAGAAAGACUUGGGCACUGGUUUGUCCAA ...(((...)))(((((.(((((.(((((((....))).)))).......((..((.(((((((((.(.((....))).))))))))).....))..))..))))))))))... ( -38.10) >DroVir_CAF1 21784 114 - 1 AAUGGGCGCAUCGAUGGUGCUGGAGGGCGGUGAGGGCCAGUCCACUGAAUCUUGAGAUUGCUGGAAUGCCAUACUAGCCUUCCAGCAGAAGGACUUGGGCACUGGCUUGUCCAG ..(((((((((.....))))...(((.(((((....(((((((....((((....))))(((((((.((.......)).)))))))....)))).))).))))).)))))))). ( -45.90) >DroGri_CAF1 19982 114 - 1 AAUGGGCGCAUCGAUGGUGCUGGAGGGCGGUGAGGGCCAGUCAACCGAAUCUUGGGAUUGUUGGAAUGCCAUGCUGGCCUUCCAGCAGAAGGAUUUCGGCACCGGCUUGUCCAG ..(((((((((.....))))...(((.(((((..((....))..(((((((((....(((((((((.(((.....))).))))))))).)))).)))))))))).)))))))). ( -47.80) >DroEre_CAF1 17499 114 - 1 AAUGGGAGCCCCGACGGCGCUUGAUGGAGGCGACGGCCAGUCAAGGGAGUCCUGAGACUGCUGGAACGACAAGCUUGCCUUCCAACAGAAAGACUUCGGCACCGGUUUGUCCAA ..((((....(((...((.((((((...(((....))).))))))((((((......(((.(((((.(.((....))).))))).)))...)))))).))..)))....)))). ( -35.80) >DroMoj_CAF1 19661 114 - 1 AAUAGGCGCAUCGAUGGUACUGGAGGGCGGCGAUGGCCAGUCCACCGAAUCUUGUGAUUGUUGGAAUGCCAUGCUGGCCUUCCAGCAGAAAGACUUGGGCACCGGCUUGUCCAA ....((.(((....((((......(((((((....))).)))).((((.((((....(((((((((.(((.....))).))))))))).)))).))))..))))...))))).. ( -42.70) >DroAna_CAF1 17738 114 - 1 GACGGGUGCUCCAACGGCGCUGGAAGGAGGCGAAGGCCAGUCUAUCGACUCCUGCGACUGCUGGAACGCCAAGCUGGCUUUCCAGCAGAAGGAUUUCGGCACUGGCUUAUCCAG ..(.((((((.....)))))).)..(((.....((((((((...((((.((((....(((((((((.(((.....))).))))))))).))))..)))).)))))))).))).. ( -51.30) >consensus AAUGGGCGCACCGAUGGCGCUGGAGGGCGGCGACGGCCAGUCAACCGAAUCCUGAGACUGCUGGAACGCCAAGCUGGCCUUCCAGCAGAAAGACUUCGGCACCGGCUUGUCCAA ..((((((..(((...((..(((.(((((((....))).)))).)))..((((....(((((((((.((.......)).))))))))).)))).....))..)))..)))))). (-30.94 = -30.75 + -0.19)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:34:22 2006