
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 3,786,038 – 3,786,316 |
| Length | 278 |
| Max. P | 0.999987 |

| Location | 3,786,038 – 3,786,156 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.59 |
| Mean single sequence MFE | -59.96 |
| Consensus MFE | -50.68 |
| Energy contribution | -52.28 |
| Covariance contribution | 1.60 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.06 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.51 |
| SVM RNA-class probability | 0.999318 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3786038 118 + 27905053 AGCUGCUGCUGCGGCCGCCGCUGCC-UGCUGGUGGUGCAGGACAUGUC-CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCGUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCU ((((.((((((((.(((((((....-.)).))))))))....((((((-((....((.....))((((.(((((((((((.....))))))))))).)))).))))))))))))).)))) ( -60.40) >DroSec_CAF1 15400 115 + 1 AGCUGCUGCUG---CCGCCGCUGCC-UGCUGGUGGUGCAGGACAUGUC-CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCU ((((.((((((---(((((((....-.)).))))))......((((((-((....((.....))((((.(((((((((((.....))))))))))).)))).))))))))))))).)))) ( -61.50) >DroSim_CAF1 19151 118 + 1 AGCUGCUGCUGCGGCCGCCGCUGCC-UGCUGGUGGUGCAGGACAUGUC-CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCU ((((.((((((((.(((((((....-.)).))))))))....((((((-((....((.....))((((.(((((((((((.....))))))))))).)))).))))))))))))).)))) ( -63.80) >DroEre_CAF1 6071 115 + 1 AGCUGCUGCUGCGGCCGCCGCUGCC-UGCUGGUGGUGCAGGACAUGUC-CGUGAUGCGAGUGGUGUC---CGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGGGCGGGAGCU ((((.((((((((.(((((((....-.)).)))))))).(..((((((-((.(((((.....)))))---((((((((((.....)))))))))).))))))))..)...))))).)))) ( -58.60) >DroYak_CAF1 18368 116 + 1 NNNNNNUGCUGCGGCCGCCGCUGCCUUGCUGGUGGUGCAGGACAUGUCCCGUGAUGCGAGUGGUGUC---CGUGUGCGUGAUG-GCAUGCACAUGGUGGGCGUGGACAUGGGCGGGAGCU .......(((.(.(((((.((..((.....))..))))....(((((((((((((((.....)))))---((((((((((...-.))))))))))....))).)))))))))).).))). ( -55.50) >consensus AGCUGCUGCUGCGGCCGCCGCUGCC_UGCUGGUGGUGCAGGACAUGUC_CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCU .(((.((((((((.(((((((......)).))))))))....((((((.((.....))......((((.(((((((((((.....))))))))))).))))...))))))))))).))). (-50.68 = -52.28 + 1.60)

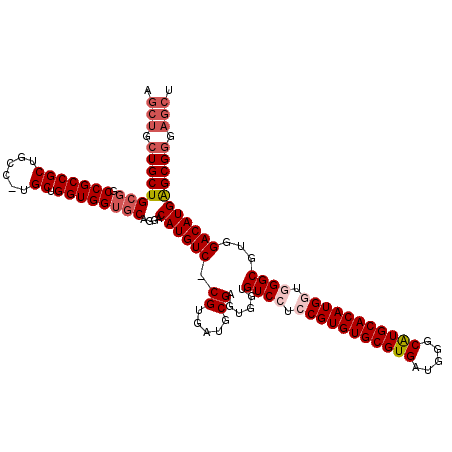
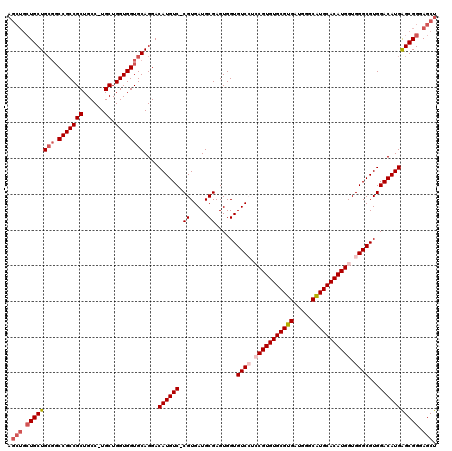
| Location | 3,786,077 – 3,786,196 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.12 |
| Mean single sequence MFE | -52.78 |
| Consensus MFE | -45.90 |
| Energy contribution | -46.86 |
| Covariance contribution | 0.96 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.60 |
| Structure conservation index | 0.87 |
| SVM decision value | 3.43 |
| SVM RNA-class probability | 0.999198 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3786077 119 + 27905053 GACAUGUC-CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCGUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGGAGGGUCGUGGCCGCCG ..((((((-(((..(((..(((.(((((.(((((((((((.....))))))))))).)))))....)))..)))...)).).))))))..(((.(((((((....))))))).))).... ( -52.90) >DroSec_CAF1 15436 119 + 1 GACAUGUC-CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGAUGAUGACUCAACAGGGUCGUGGCCGCCG ..((((((-((....((.....))((((.(((((((((((.....))))))))))).)))).))))))))..(((..((..((((.((.(((.......))).))..))))..))..))) ( -55.10) >DroSim_CAF1 19190 119 + 1 GACAUGUC-CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGAUGAUGACUCAACAGGGUCGUGGCCGCCG ..((((((-((....((.....))((((.(((((((((((.....))))))))))).)))).))))))))..(((..((..((((.((.(((.......))).))..))))..))..))) ( -55.10) >DroEre_CAF1 6110 116 + 1 GACAUGUC-CGUGAUGCGAGUGGUGUC---CGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGGGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGCAAAGUUGUGGCCGCCG ..((((((-((((((((.....))))(---((((((((((.....)))))))))))....))))))))))((((...((..((((....((.(((....))).))..))))..)))))). ( -48.90) >DroYak_CAF1 18408 116 + 1 GACAUGUCCCGUGAUGCGAGUGGUGUC---CGUGUGCGUGAUG-GCAUGCACAUGGUGGGCGUGGACAUGGGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGCAAAGUUGUGGCCGCCG ..(((((((((((((((.....)))))---((((((((((...-.))))))))))....))).)))))))((((...((..((((....((.(((....))).))..))))..)))))). ( -51.90) >consensus GACAUGUC_CGUGAUGCGAGUGGUGUCCUCCGUGUGCGUGAUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGCAGGGUCGUGGCCGCCG ..((((((..((..(((..(((.(((((.(((((((((((.....))))))))))).)))))....)))..)))...))...))))))..(((.(((((((....))))))).))).... (-45.90 = -46.86 + 0.96)


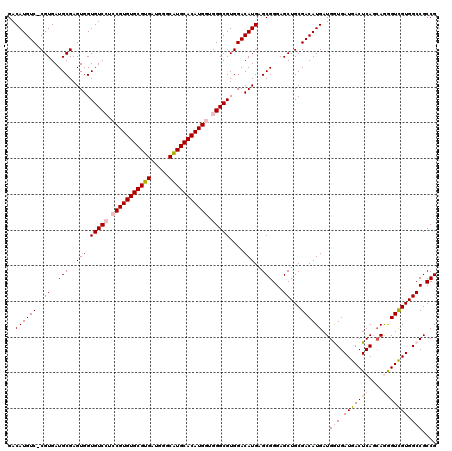
| Location | 3,786,116 – 3,786,236 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.50 |
| Mean single sequence MFE | -57.74 |
| Consensus MFE | -57.32 |
| Energy contribution | -56.04 |
| Covariance contribution | -1.28 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.72 |
| Structure conservation index | 0.99 |
| SVM decision value | 5.45 |
| SVM RNA-class probability | 0.999987 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3786116 120 + 27905053 AUGGGCGUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGGAGGGUCGUGGCCGCCGGAUUGGCAGGCGGCACCUCGUGCAUGUGCAGCGCCCGCUG (((.((((.(((...))).))))...))).((((((.((((((.(((((.(((.(((((((....))))))).((((((.........))))))))))))))....)))))).)))))). ( -60.60) >DroSec_CAF1 15475 120 + 1 AUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGAUGAUGACUCAACAGGGUCGUGGCCGCCGGAUUGGCGGGCGGUACCUCGUGCAUGUGCAGUGCCCGCUG (((..(((((.(.....).)))))..))).((((((.(((((.(((((((((.((((((((....))))))).((((((.(.....).)))))).).)))).)))))))))).)))))). ( -58.10) >DroSim_CAF1 19229 120 + 1 AUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGAUGAUGACUCAACAGGGUCGUGGCCGCCGGAUUGGCAGGCGGCACCUCGUGCAUGUGCAGUGCCCGCUG (((..(((((.(.....).)))))..))).((((((.(((((.(((((((((.((((((((....))))))).((((((.........)))))).).)))).)))))))))).)))))). ( -58.80) >DroEre_CAF1 6146 120 + 1 AUGGGCAUGCACAUGGUGGGCGUGGACAUGGGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGCAAAGUUGUGGCCGCCGGAUUGGCAGGCGGCACCUCGUGCAUGUGCAGCGCCCGCUG (((........)))((((((((((.(((((.(((((.(((((........(((.(..(((......)))..).)))(((.....)))..))))).)).))).))))).)).)))))))). ( -57.40) >DroYak_CAF1 18445 119 + 1 AUG-GCAUGCACAUGGUGGGCGUGGACAUGGGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGCAAAGUUGUGGCCGCCGGAUUGGCAGGCGGCACCUCAUGCAUGUGCAGUGCCCGCUG ...-.((((..((((.....))))..))))((((((.((((((.(((((.(((.(..(((......)))..).((((((.........))))))))))))))....)))))).)))))). ( -53.80) >consensus AUGGGCAUGCACAUGGUGGGCGUGGACAUGAGCGGGAGCUGCGACAUGAUGGUGAUGACUCAGCAGGGUCGUGGCCGCCGGAUUGGCAGGCGGCACCUCGUGCAUGUGCAGUGCCCGCUG .....((((..((((.....))))..))))((((((.(((((.(((((((((.((((((((....))))))).((((((.........)))))).).)))).)))))))))).)))))). (-57.32 = -56.04 + -1.28)

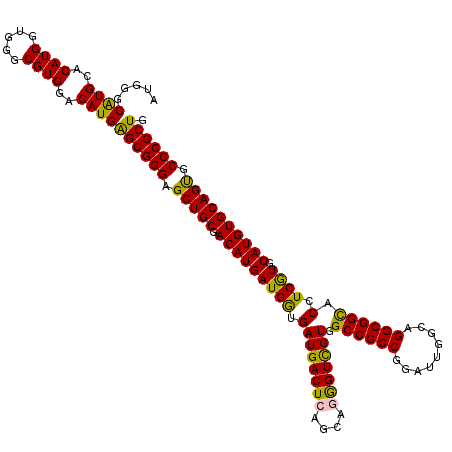
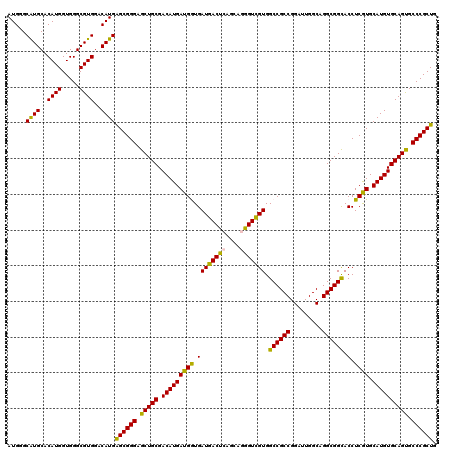
| Location | 3,786,116 – 3,786,236 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.50 |
| Mean single sequence MFE | -44.04 |
| Consensus MFE | -39.90 |
| Energy contribution | -39.86 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.91 |
| SVM decision value | 3.60 |
| SVM RNA-class probability | 0.999439 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3786116 120 - 27905053 CAGCGGGCGCUGCACAUGCACGAGGUGCCGCCUGCCAAUCCGGCGGCCACGACCCUCCUGAGUCAUCACCAUCAUGUCGCAGCUCCCGCUCAUGUCCACGCCCACCAUGUGCACGCCCAU .((((((.((((((((((.....(((((((((.........))))))...(((.(....).)))...)))..))))).))))).))))))...((.((((.......)))).))...... ( -45.90) >DroSec_CAF1 15475 120 - 1 CAGCGGGCACUGCACAUGCACGAGGUACCGCCCGCCAAUCCGGCGGCCACGACCCUGUUGAGUCAUCAUCAUCAUGUCGCAGCUCCCGCUCAUGUCCACGCCCACCAUGUGCAUGCCCAU ....(((((.((((((((...(.(((...(((((((.....)))))....(((..((.(((.......))).)).)))))(((....))).........))))..))))))))))))).. ( -41.90) >DroSim_CAF1 19229 120 - 1 CAGCGGGCACUGCACAUGCACGAGGUGCCGCCUGCCAAUCCGGCGGCCACGACCCUGUUGAGUCAUCAUCAUCAUGUCGCAGCUCCCGCUCAUGUCCACGCCCACCAUGUGCAUGCCCAU ....(((((.((((((((...(((.(((.(((.(((.....))))))...(((..((.(((.......))).)).)))))).)))..((..........))....))))))))))))).. ( -44.00) >DroEre_CAF1 6146 120 - 1 CAGCGGGCGCUGCACAUGCACGAGGUGCCGCCUGCCAAUCCGGCGGCCACAACUUUGCUGAGUCAUCACCAUCAUGUCGCAGCUCCCGCCCAUGUCCACGCCCACCAUGUGCAUGCCCAU ..(((((.((((((((((..((((((((((((.........))))))....)))))).(((....)))....))))).))))).))))).((((..((.(.....).))..))))..... ( -45.40) >DroYak_CAF1 18445 119 - 1 CAGCGGGCACUGCACAUGCAUGAGGUGCCGCCUGCCAAUCCGGCGGCCACAACUUUGCUGAGUCAUCACCAUCAUGUCGCAGCUCCCGCCCAUGUCCACGCCCACCAUGUGCAUGC-CAU .....((((.((((((((((((.(((((((((.........)))))).........((((.(.(((.......))).).))))....)))))))...........)))))))))))-).. ( -43.00) >consensus CAGCGGGCACUGCACAUGCACGAGGUGCCGCCUGCCAAUCCGGCGGCCACGACCCUGCUGAGUCAUCACCAUCAUGUCGCAGCUCCCGCUCAUGUCCACGCCCACCAUGUGCAUGCCCAU ....(((((.((((((((...(.(((((((((.........)))))).........((((.(.(((.......))).).))))................))))..))))))))))))).. (-39.90 = -39.86 + -0.04)

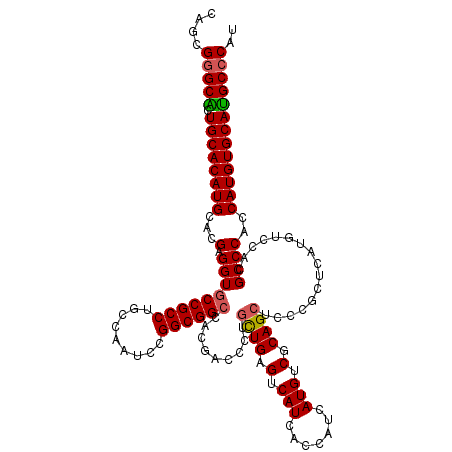
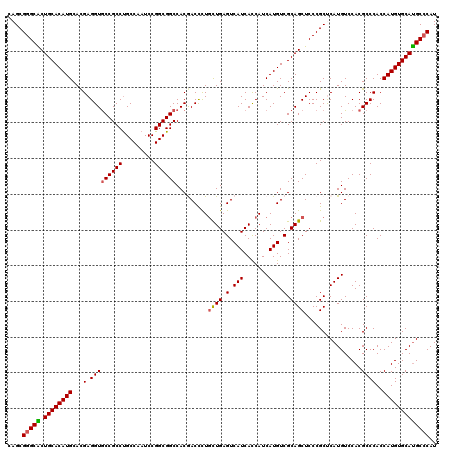
| Location | 3,786,196 – 3,786,316 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.00 |
| Mean single sequence MFE | -55.92 |
| Consensus MFE | -53.50 |
| Energy contribution | -52.62 |
| Covariance contribution | -0.88 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.68 |
| SVM RNA-class probability | 0.971971 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3786196 120 + 27905053 GAUUGGCAGGCGGCACCUCGUGCAUGUGCAGCGCCCGCUGCUCAUCCUGCGCCUGGGCUCGCCUCAAGGCCGUUUGCAGAGCCAUCACCCGCUGGCGGUCCGCCGUCAGUCGGCACUUCU (((...((((((((((...))))(((.(((((....))))).)))....))))))((((((((....))).(....).))))))))..((((((((((....))))))).)))....... ( -55.90) >DroSec_CAF1 15555 120 + 1 GAUUGGCGGGCGGUACCUCGUGCAUGUGCAGUGCCCGCUGCUCAUCCUGGGCCUGGGCUCGCCUCAAGGCCGUUUGCAGGGCCAUCACCCGCUGGCGGUCCGCCGUCAGUCGGCACUUCU (((((((((.(((.(((.(.((((((.(((((....))))).)).....((((((((....)))..)))))...)))).)((((.(....).)))))))))))))))))))......... ( -55.90) >DroSim_CAF1 19309 120 + 1 GAUUGGCAGGCGGCACCUCGUGCAUGUGCAGUGCCCGCUGCUCAUCCUGCGCCUGGGCUCGCCUCAAGGCCGUUUGCAGGGCCAUCACCCGCUGGCGGUCCGCCGUCAGUCGGCACUUCU ((((((((((((((.((..(((((((.(((((....))))).))...)))))..)))).)))))...((((((..((.(((......)))))..))))))....)))))))......... ( -56.40) >DroEre_CAF1 6226 120 + 1 GAUUGGCAGGCGGCACCUCGUGCAUGUGCAGCGCCCGCUGCUCGUCCUGCGCCUGGGCUCGCCUCAAGGCCGUUUGCAGAGCCAUCACCCGCUGGCGGUCCGCCGUCAGCCGGCACUUCU .....(((((((((((...))))....(((((....))))).)).)))))(((..((((((((....))).(....).))))).......((((((((....)))))))).)))...... ( -57.00) >DroYak_CAF1 18524 119 + 1 GAUUGGCAGGCGGCACCUCAUGCAUGUGCAGUGCCCGCUGCUCGU-CUGCGCCUGGGCUCGCCUCAAGGCCGUUUGCAGAGCCAUCACCCGCUGGCGGUCCGCCGUCAGCCGGCACUUUU .....((((((((((.....)))....(((((....))))).)))-))))(((..((((((((....))).(....).))))).......((((((((....)))))))).)))...... ( -54.40) >consensus GAUUGGCAGGCGGCACCUCGUGCAUGUGCAGUGCCCGCUGCUCAUCCUGCGCCUGGGCUCGCCUCAAGGCCGUUUGCAGAGCCAUCACCCGCUGGCGGUCCGCCGUCAGUCGGCACUUCU (((...((((((((((...))))(((.(((((....))))).)))....))))))((((((((....))).(....).)))))))).((.((((((((....)))))))).))....... (-53.50 = -52.62 + -0.88)

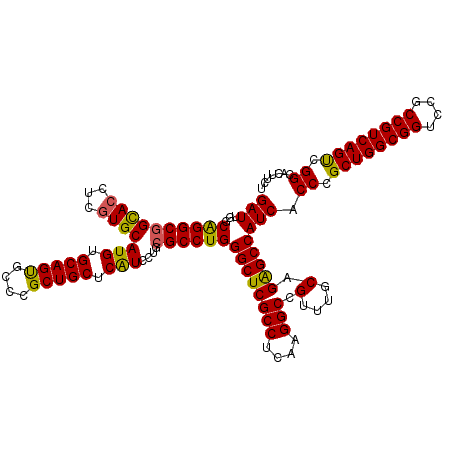

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:05:23 2006