
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 3,684,615 – 3,684,796 |
| Length | 181 |
| Max. P | 0.979098 |

| Location | 3,684,615 – 3,684,733 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 93.41 |
| Mean single sequence MFE | -40.95 |
| Consensus MFE | -34.27 |
| Energy contribution | -34.22 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736743 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
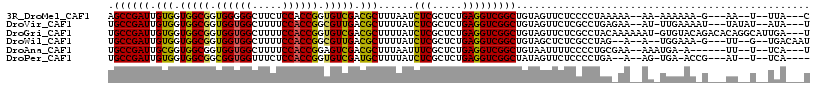
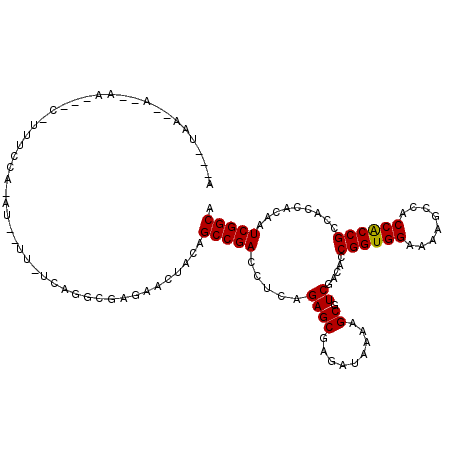
>3R_DroMel_CAF1 3684615 118 + 27905053 UAUGGAAGUUUUGCGUUUUUAUUGAAGCGUGCUUAAUGGCUUACCGUCCAUCUUAGCCUGUUCGGUUAAUUGUGGCGGAGCCGAUUGUGGUGGCGGUGGGGGCUUCUCCACCGGUGUC .....((((...(((((((....)))))))))))..(((((..(((.((((.((((((.....))))))..)))))))))))).....(..(.((((((((....)))))))).)..) ( -43.30) >DroVir_CAF1 57041 114 + 1 UAUGGAAGU--GGCGU--UUAUUGAAGCGUGCUUAAAGGCUUACCGUCCAUUUUAGCCUGUUCGGUUAGUUGUGGCGGUGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGCGUU .....((((--(.(((--((....))))))))))...(((..((((.((((.((((((.....))))))..))))))))))).........((((.((((((.....)))))).)))) ( -40.20) >DroGri_CAF1 14274 114 + 1 UAUGGAAGU--UGCGU--UUAUUGAAGCGUGCUUAUAGGCUUACCGUCCAUUUUAGCCUGUUCGGUUAGUUGCGGCGGUGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGUGUC .....((((--.((((--((....))))))))))...(((..((((((((..((((((.....)))))).)).))))))))).........((((.((((((.....)))))).)))) ( -39.10) >DroWil_CAF1 51557 114 + 1 AAUGGAAGU--UGCGU--UUAUUGAAGCGUGCUUAAAGGCUUACCGUCCAUUUUAGCCUGUUCGGUUAGUUGUGGCGGUGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGCGUU .....((((--.((((--((....))))))))))...(((..((((.((((.((((((.....))))))..))))))))))).........((((.((((((.....)))))).)))) ( -40.20) >DroMoj_CAF1 62018 114 + 1 UAUGGAAGU--UGCGU--UUAUUGAAGCGUGCUUUAAGGCUUACCGUCCAUUUUAGCCUGUUCGGUUAGUUGUGGCGGUGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGUGUU ....(((((--.((((--((....)))))))))))..(((..((((.((((.((((((.....))))))..))))))))))).........((((.((((((.....)))))).)))) ( -39.90) >DroAna_CAF1 29977 118 + 1 UAUGGAAGUUUUGCGUUUUUAUUGAAGCGUGCUUAAUGGCUUACCGUCCAUUUUAGCCUGUUCGGUUAGUUGUGGCGGUGCCGAUUGCGGUGGCGGUGGUGGCUUUUCCACCGGAGUC .....((((...(((((((....)))))))))))...(((..((((.((((.((((((.....))))))..)))))))))))((((.((((((.(((....)))...)))))).)))) ( -43.00) >consensus UAUGGAAGU__UGCGU__UUAUUGAAGCGUGCUUAAAGGCUUACCGUCCAUUUUAGCCUGUUCGGUUAGUUGUGGCGGUGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGUGUC .....((((...((((..........))))))))...(((..((((((((..((((((.....)))))).)).))))))))).........((((.((((((.....)))))).)))) (-34.27 = -34.22 + -0.06)

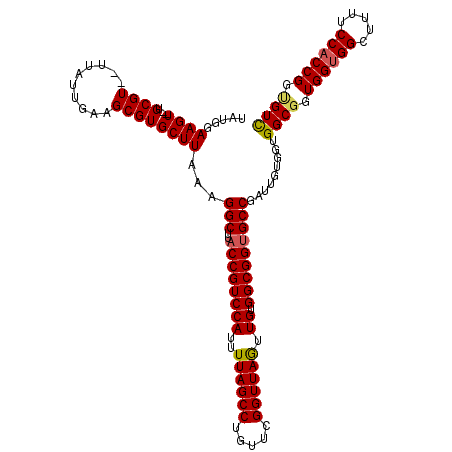

| Location | 3,684,615 – 3,684,733 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 93.41 |
| Mean single sequence MFE | -25.60 |
| Consensus MFE | -23.29 |
| Energy contribution | -23.32 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.979098 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
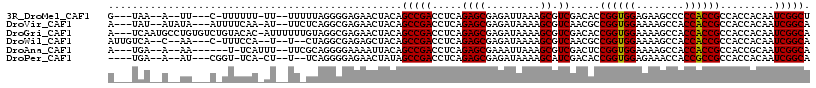
>3R_DroMel_CAF1 3684615 118 - 27905053 GACACCGGUGGAGAAGCCCCCACCGCCACCACAAUCGGCUCCGCCACAAUUAACCGAACAGGCUAAGAUGGACGGUAAGCCAUUAAGCACGCUUCAAUAAAAACGCAAAACUUCCAUA ......(((((........)))))((..........((((((((((...(((.((.....)).)))..))).)))..))))((((((....))).)))......))............ ( -27.10) >DroVir_CAF1 57041 114 - 1 AACGCCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCACCGCCACAACUAACCGAACAGGCUAAAAUGGACGGUAAGCCUUUAAGCACGCUUCAAUAA--ACGCC--ACUUCCAUA ...(.((((((........)))))).).........((((((((((....((.((.....)).))...))).))))..((......))............--..)))--......... ( -26.80) >DroGri_CAF1 14274 114 - 1 GACACCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCACCGCCGCAACUAACCGAACAGGCUAAAAUGGACGGUAAGCCUAUAAGCACGCUUCAAUAA--ACGCA--ACUUCCAUA ......(((((.....)))))(((((((.......((((...)))).......((.....))......))).))))..((......))..((........--..)).--......... ( -23.60) >DroWil_CAF1 51557 114 - 1 AACGCCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCACCGCCACAACUAACCGAACAGGCUAAAAUGGACGGUAAGCCUUUAAGCACGCUUCAAUAA--ACGCA--ACUUCCAUU ...((((((((.....)))))...(((.........))).....................)))...((((((.(((..((......))..((........--..)).--))))))))) ( -25.70) >DroMoj_CAF1 62018 114 - 1 AACACCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCACCGCCACAACUAACCGAACAGGCUAAAAUGGACGGUAAGCCUUAAAGCACGCUUCAAUAA--ACGCA--ACUUCCAUA ......(((((.....)))))...((..........((((((((((....((.((.....)).))...))).))))..)))...(((....)))......--..)).--......... ( -24.00) >DroAna_CAF1 29977 118 - 1 GACUCCGGUGGAAAAGCCACCACCGCCACCGCAAUCGGCACCGCCACAACUAACCGAACAGGCUAAAAUGGACGGUAAGCCAUUAAGCACGCUUCAAUAAAAACGCAAAACUUCCAUA .....((((((........)))))).....((....((((((((((....((.((.....)).))...))).))))..)))((((((....))).)))......))............ ( -26.40) >consensus AACACCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCACCGCCACAACUAACCGAACAGGCUAAAAUGGACGGUAAGCCUUUAAGCACGCUUCAAUAA__ACGCA__ACUUCCAUA ......(((((........)))))((..........((((((((((....((.((.....)).))...))).))))..)))...(((....)))..........))............ (-23.29 = -23.32 + 0.03)

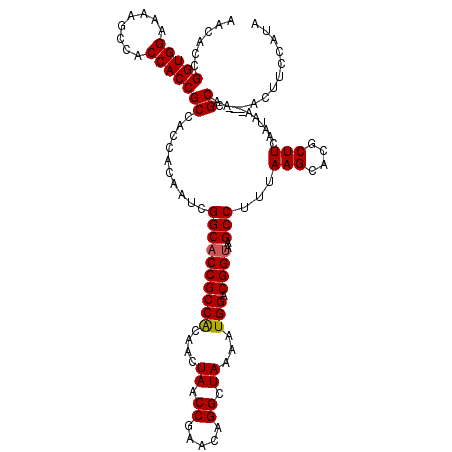

| Location | 3,684,693 – 3,684,796 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.43 |
| Mean single sequence MFE | -34.09 |
| Consensus MFE | -28.24 |
| Energy contribution | -27.63 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.04 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.679674 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3684693 103 + 27905053 AGCCGAUUGUGGUGGCGGUGGGGGCUUCUCCACCGGUGUCGACGCUUUAAUCUCGCUCUGAGGUCGGCUGUAGUUCUCCCCUAAAAA--AA-AAAAAA-G---AA--U--UUA---C (((((((..(((.((((((((((....)))))))((((....))))........))))))..)))))))..((((((..........--..-.....)-)---))--)--)..---. ( -31.73) >DroVir_CAF1 57115 106 + 1 UGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGCGUUGACGCUUUUAUCUCGCUCUGAGGUCGGCUGUAGUUCUCGCCUGAGAA--AU-UUGAAAAU---UAUAU--AUA---U .((((((..(((.(((((.(((((.....)))))((((....))))......))))))))..)))))).....(((((....)))))--..-........---.....--...---. ( -33.50) >DroGri_CAF1 14348 113 + 1 UGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGUGUCGACGCUUUUAUCUCGCUCUGAGGUCGGCUGUAGUUCUCGCCUACAAAAAAU-GUGUACAGACACAGGCAUUGA---U ((((..(((((((((((((((........))))))..((((((.(((............)))))))))........))))).))))....(-((((....)))))))))....---. ( -38.60) >DroWil_CAF1 51631 103 + 1 UGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGCGUUGACGCUUUUAUCUCGCUCUGAGGUCGGCUGUAGCUCUCGCCUAG--A--A--UGGAAA-G---UU--G--UGACAAU .((((((..(((.(((((.(((((.....)))))((((....))))......))))))))..))))))(((.((.((..((...--.--.--.))..)-)---..--)--).))).. ( -35.70) >DroAna_CAF1 30055 101 + 1 UGCCGAUUGCGGUGGCGGUGGUGGCUUUUCCACCGGAGUCGACGCUUUAAUUUCGCUCUGAGGUCGGCUGUAAUUUUCCCCUGCGAA--AAAUGA-A------UU--U--UCA---U .((((((..(((.(((((.(((((.....)))))(((((....)))))....))))))))..))))))................(((--((....-.------))--)--)).---. ( -31.40) >DroPer_CAF1 51707 100 + 1 UGCCGAUUGUGGUGGCGGCGGUGGUUUCUCCACCGGUGUCGAUGCUUUUAUCUCGCUCUGAGGUCGGCUAUAGUUCUCCCCUGA--A--AG-UGA-ACCG---AU--U--UCA---- .((.(((.((..(((((.((((((.....)))))).)))))..))....)))..))...((((((((......(((......))--)--..-...-.)))---))--)--)).---- ( -33.60) >consensus UGCCGAUUGUGGUGGCGGUGGUGGCUUUUCCACCGGUGUCGACGCUUUUAUCUCGCUCUGAGGUCGGCUGUAGUUCUCCCCUAA_AA__AA_UGAAAA_G___UA__U__UCA___U .((((((.(((.(((((.((((((.....)))))).))))).)))......(((.....)))))))))................................................. (-28.24 = -27.63 + -0.61)


| Location | 3,684,693 – 3,684,796 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 78.43 |
| Mean single sequence MFE | -27.13 |
| Consensus MFE | -20.45 |
| Energy contribution | -20.32 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.03 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.625958 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3684693 103 - 27905053 G---UAA--A--UU---C-UUUUUU-UU--UUUUUAGGGGAGAACUACAGCCGACCUCAGAGCGAGAUUAAAGCGUCGACACCGGUGGAGAAGCCCCCACCGCCACCACAAUCGGCU (---((.--.--((---(-((((((-..--.....))))))))).)))((((((.(((.....)))................((((((........)))))).........)))))) ( -25.90) >DroVir_CAF1 57115 106 - 1 A---UAU--AUAUA---AUUUUCAA-AU--UUCUCAGGCGAGAACUACAGCCGACCUCAGAGCGAGAUAAAAGCGUCAACGCCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCA .---...--.....---........-..--(((((....))))).....(((((.......(((........(((....))).(((((.....)))))..)))........))))). ( -27.46) >DroGri_CAF1 14348 113 - 1 A---UCAAUGCCUGUGUCUGUACAC-AUUUUUUGUAGGCGAGAACUACAGCCGACCUCAGAGCGAGAUAAAAGCGUCGACACCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCA .---....(((((((((....))))-)....((((.((((.........(.(((((((.....)))........)))).)...(((((.....)))))..))))...))))..)))) ( -29.50) >DroWil_CAF1 51631 103 - 1 AUUGUCA--C--AA---C-UUUCCA--U--U--CUAGGCGAGAGCUACAGCCGACCUCAGAGCGAGAUAAAAGCGUCAACGCCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCA .......--.--..---.-......--.--.--...(((....)))...(((((.......(((........(((....))).(((((.....)))))..)))........))))). ( -28.16) >DroAna_CAF1 30055 101 - 1 A---UGA--A--AA------U-UCAUUU--UUCGCAGGGGAAAAUUACAGCCGACCUCAGAGCGAAAUUAAAGCGUCGACUCCGGUGGAAAAGCCACCACCGCCACCGCAAUCGGCA .---...--.--..------.-..((((--(((.....)))))))....(((((.....((((((..........))).)))((((((........)))))).........))))). ( -24.80) >DroPer_CAF1 51707 100 - 1 ----UGA--A--AU---CGGU-UCA-CU--U--UCAGGGGAGAACUAUAGCCGACCUCAGAGCGAGAUAAAAGCAUCGACACCGGUGGAGAAACCACCGCCGCCACCACAAUCGGCA ----...--.--..---.(((-((.-((--(--....))).)))))...(((((.......(((.(((......))).....((((((.....)))))).)))........))))). ( -26.96) >consensus A___UAA__A__AA___C_UUUCCA_AU__UU_UCAGGCGAGAACUACAGCCGACCUCAGAGCGAGAUAAAAGCGUCGACACCGGUGGAAAAGCCACCACCGCCACCACAAUCGGCA .................................................(((((.....((((.........)).)).....((((((........)))))).........))))). (-20.45 = -20.32 + -0.14)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:03:58 2006