
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 3,596,622 – 3,596,758 |
| Length | 136 |
| Max. P | 0.864598 |

| Location | 3,596,622 – 3,596,718 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 82.45 |
| Mean single sequence MFE | -24.22 |
| Consensus MFE | -9.38 |
| Energy contribution | -9.93 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.08 |
| Mean z-score | -4.00 |
| Structure conservation index | 0.39 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.785059 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3596622 96 + 27905053 GGUAAUUUUAUCAGUUUUGCAGAGAAUAAUUGCAA---AAUUAAAUGAGCUAAUUAAACCAUUUCCAAGUGGAAUAAAAAGGCUCAUUUACUUUGCAAU ((((....))))....((((((((..(((((....---)))))((((((((..(((..(((((....)))))..)))...))))))))..)))))))). ( -23.50) >DroSec_CAF1 46909 96 + 1 GGUAAUUGCAUCAGUUUUGCACAGAAUAAUUGCAA---AAUUAAAUGAGCUAAUUAAACCAUUUCCAAAUGGAAUAAAAAGGCUCAUUUACUUUGCAAU ....((((((..(((((((((.........)))))---))))(((((((((..(((..(((((....)))))..)))...)))))))))....)))))) ( -25.60) >DroSim_CAF1 47747 96 + 1 GGUAAUUGCAUCAGUUUUGCACAGAAUAAUUGCAA---AAUUAAAUGAGCUAAUUAAACCAUUUCCAAAUGGAAUAAAAAGGCUCAUUUACUUUGCAAU ....((((((..(((((((((.........)))))---))))(((((((((..(((..(((((....)))))..)))...)))))))))....)))))) ( -25.60) >DroEre_CAF1 53372 96 + 1 AGUAAUUUCAUAAUUUUUGCACAGAAUAAUUGCAA---AAUUAAAUGUGCUACUUAAACCGUUUCCAAGCGGAAUAAAAAGCCUCAUUUAUUUUGCAAU .((((...........))))........(((((((---(((.(((((.(((..(((..(((((....)))))..)))..)))..))))))))))))))) ( -21.80) >DroYak_CAF1 70502 99 + 1 GGUAAUUUCAUAAUUAUUGCACGAAAUAAUUGCAAAAAAAUUAAAUGUGCUACUUAAUCCGUUUCCAAAAGGAUUAAAAAGCCUCAUUUACUUUGCAAU .(((((.........)))))........((((((((.....((((((.(((..(((((((..........)))))))..)))..)))))).)))))))) ( -21.00) >DroAna_CAF1 42726 97 + 1 AGUAAUUAGCUGGGAAAUGGCUAGCUUGAUUGCAAA--AAACAAAUGUGCUAAUUAAACCAUUUUCGAAUGGAAUAAAUAGCAUAAUUGCUUUUUCAAU .((((((((((((.......))))).)))))))(((--((.(((.(((((((.(((..(((((....)))))..))).))))))).))).))))).... ( -27.80) >consensus GGUAAUUUCAUCAGUUUUGCACAGAAUAAUUGCAA___AAUUAAAUGAGCUAAUUAAACCAUUUCCAAAUGGAAUAAAAAGCCUCAUUUACUUUGCAAU ............................(((((((......((((((.(((..(((..(((((....)))))..)))..)).).))))))..))))))) ( -9.38 = -9.93 + 0.56)

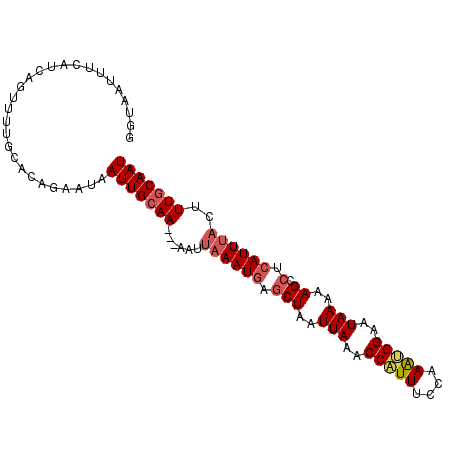

| Location | 3,596,622 – 3,596,718 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 82.45 |
| Mean single sequence MFE | -24.92 |
| Consensus MFE | -10.94 |
| Energy contribution | -12.05 |
| Covariance contribution | 1.11 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.94 |
| Structure conservation index | 0.44 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.864598 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3596622 96 - 27905053 AUUGCAAAGUAAAUGAGCCUUUUUAUUCCACUUGGAAAUGGUUUAAUUAGCUCAUUUAAUU---UUGCAAUUAUUCUCUGCAAAACUGAUAAAAUUACC ((((((((((((((((((.(..(((..(((........)))..)))..))))))))).)))---)))))))............................ ( -23.00) >DroSec_CAF1 46909 96 - 1 AUUGCAAAGUAAAUGAGCCUUUUUAUUCCAUUUGGAAAUGGUUUAAUUAGCUCAUUUAAUU---UUGCAAUUAUUCUGUGCAAAACUGAUGCAAUUACC ((((((((((((((((((.(..(((..(((((....)))))..)))..))))))))).)))---))))))).......((((.......))))...... ( -27.70) >DroSim_CAF1 47747 96 - 1 AUUGCAAAGUAAAUGAGCCUUUUUAUUCCAUUUGGAAAUGGUUUAAUUAGCUCAUUUAAUU---UUGCAAUUAUUCUGUGCAAAACUGAUGCAAUUACC ((((((((((((((((((.(..(((..(((((....)))))..)))..))))))))).)))---))))))).......((((.......))))...... ( -27.70) >DroEre_CAF1 53372 96 - 1 AUUGCAAAAUAAAUGAGGCUUUUUAUUCCGCUUGGAAACGGUUUAAGUAGCACAUUUAAUU---UUGCAAUUAUUCUGUGCAAAAAUUAUGAAAUUACU (((((((((((((((..(((.((((..(((........)))..)))).))).))))).)))---)))))))............................ ( -22.20) >DroYak_CAF1 70502 99 - 1 AUUGCAAAGUAAAUGAGGCUUUUUAAUCCUUUUGGAAACGGAUUAAGUAGCACAUUUAAUUUUUUUGCAAUUAUUUCGUGCAAUAAUUAUGAAAUUACC (((((((((((((((..(((.((((((((....(....))))))))).))).))))))....))))))))).((((((((.......)))))))).... ( -25.90) >DroAna_CAF1 42726 97 - 1 AUUGAAAAAGCAAUUAUGCUAUUUAUUCCAUUCGAAAAUGGUUUAAUUAGCACAUUUGUUU--UUUGCAAUCAAGCUAGCCAUUUCCCAGCUAAUUACU (((((((((((((...(((((.(((..(((((....)))))..))).)))))...))))))--))).))))..((((...........))))....... ( -23.00) >consensus AUUGCAAAGUAAAUGAGCCUUUUUAUUCCAUUUGGAAAUGGUUUAAUUAGCACAUUUAAUU___UUGCAAUUAUUCUGUGCAAAACUGAUGAAAUUACC (((((((..((((((.(.((..(((..(((((....)))))..)))..))).))))))......)))))))............................ (-10.94 = -12.05 + 1.11)

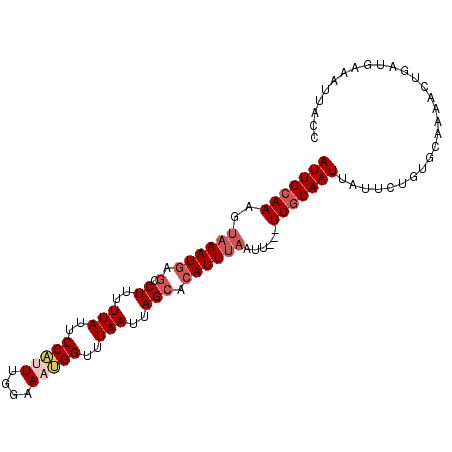

| Location | 3,596,646 – 3,596,758 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 84.52 |
| Mean single sequence MFE | -27.22 |
| Consensus MFE | -11.36 |
| Energy contribution | -12.03 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.15 |
| Mean z-score | -4.38 |
| Structure conservation index | 0.42 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.663385 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3596646 112 + 27905053 AAUAAUUGCAA---AAUUAAAUGAGCUAAUUAAACCAUUUCCAAGUGGAAUAAAAAGGCUCAUUUACUUUGCAAUUGGAUCAGUUUUAGCAACCCAGACUUAAAUCCGUUUUCAA ...((((((((---(..((((((((((..(((..(((((....)))))..)))...)))))))))).)))))))))((((.(((((.........)))))...))))........ ( -28.10) >DroSec_CAF1 46933 112 + 1 AAUAAUUGCAA---AAUUAAAUGAGCUAAUUAAACCAUUUCCAAAUGGAAUAAAAAGGCUCAUUUACUUUGCAAUUGGAUCAGUUUUAGCAACUCAGACUUAAAUCCGUUUUCAA ...((((((((---(..((((((((((..(((..(((((....)))))..)))...)))))))))).)))))))))((((.(((((.((...)).)))))...))))........ ( -29.10) >DroSim_CAF1 47771 112 + 1 AAUAAUUGCAA---AAUUAAAUGAGCUAAUUAAACCAUUUCCAAAUGGAAUAAAAAGGCUCAUUUACUUUGCAAUUGGAUCAGUUUUAGCAACUCAGACUUAAAUCCGUUUUCAA ...((((((((---(..((((((((((..(((..(((((....)))))..)))...)))))))))).)))))))))((((.(((((.((...)).)))))...))))........ ( -29.10) >DroEre_CAF1 53396 112 + 1 AAUAAUUGCAA---AAUUAAAUGUGCUACUUAAACCGUUUCCAAGCGGAAUAAAAAGCCUCAUUUAUUUUGCAAUUUGAUAAGUUCUAGCAACCCAGACUUAAAUCCGUUUUCAA ...((((((((---(((.(((((.(((..(((..(((((....)))))..)))..)))..))))))))))))))))...(((((.((........)))))))............. ( -24.40) >DroYak_CAF1 70526 115 + 1 AAUAAUUGCAAAAAAAUUAAAUGUGCUACUUAAUCCGUUUCCAAAAGGAUUAAAAAGCCUCAUUUACUUUGCAAUUAAAUCGGUCUUAGCCACCCAGACUUAAAUCUGUUUUCAG ..((((((((((.....((((((.(((..(((((((..........)))))))..)))..)))))).))))))))))....(((....)))...((((......))))....... ( -27.00) >DroAna_CAF1 42750 112 + 1 CUUGAUUGCAAA--AAACAAAUGUGCUAAUUAAACCAUUUUCGAAUGGAAUAAAUAGCAUAAUUGCUUUUUCAAUUUUUUGA-CUCUAGUUUCCUAGACUUAAAUCUGUUUUCAU ...(((((.(((--((.(((.(((((((.(((..(((((....)))))..))).))))))).))).)))))))))).(((((-.(((((....))))).)))))........... ( -25.60) >consensus AAUAAUUGCAA___AAUUAAAUGAGCUAAUUAAACCAUUUCCAAAUGGAAUAAAAAGCCUCAUUUACUUUGCAAUUGGAUCAGUUUUAGCAACCCAGACUUAAAUCCGUUUUCAA ..(((((((((......((((((.(((..(((..(((((....)))))..)))..)).).))))))..)))))))))...................................... (-11.36 = -12.03 + 0.67)

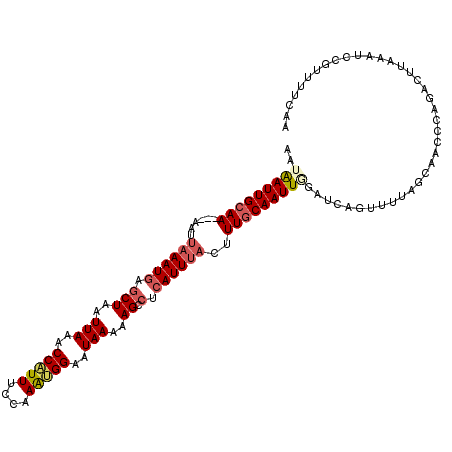

| Location | 3,596,646 – 3,596,758 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 84.52 |
| Mean single sequence MFE | -31.52 |
| Consensus MFE | -15.83 |
| Energy contribution | -17.05 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.07 |
| Mean z-score | -4.13 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.50 |
| SVM RNA-class probability | 0.762076 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3596646 112 - 27905053 UUGAAAACGGAUUUAAGUCUGGGUUGCUAAAACUGAUCCAAUUGCAAAGUAAAUGAGCCUUUUUAUUCCACUUGGAAAUGGUUUAAUUAGCUCAUUUAAUU---UUGCAAUUAUU .......(((((....)))))((((.....)))).....(((((((((((((((((((.(..(((..(((........)))..)))..))))))))).)))---))))))))... ( -30.50) >DroSec_CAF1 46933 112 - 1 UUGAAAACGGAUUUAAGUCUGAGUUGCUAAAACUGAUCCAAUUGCAAAGUAAAUGAGCCUUUUUAUUCCAUUUGGAAAUGGUUUAAUUAGCUCAUUUAAUU---UUGCAAUUAUU .......(((((....)))))((((.....)))).....(((((((((((((((((((.(..(((..(((((....)))))..)))..))))))))).)))---))))))))... ( -32.50) >DroSim_CAF1 47771 112 - 1 UUGAAAACGGAUUUAAGUCUGAGUUGCUAAAACUGAUCCAAUUGCAAAGUAAAUGAGCCUUUUUAUUCCAUUUGGAAAUGGUUUAAUUAGCUCAUUUAAUU---UUGCAAUUAUU .......(((((....)))))((((.....)))).....(((((((((((((((((((.(..(((..(((((....)))))..)))..))))))))).)))---))))))))... ( -32.50) >DroEre_CAF1 53396 112 - 1 UUGAAAACGGAUUUAAGUCUGGGUUGCUAGAACUUAUCAAAUUGCAAAAUAAAUGAGGCUUUUUAUUCCGCUUGGAAACGGUUUAAGUAGCACAUUUAAUU---UUGCAAUUAUU .............(((((((((....)))).)))))...((((((((((((((((..(((.((((..(((........)))..)))).))).))))).)))---))))))))... ( -30.40) >DroYak_CAF1 70526 115 - 1 CUGAAAACAGAUUUAAGUCUGGGUGGCUAAGACCGAUUUAAUUGCAAAGUAAAUGAGGCUUUUUAAUCCUUUUGGAAACGGAUUAAGUAGCACAUUUAAUUUUUUUGCAAUUAUU .......(((((....)))))(((.......)))....(((((((((((((((((..(((.((((((((....(....))))))))).))).))))))....))))))))))).. ( -32.40) >DroAna_CAF1 42750 112 - 1 AUGAAAACAGAUUUAAGUCUAGGAAACUAGAG-UCAAAAAAUUGAAAAAGCAAUUAUGCUAUUUAUUCCAUUCGAAAAUGGUUUAAUUAGCACAUUUGUUU--UUUGCAAUCAAG .(((.......(((.(.(((((....))))).-).)))...((((((((((((...(((((.(((..(((((....)))))..))).)))))...))))))--))).)))))).. ( -30.80) >consensus UUGAAAACGGAUUUAAGUCUGGGUUGCUAAAACUGAUCCAAUUGCAAAGUAAAUGAGCCUUUUUAUUCCAUUUGGAAAUGGUUUAAUUAGCACAUUUAAUU___UUGCAAUUAUU .......(((((....)))))..................((((((((..((((((.(.((..(((..(((((....)))))..)))..))).))))))......))))))))... (-15.83 = -17.05 + 1.22)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:03:23 2006