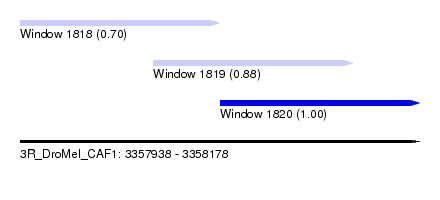
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 3,357,938 – 3,358,178 |
| Length | 240 |
| Max. P | 0.999231 |

| Location | 3,357,938 – 3,358,058 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.75 |
| Mean single sequence MFE | -44.36 |
| Consensus MFE | -41.32 |
| Energy contribution | -41.40 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.22 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.701631 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3357938 120 + 27905053 GCGGCGAUGUGGUGCCCAAGGACGUGAACGCCGCCAUCGCCACCAUCAAGACAAAGCGUACCAUUCAGUUCGUGGACUGGUGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCAC ..((((((((((((.....((.((....)))).....))))).))))........................((((..((((((((.(((........))))))))))).))))..))).. ( -43.90) >DroSec_CAF1 51369 120 + 1 GCGGCGAUGUGGUGCCCAAGGACGUGAACGCCGCCAUCGCCACCAUCAAGACGAAGCGCACCAUUCAGUUCGUGGACUGGUGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCAC ..((((((((((((.....((.((....)))).....))))).)))).....((.((((((((((((.....)))).)))))).(((((........))))))).))........))).. ( -46.90) >DroSim_CAF1 45675 120 + 1 GCGGCGAUGUGGUGCCCAAGGAUGUGAACGCCGCCAUCGCCACCAUUAAGACGAAGCGCACCAUUCAGUUCGUGGACUGGUGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCAC ((((((((((((((..((......))..))))).)))))))...........((.((((((((((((.....)))).)))))).(((((........))))))).))........))... ( -45.50) >DroEre_CAF1 44642 120 + 1 GCGGCGAUGUUGUGCCCAAGGACGUGAACGCCGCCAUCGCCACCAUCAAGACGAAGCGCACUAUUCAGUUCGUGGACUGGUGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCAC ..(((...((((((((((....(.((((.(((((..(((.(........).))).))((((....(((((....)))))))))......))))))).).)))))).)))).....))).. ( -41.70) >DroYak_CAF1 65907 120 + 1 GCGGCGAUGUGGUGCCCAAGGACGUGAACGCCGCCAUCGCCACCAUCAAGACUAAGCGCACCAUUCAGUUCGUGGACUGGUGCCCCACAGGCUUUAAGGUGGGCAUUAACUACCAGCCAC ..((((((((((((.....((.((....)))).....))))).))))........((.((((..((((((....)))))).(((.....))).....)))).))...........))).. ( -43.80) >consensus GCGGCGAUGUGGUGCCCAAGGACGUGAACGCCGCCAUCGCCACCAUCAAGACGAAGCGCACCAUUCAGUUCGUGGACUGGUGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCAC ..((((((((((((..((......))..))))).)))))))..............((((((((((((.....)))).))))))((((((........))))))............))... (-41.32 = -41.40 + 0.08)

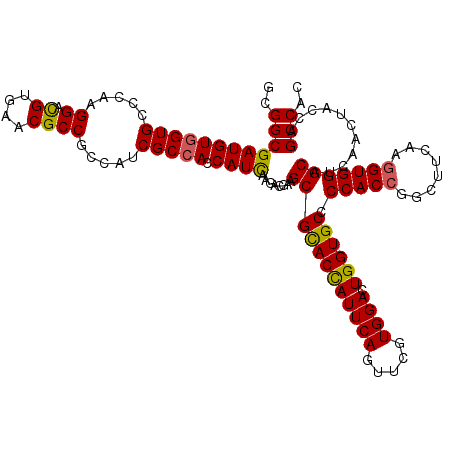
| Location | 3,358,018 – 3,358,138 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.83 |
| Mean single sequence MFE | -48.68 |
| Consensus MFE | -41.14 |
| Energy contribution | -41.38 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.92 |
| SVM RNA-class probability | 0.882364 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3358018 120 + 27905053 UGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCACCCACUGUCGUCCCAGGCGGGGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAAUACCACGGCUAUUGCCGAG (((((.(((........))))))))..........((((.((.(((((......)))))))...))))...((.(((((((((.....))))))))))).......((((....)))).. ( -47.30) >DroSec_CAF1 51449 120 + 1 UGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCACCCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAG (((((.(((........)))))))).......((.(((((.....)))))....))((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))).. ( -52.30) >DroSim_CAF1 45755 120 + 1 UGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCACCCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAG (((((.(((........)))))))).......((.(((((.....)))))....))((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))).. ( -52.30) >DroEre_CAF1 44722 120 + 1 UGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCACCCACUGUGGUUCCAGGCGGAGAUCUGGCCAAGGUUCAGCGUGCCGUGUGCAUGCUGUCCAAUACCACUGCUAUCGCCGAG (((((.(((........))))))))((.......((((((.....))))))...(((((((...(((....((..((((((((.....)))))))).))....)))...)).))))))). ( -45.60) >DroYak_CAF1 65987 120 + 1 UGCCCCACAGGCUUUAAGGUGGGCAUUAACUACCAGCCACCCACUGUGGUUCCUGGCGGGGAUCUGGCCAAGGUUCAGCGUGCUGUGUGCAUGCUGUCCAAUACCACUGCUAUUGCCGAG ..((((.((((......(((((.......)))))((((((.....))))))))))..))))...((((...((..((((((((.....)))))))).))((((.......)))))))).. ( -45.90) >consensus UGCCCCACCGGCUUCAAGGUGGGCAUCAACUACCAGCCACCCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAAUACCACGGCUAUCGCCGAG (((((.(((........))))))))((........(((((.....)))))....(((((.....(((((..((((((((((((.....)))))))).....))))..)))))))))))). (-41.14 = -41.38 + 0.24)

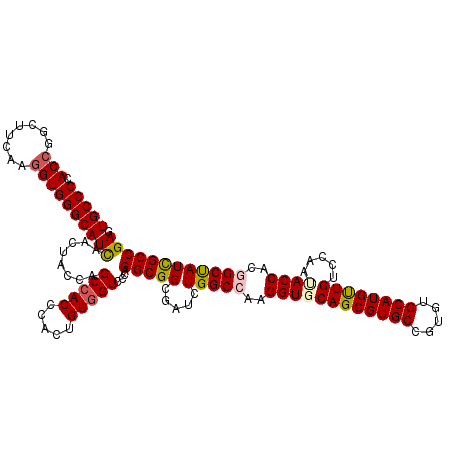
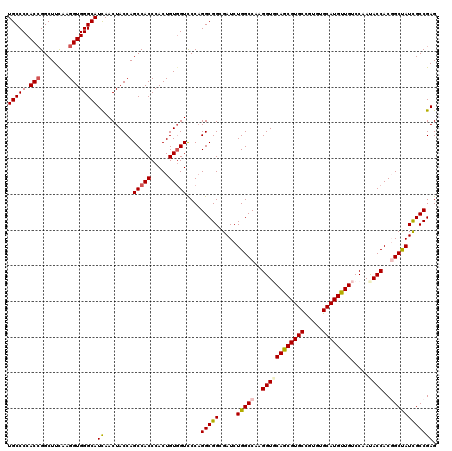
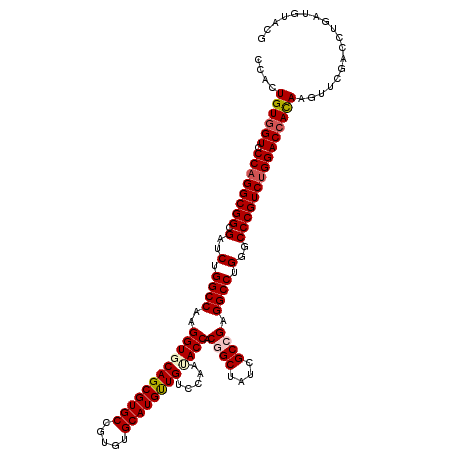
| Location | 3,358,058 – 3,358,178 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.67 |
| Mean single sequence MFE | -53.30 |
| Consensus MFE | -50.38 |
| Energy contribution | -51.02 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.45 |
| SVM RNA-class probability | 0.999231 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3358058 120 + 27905053 CCACUGUCGUCCCAGGCGGGGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAAUACCACGGCUAUUGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGAUCUGAUGUACG ....(((.((((.(((((((..((.((((..((.(((((((((.....))))))))))).......((((....)))).)))).)).))))))))))).))).................. ( -52.90) >DroSec_CAF1 51489 120 + 1 CCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAGGCCUGGGCCCGUCUGGACCAUAAGUUCGACCUGAUGUACG ....((((((((.(((((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))(.(((....)))))))))))))))).................. ( -55.90) >DroSim_CAF1 45795 120 + 1 CCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAACACCACGGCCAUCGCCGAGGCCUGGGCCCGUCUGGACCAUAAGUUCGACCUGAUGUACG ....((((((((.(((((((((..(((((..((((((((((((.....)))))))).....))))..))))))))))(.(((....)))))))))))))))).................. ( -55.90) >DroEre_CAF1 44762 120 + 1 CCACUGUGGUUCCAGGCGGAGAUCUGGCCAAGGUUCAGCGUGCCGUGUGCAUGCUGUCCAAUACCACUGCUAUCGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGACCUGAUGUACG ....((((((.((((((((.(..(.((((..((..((((((((.....)))))))).)).......(.((....)).).)))).)..))))))))))))))).................. ( -50.90) >DroYak_CAF1 66027 120 + 1 CCACUGUGGUUCCUGGCGGGGAUCUGGCCAAGGUUCAGCGUGCUGUGUGCAUGCUGUCCAAUACCACUGCUAUUGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUUGACCUGAUGUACG ....((((((((..((((((..((.((((..((..((((((((.....)))))))).)).......(.((....)).).)))).)).)))))).)))))))).................. ( -50.90) >consensus CCACUGUGGUCCCAGGCGGCGAUCUGGCCAAGGUGCAGCGUGCCGUGUGCAUGUUGUCCAAUACCACGGCUAUCGCCGAGGCCUGGGCCCGUCUGGACCACAAGUUCGACCUGAUGUACG ....((((((.((((((((.(..(.((((..((((((((((((.....)))))))).....)))).((((....)))).)))).)..))))))))))))))).................. (-50.38 = -51.02 + 0.64)

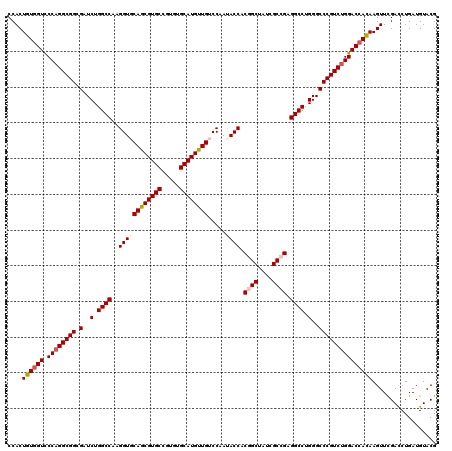
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:01:12 2006